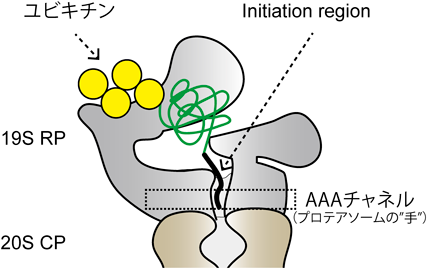
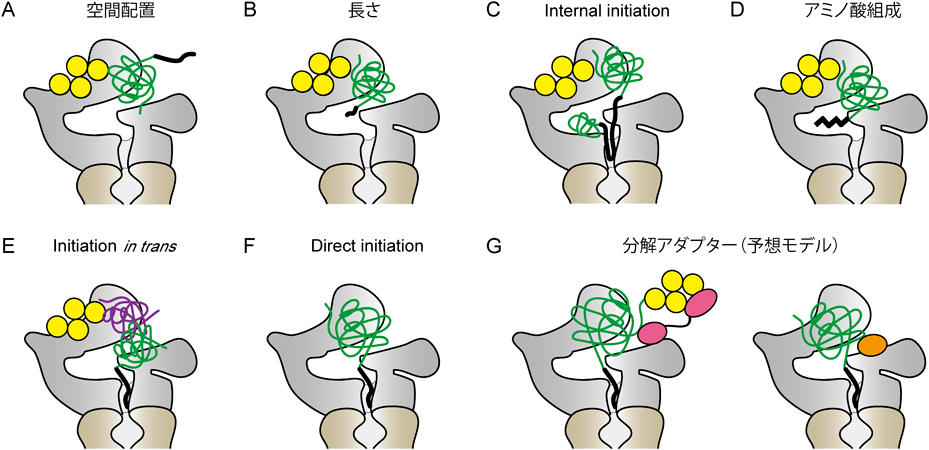
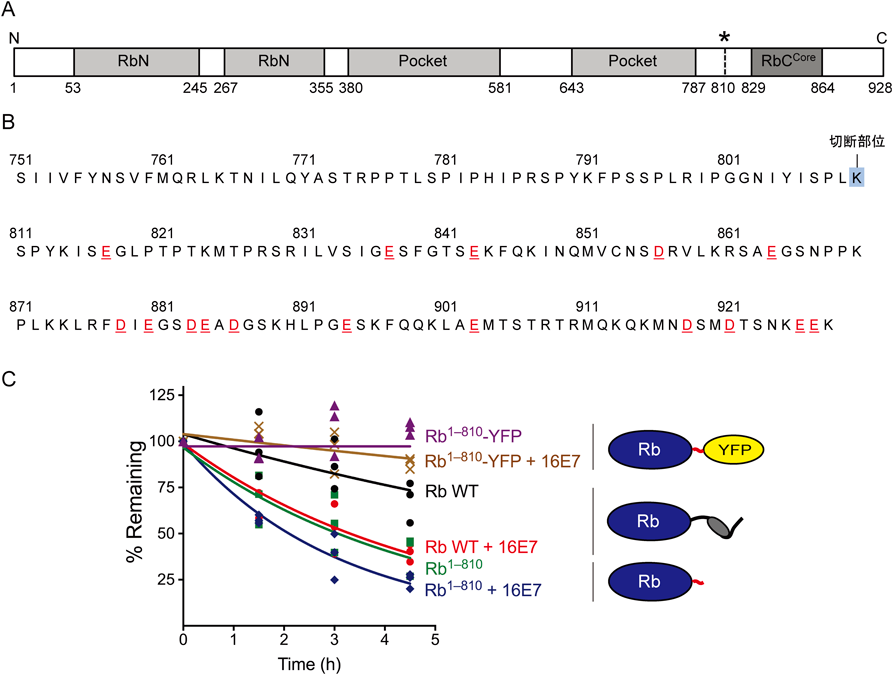
initiation regionを介したプロテアソームによるタンパク質分解制御Regulation of proteasomal degradation via initiation region
公益財団法人東京都医学総合研究所基礎医科学研究分野蛋白質代謝プロジェクトProtein Metabolism Project, Tokyo Metropolitan Institute of Medical Science ◇ 〒156–8506 東京都世田谷区上北沢2–1–6 ◇ 2–1–6 Kamikitazawa, Setagaya-ku, Tokyo 156–8506, Japan