翻訳品質管理機構RQCは,異常な翻訳を監視し異常タンパク質を合成途中で分解除去する翻訳品質管理機構であり,タンパク質恒常性維持のごく初期段階としてきわめて重要な役割を果たす.急速に理解が進んでいるRQCの分子機構の概略を以下に記述する1–3).
1)RQCの概要
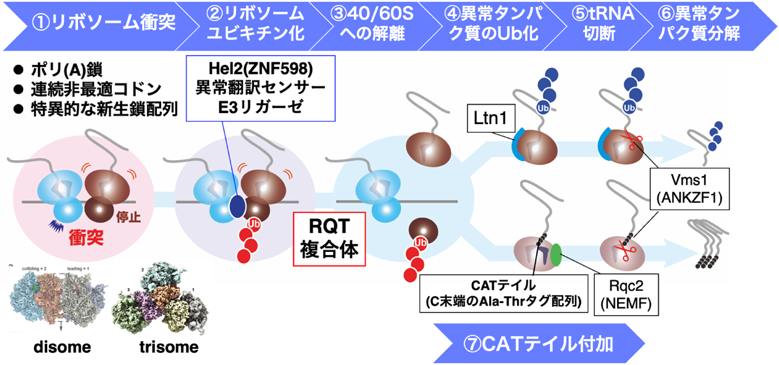
RQCにおいて,異常翻訳を認識し異常タンパク質を分解するまでの,各段階を担う因子が明らかになった.翻訳伸長中のリボソームがmRNA上で強く停滞すると,後続のリボソームが追いつき,先頭で停止したリボソームと衝突する.E3ユビキチン化酵素であるHel2(ヒトではZNF598)は,このリボソームどうしの衝突を異常な翻訳とみなし,衝突したリボソームをユビキチン化する.ユビキチン化されたリボソームはサブユニットへと解離し,途中まで合成された新生ペプチド鎖は分解経路に進む(図1).
2)異常翻訳の分子実体としての衝突リボソームとRQT(RQC trigger)複合体による解消
過去数年の研究の進展により,E3ユビキチンリガーゼHel2(ヒトZNF598)による特異的な基質認識が解明され,異常翻訳の実体が明らかとなった(図1①).クライオ電子顕微鏡による単粒子解析で解明された二つの衝突したリボソーム構造であるdisomeの構造を複数の例で比較することにより,衝突リボソームセンサーであるHel2が,40S–40Sリボソーム間で形成される特殊な構造を認識することが示された.まず出芽酵母において,RQCの基質となる連続したレアコドンを用いて形成されたdisomeが,Hel2によるユビキチン化の特異的基質であることが示された4–6).また哺乳類細胞においても,翻訳阻害剤存在下での試験管内翻訳反応系で形成したdisomeが,Hel2のヒトオルソログZNF598によりユビキチン化されることが示された7, 8).
出芽酵母のSDD1は野生型配列が翻訳停滞を示すユニークな遺伝子であり,翻訳停滞によってタンパク質合成を分解経路へと導くSDD1遺伝子の翻訳停滞配列が同定された.この翻訳停滞の配列を利用することで,RQCの各段階が生化学的に解析可能となった6).SDD1はミトコンドリアのATP/ADP交換機能欠損を過剰発現で相補する因子として同定されたが9),その生理機能とRQCによる制御の役割は不明であり,今後明らかにする必要がある.リボソームプロファイリングにより,SDD1内の翻訳停滞配列が同定された.このSDD1内の翻訳停滞配列を用いて,1)翻訳停滞,2)リボソームの衝突によるtrisomeの形成,3)衝突リボソームに特異的なユビキチン化反応,を試験管内翻訳反応で再構築した.さらに,クライオ電子顕微鏡による単粒子解析でtrisomeの構造を明らかにし,disome構造と比較した.その結果,40S–40S間で形成される特殊な構造が,Hel2が認識する特異的構造であり,異常翻訳の実体であることが強く示唆された(図1①).RQT(ribosome-associated quality control trigger)複合体はRQCに必須な因子として筆者らが同定した因子であり,4)ユビキチン化に依存した翻訳伸長途中でのサブユニット解離を担う5, 6).SDD1内の翻訳停滞配列を用いて試験管内翻訳反応で形成させたユビキチン化リボソームが,RQT複合体によってサブユニットに解離する反応を試験管内で再現した(図1②,③).以上により,RQT複合体がユビキチン化衝突リボソームのサブユニット解離を誘導することが証明された.哺乳類細胞においてもRQT複合体が解離反応を担う因子であることが示されているが10, 11),細胞内のRQC基質である翻訳停滞配列を用いた解離反応の完全再構成が今後の重要な課題である.
3)RQCとストレス応答
リボソーム衝突により誘起されるシグナル伝達と細胞死との関連も明らかになってきている.翻訳伸長阻害剤とアミノ酸飢餓や紫外線(UV)照射を含む一般的な細胞ストレス条件時に,リボソーム衝突はストレス活性化プロテインキナーゼ(SAPK)とGCN2を介したストレス応答経路を活性化する12).MAPKKKであるZAKがリボソーム衝突のセンサーとして機能し,SAPK(p38/JNK)とGCN2シグナル伝達経路の両方を活性化する.ZAKは,衝突するリボソームの最小単位であるdisome上で,特異的に自己リン酸化を行う.また,GCN2による統合ストレス応答(integrated stress response:IRS)の活性化と,リボソーム衝突の関連が示唆されている13).リボソーム衝突によってRQCとIRSの両方が活性化され,かつ一方の経路の不活性化は他方の過剰活性化をもたらすモデルが提唱された.しかしながら,GCN2とHel2の認識する基質は異なっており,GCN2はアミノアシルtRNAの細胞内量の低下の結果,空になったリボソームのA部位を認識する.一方で,Hel2(ヒトZNF598)は衝突リボソームの40Sどうしの相互作用部位を識別する.したがって,GCN2による統合ストレス応答と異常翻訳品質管理機構RQCの相互の関係を明らかにすることが,今後の重要な課題である.
4)CATテイルの分子機構と生理的意義
合成途上の異常タンパク質は,60Sサブユニットに特異的に結合するLtn1によって分解の目印となるユビキチン化を受ける14–17).ユビキチンプロテアソーム依存分解系の主要因子Cdc48とUfd3が,ユビキチン化異常タンパク質をプロテアソーム依存の分解に導く(図1④~⑥)17, 18).出芽酵母において,Ltn1によるユビキチン化が起きない場合に,60Sサブユニットの異常新生タンパク質のカルボキシ末端(C末端)に,アラニンとトレオニンからなるCATテイル(C-terminal alanine-threonine tail)と呼ばれるタグ配列がmRNA非依存的に付加される(図1⑦)19).
CATテイルは,異常タンパク質の分解を促進する1, 20).ユビキチン化部位のリシン残基をリボソーム外に露出させることで,Ltn1によるユビキチン化の基質とする機能が示唆されている.また異常新生タンパク質にCATテイルを付加する反応は,原核生物にも保存されており,枯草菌でのRqcHによるAla連続配列の付加は分解のシグナルになる21, 22).Hsp15に相同性を持ち,RqcHと共役して機能する新規因子としてRqcPも同定された.クライオ電子顕微鏡による複合体の構造解析により,RqcPはリボソームP部位のペプチジルtRNAの構造を安定化することが明らかにされ23),60Sサブユニットでのペプチド形成機構が明らかになりつつある.終止コドンを欠失したノンストップmRNAの品質管理システムにおいて,異常タンパク質のC末端に付加される連続したAla配列は,プロテアーゼに認識され,異常タンパク質の分解を誘導する24, 25).したがって,異常タンパク質のC末端への分解タグの付加と迅速な分解は,普遍的な品質管理機構として広く保存されていると考えられる.さらに最近,E3ユビキチン化酵素複合体CRL2KLHDC10と,C末端ルール依存の分解に関与する新規のE3ユビキチン化酵素であるPIRH2/RCHY1が,C末端のAlaテイルを持つRQC産物に直接結合して分解することが報告され,新規のRQC経路として注目されている26).
一方で,CATテイルは凝集体の蓄積を誘導することも報告されている27).LTN1欠損マウスでは進行性の神経変性と運動機能障害が観察された28).しかしながら,神経変性が発症する原因がCATテイルによる凝集体の蓄積であるかは不明であった.その後,ショウジョウバエでもミトコンドリア機能欠損により翻訳終結活性が低下した場合に,ミトコンドリアタンパク質にCATテイルが付加されることが報告され,哺乳類細胞でも異常タンパク質にCATテイルの付加が起こることが示唆された29).最近,RQCの機能不全による細胞障害の原因に,CATテイル化タンパク質の蓄積が関わることが明らかになった.LTN1依存の異常タンパク質分解が機能不全に陥ると,CATテイル化されたタンパク質が凝集体を形成し,神経細胞の形態形成の阻害やプログラム細胞死を引き起こす30).タンパク質恒常性の破綻は,神経変性疾患をはじめとするさまざまなヒト疾患の発症のリスク要因となるため,CATテイル化されたタンパク質の運命決定機構の理解は,これらの疾患の分子機序の理解につながると期待される.
CATテイルの形成には酵母Rqc2が必須であり,試験管内で再構成されているが20),その形成効率を制御する機構はいまだ不明である.RQCを引き起こす配列として,対応するtRNAの量が少ないレアコドンや,連続した塩基性アミノ酸配列が同定されている.トリプトファンの連続配列もRQCを引き起こすが,Rqc2依存のCATテイルの形成を阻害することが最近判明した31).60Sサブユニット中で,8個以上の連続トリプトファンがリボソームトンネルの特定の位置に配置した場合に,CATテイルの形成を阻害した.以上の結果から,Rqc2依存にペプチド結合が形成される分子機構の一端が明らかになった.CATテイルの形成効率の制御機構の理解には,60Sサブユニット中での新生ポリペプチド鎖とリボソームトンネルとの相互作用の詳細や,Rqc2によるアミノアシルtRNAの識別機構を明らかにする必要がある.
5)異常タンパク質のtRNAからの解離機構
RQCの最終段階では,Ltn1でユビキチン化された異常タンパク質がリボソームから解離し,プロテアソームによって分解される(図1④~⑥).60Sサブユニット中の異常タンパク質はペプチジルtRNAであり,最終的にユビキチン化異常タンパク質がプロテアソームで分解されるためには,異常タンパク質がtRNAから解離する必要がある.最近,RQCのtRNAから異常タンパク質が解離する分子機構が明らかになってきた32).通常の翻訳終結時にはeRF1-ABCE1が機能するが33–36),60S上ではVms1(ヒトではANKZF1)がtRNAの解離を担う37, 38).生化学的解析により,Vms1/ANKZF1がtRNA部分を切断することで,異常タンパク質をリボソームから解離することが示された39, 40).次に,クライオ電子顕微鏡によるVms1/ANKZF1と60Sサブユニットの複合体の構造が解明され,Vms1/ANKZF1がtRNAを切断し,合成途上のタンパク質をリボソームから解離させ,分解に導く構造基盤が明らかになった41).さらに,ANKZF1で切断されたtRNAの最適なリサイクル経路が明らかになった40).ELAC1がANKZF1で切断されたtRNA上の2′,3′-環状リン酸を除去し,その後CCAが再付加され,修復されたtRNAは再利用される.
Vms1/ANKZF1によるタンパク質のtRNAからの解離は,CATテイル付加の制御にも重要である.Vms1の欠損細胞では,合成途上の異常タンパク質のリボソームからの解離が遅れ,Rqc2によるCATテイルの付加が異常に亢進する26).Vms1/ANKZF1のtRNA切断活性に異常が生じると,CATテイルの付加が異常に亢進し,CATテイルが伸長したタンパク質は凝集体を形成し,細胞機能の破綻を引き起こす.特に,エネルギー産生をはじめ老化や疾患とも密接に関わるミトコンドリアにおいてタンパク質の顕著な凝集が観察され,ミトコンドリアの機能が破綻することが明らかになった.
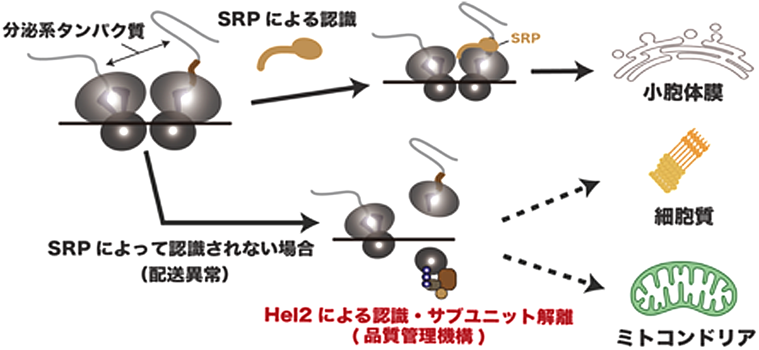
6)RQCの生理的意義と疾患
RQCの分子機構の解明が進んだ結果,疾患との関連も明らかになってきている.LTN1欠損下でのCATテイル形成は,神経突起伸長を阻害し,神経細胞死を誘導する29).ZNF598変異が自閉症の患者で確認され42),Rqc2のヒトホモログであるNEMF遺伝子の変異は神経筋疾患との関連が示唆されている43).RQCの細胞内標的を同定する目的で,異常翻訳センサータンパク質であるHel2が結合したリボソームに含まれるmRNAの網羅的解析(Hel2の選択的リボソームプロファイリング)が行われた.その結果,分泌系タンパク質の配送異常を伴う異常翻訳がHel2によって認識され,除去されることが明らかとなった44).膜タンパク質などの分泌系タンパク質の大部分は,翻訳伸長段階でSRP(signal recognition particle)に認識されることで共翻訳的に小胞体膜へと配送される.膜タンパク質には疎水性に富んだ領域が多く存在し,親水性の細胞質ではペプチド鎖の折りたたみに支障を来す.そのため,配送過程に異常が生じると,細胞質でタンパク質の凝集体が形成される危険がある.また,一部の膜タンパク質では,SRPの機能低下によるミトコンドリアへの誤配送が報告されており,それによりミトコンドリアの機能不全が惹起される.この解析により,分泌系タンパク質の翻訳伸長過程のごく初期に,Hel2がリボソームと結合するようすが観察され,SRPの機能低下によって,Hel2の結合量がさらに増加することが明らかになった.また,Hel2の欠損下では,SRPの機能低下によって惹起されるミトコンドリアの機能不全や分泌系タンパク質のミトコンドリアへの誤配送も亢進することが明らかになった.以上の結果から,Hel2が分泌系タンパク質の配送異常を早期に識別し,強制的に翻訳を終結させることで,毒性を持った不良タンパク質の蓄積を未然に防ぐことが示唆された(図2).翻訳に共役した品質管理機構の新たな機能が明らかになったため,Hel2/ZNF598が神経細胞等で同様の機能を保持するか確認する必要がある.
7)酸化ストレス時の異常翻訳と品質管理
酸化ストレス時の翻訳終結低下による異常翻訳と,品質管理の関係も示唆されている.ABCE1は,翻訳終結後にリボソームをリサイクルする反応に必須な因子であるが,その活性はFe/Sクラスター([4Fe–4S]2+)に依存している45).Fe/SクラスターはDNAポリメラーゼやDNAヘリカーゼの活性に必須であり,核DNAの合成と修復,リボソームタンパク質合成,およびヌクレオチド代謝や細胞の鉄調節等の生理活性に重要である46, 47).Fe/Sクラスターのアセンブリとアポタンパク質への挿入はミトコンドリアに局在する因子に依存しており,Fe/Sクラスター生合成の欠損は,重度の神経疾患や血液疾患,または代謝性疾患を引き起こす.多くのFe/Sクラスターを含むタンパク質と同様に,ABCE1も酸化ストレス状態で活性が低下することが報告されている48).特定の脳領域では,加齢に伴いABCE1の発現が低下し3′ UTRを翻訳するリボソームが増加し,かつ3′ UTR由来のRNA断片が多く検出される49).本来翻訳されない3′ UTRが翻訳された場合に,翻訳伸長が阻害されることが報告されており,衝突リボソームは,RQCと共役してmRNAの切断であるNGD(No-go decay)も引き起こす結果,終止コドン近くのmRNAがエンドヌクレアーゼで切断されると考えられる.また鉄欠乏状態においてABCE1の機能が低下し,3′ UTRを翻訳するリボソームが増加する結果,ナンセンス依存mRNA分解(nonsense-mediated mRNA decay:NMD)が抑制される50).NMDに必須なUPF1が3′ UTRに結合することが,NMDにおける異常な翻訳終結の認識に必須であることから,3′ UTRを翻訳するリボソームによってUPF1が解離した結果,NMDが抑制されると考えることができる.ミトコンドリア機能欠損時には,ABCE1の発現が低下し,ミトコンドリア外膜上の複合体Iのサブユニットの翻訳終結が抑制される結果,E3ユビキチン化酵素Not4によってABCE1がユビキチン化され,オートファジー受容体をミトコンドリアにリクルートし,最終的にマイトファジーを誘起するポリユビキチンシグナルが形成されることが示唆されている51).ABCE1の発現低下により翻訳終結が抑制される結果,どのような翻訳異常が起こり品質管理RQCが誘導されるか,また生理機能の低下につながるか,個々のストレスや老化との関連を解析することで明らかにする必要がある.その知見は,酸化ストレスが関与するミトコンドリア機能阻害や老化,また多くの疾患発症のメカニズムの理解にも貢献するものと考えられる.
4. 翻訳速度を感知しmRNAの安定性を決定する仕組み
リボソームユビキチン化は,翻訳品質管理RQCとリボソーム品質管理NRDのみでなく,翻訳速度に依存したmRNA安定性制御にも必須であることが最近見いだされた.
ストレスや環境対応における遺伝子発現制御において,細胞内のmRNA量の調節は重要な役割を果たす.mRNAの発現量は合成と分解の割合で決まっており,翻訳効率とmRNA安定性が比例することが知られている.ほとんどのアミノ酸は複数種類のコドンによってコードされており,同じアミノ酸に対応する同義コドンに対応するtRNAの細胞内存在量には偏りがある.同じアミノ酸をコードする場合でも異なるコドン間では翻訳の伸長速度が異なり,多く使われるコドン(最適コドン)に対応するtRNAは存在量が多く,翻訳の伸長速度が早い.その結果,最適コドンを多く含むmRNAは,翻訳効率も高いことが知られている.翻訳の伸長速度は発現量の調節だけでなく,合成されるペプチド鎖のフォールディングやターゲティングなどとも共役しており,コドンの選択は遺伝子発現において非常に重要な役割を果たしている.
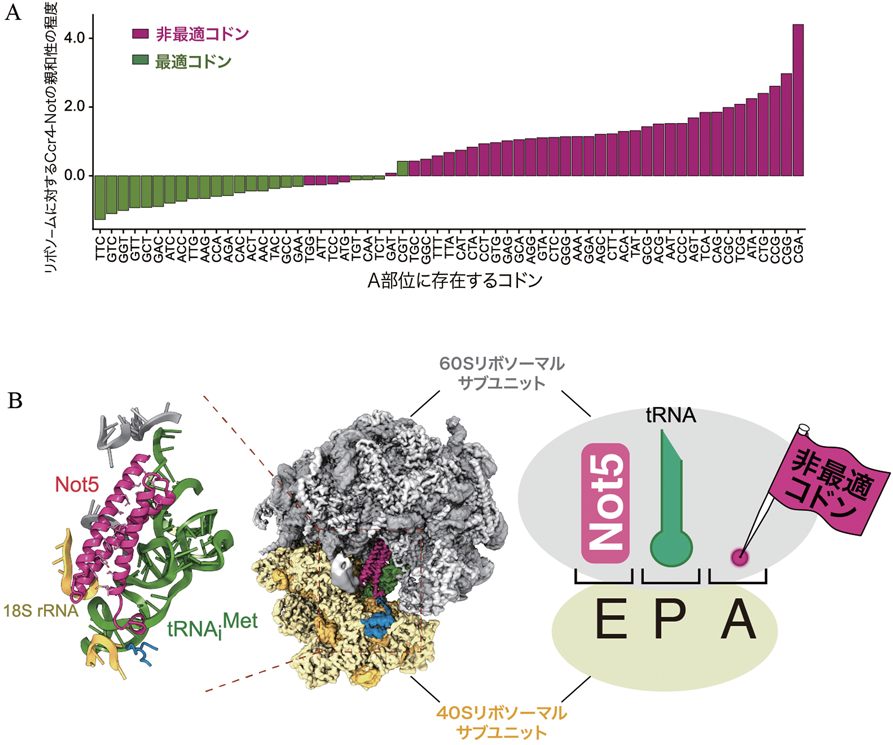
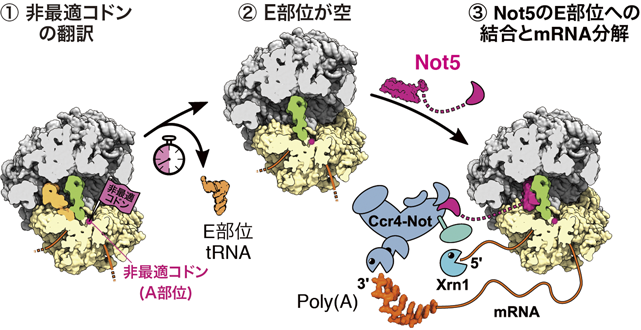
近年,個々のmRNAが持つ固有の安定性が,コドンの最適化で調節されることが示された.コドンの最適度が高いmRNAは安定であり,逆に低いものは不安定であることが網羅的な解析で確認された.これは,翻訳の伸長速度でmRNAの半減期が決定されることを強く示唆している.一方で,コドンの最適度(翻訳の伸長速度)を監視し,個々のmRNAが持つ固有の安定性を決定する機構は依然として不明であった.キャップ構造を除去するDhh1を含め,翻訳速度依存にリボソームと相互作用しmRNA安定性を決定する候補因子の解析が行われたが,リボソームに直接結合する因子は未同定のままであった.この問題を解決するため我々は,mRNAの転写・分解や翻訳抑制に関与するCcr4–Not複合体に注目した56).Ccr4–Not複合体は細胞内の主要なポリ(A)短鎖化複合体であり,特異的なRNA結合タンパク質を介して,個別のmRNAの安定性を制御することが広く知られている.我々は,Ccr4–Not複合体がリボソームに直接結合することを見いだした.選択的リボソームプロファイリングにより,Ccr4–Not複合体が特異的に結合するリボソームが翻訳するmRNAの特徴を網羅的に解析した結果,Ccr4–Not複合体は,コドンの最適性が低いmRNAを翻訳するリボソームに対して強い親和性を持つことが明確になった(図3A).コドンレベルの解析では,Ccr4–Not複合体とリボソームの親和性がコドンの最適度と非常に強い逆相関を示した(図3A).また,Ccr4–Not複合体の機能欠損によって,コドン最適度依存的なmRNAの安定性制御が失われた.クライオ電子顕微鏡を用いた単粒子解析により,リボソームに結合したCcr4–Not複合体の構造を決定したところ,Ccr4–Not複合体の構成タンパク質の一つであるNot5が,A部位にtRNAを含まないリボソームのE部位に結合することが明らかになった(図3B).翻訳過程において,コドン–アンチコドンの認識はリボソームのA部位で行われる.非最適コドンを翻訳しているリボソームでは,対応するtRNAの存在量が少ないため,A部位にtRNAが結合するまでに長い時間が必要となる.A部位にtRNAが結合しない状態が継続すると,E部位からtRNAが解離する.tRNAが解離し空になったE部位にCcr4–Not複合体が結合し,効率よくmRNAを分解に導くことが示された.以上の結果より,Ccr4–Not複合体は,非最適コドンを翻訳中のリボソームのE部位に効率よく結合することで,コドンの最適度を監視し,mRNAの安定性を制御することが明らかとなった(図4).コドンの最適化によるmRNAの分解制御機構の実体が明らかになり,長年不明であった遺伝暗号における同義コドンの存在意義がより明確になった56).遺伝子の発現制御は生命の根幹であり,翻訳速度に応じたmRNAの安定性制御の理解はさまざまな生命現象と幅広い疾患の病態の理解につながると期待される.このCcr4–Notとリボソームの結合には,リボソームタンパク質RPS7(eS7)のユビキチン化が必須であり56),ユビキチン化eS7にリクルートされる因子が足場となってCcr4–Notがリボソームに安定して結合可能になると予想される(表1).
小胞体内での異常タンパク質の蓄積は細胞に有害であるため,UPR経路を誘導することで応答する.出芽酵母では,小胞体ストレスにより活性化された膜タンパク質Ire1が,転写因子Hac1の前駆体mRNAをスプライシングし,Hac1が合成されてシャペロン等の転写が誘導される.小胞体ストレス時にリボソームタンパク質のユビキチン化が亢進するため,小胞体ストレス応答に必須なユビキチン化部位を同定した.その結果,E3リガーゼNot4によるeS7のユビキチン化が,小胞体ストレス時の翻訳制御に必須であることが明らかとなった57, 58).eS7のユビキチン化は,翻訳速度に応じたmRNAの安定性制御にも必要であり,両者の関連を明らかにすることで,リボソーム修飾による発現制御の理解が深まると期待される.