CRISPR(clustered regularly interspaced short palindromic repeat)-Cas(CRISPR-associated)システムは多くの細菌が持つ獲得免疫システムで,細菌に侵入してきたプラスミドや,ファージ由来のDNAあるいはRNAを異物として認識し分解する.CRISPRは数十塩基からなる回文様配列(リピート)が同程度の長さのスペーサー配列を介して数回から数十回繰り返しているゲノム上の領域である.CRISPRの転写産物であるpre-CRISPR RNA(pre-crRNA)はリピート部分で分解されてスペーサー単位のcrRNAが生じ,それがある種のCasタンパク質に取り込まれてエフェクター分子となる.エフェクター分子はcrRNAと相補的な配列を持つ侵入DNAやRNAを分解する.このシステムは,システムを構成している遺伝子領域や分子の特徴によって大きく二つのクラスと六つのタイプに分類され,それぞれのタイプはさらにいくつかのサブタイプに細分類されている1).Class 1はエフェクター分子が多量体のもので,Type I,-III,-IVがこれに該当し,Type II,-V,-VIはエフェクター分子が単量体でありClass 2に含まれる.ゲノム編集のツールとして利用されているCas9はType IIのエフェクター分子であり,RNAの検出に利用されているCas13はType VIのエフェクター分子である.CRISPR領域の長さ,数,配列,および,タイプ,サブタイプは細菌によって異なり,複数のシステムを持つ細菌もいる.
Type IIIシステムでは,pre-crRNAはCas6タンパク質によりcrRNAに分解される.Cas6は各リピート由来部分の3′末端から8塩基上流を切断するので,crRNAは5′末端側にリピート由来の8塩基(5′-タグ)を持っている(後述).Type IIIはA~Fの六つのサブタイプに分類されている.いずれの場合も,エフェクター分子はCas10スーパーファミリーに属する分子を含む複数のサブユニットから構成されていることが特徴である.これらのサブタイプの中で,Type III-A,-Bの解析が進んでいる.Type III-A,-Bのエフェクター分子は,それぞれ,Csm複合体,Cmr複合体と呼ばれている.高度好熱菌Thermus thermophilus株の場合を例にあげると,Csm複合体は5種類のタンパク質(Csm1~5)とcrRNAから構成されている約430 kDaの分子で,サブユニットの構成比はCsm1123364251 : crRNA1(下付きの数字は分子数)である2).Cmr複合体は6種類のCmrタンパク質(Cmr1~6)とcrRNAから構成されている約360 kDaの分子で,サブユニットの構成比はCmr112131445361 : crRNA1である3, 4).両複合体分子の構造は類似しており,いずれもらせん状に位置したタンパク質にcrRNAが巻きついている.この構造はDNAを標的とするType Iのエフェクター分子であるCascade複合体にも共通している5).
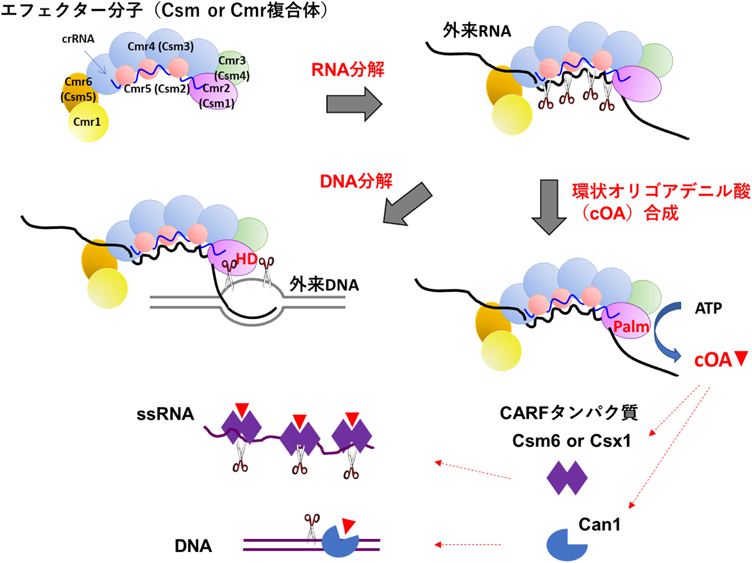
Csm複合体とCmr複合体は,これまで明らかにされている他のタイプのエフェクター分子にはないユニークな活性を持っている(図1).すなわち,Type I,-II,-VシステムはDNAを標的とし,Type VIシステムではRNAを標的とするのに対し,Type III-A,-Bシステムでは,DNAとRNAの両方を標的とする.Csm複合体とCmr複合体は,crRNAに対して相補的な配列を持つ外来のRNAに結合すると分子の中央部にらせん状に位置しているサブユニットであるCas7スーパーファミリータンパク質,すなわち,Csm複合体では6分子のCsm3, Cmr複合体では4分子のCmr4が持つRNase活性が惹起される.すると,外来のRNAは,crRNAの5′側から順に6塩基ごとに,Csm複合体の場合は6か所,Cmr複合体の場合は4か所切断される(5′ルーラーメカニズム).なお,T. thermophilus株のCmr複合体では5か所の切断が観察されているが,5か所目での切断の機構は明らかにされていない3, 4).複合体分子に外来のRNAが結合すると,RNase活性に加え,さらに二つの反応の活性化が起こる.これらの活性は,複合体を構成しているサブユニットの中で最も大きく,複合体の末端に位置しているサブユニットであるCas10スーパーファミリータンパク質,すなわち,Csm複合体ではCsm1, Cmr複合体ではCmr2が担っている.その一つは配列非特異的なDNase活性で,ssDNAを分解する.その活性中心はHDドメインにある6–8).このDNaseの活性化は,in vivoでは転写に依存していると考えられている.もう一つは,環状オリゴアデニル酸(cOA)を合成する活性で,その活性中心はPalmドメインにある.合成されたcOAはセカンドメッセンジャーとしてCARF(CRISPR-associated Rossmann fold)タンパク質に結合し,それらのRNase活性やDNase活性を活性化する.たとえば,T. thermophilus株やThermococcus onnurineus株ではcA4(四つのAMPからなる環状アデニル酸)が,Streptococcus thermophilus株やEnterococcus italicus株ではcA6が,それぞれ,Csm6タンパク質のRNase活性を活性化する9–11).T. thermophilus株では,さらに,cA4の結合によって活性化されたCan1がDNAにニックを導入する12).CARFタンパク質は侵入DNA/RNAのみならず細菌自身のDNA/RNAをも分解し,その結果,細胞死あるいは休眠が誘導され,DNA/RNAによる感染が種全体に及ぶことを回避する.
Type IIIシステムには,以下に述べるT. thermophilus株のCmr複合体のように,Cas10タンパク質のHDドメインが欠失しているためにDNase活性を持たないものもあるので1),Type IIIシステムの本来の標的はRNAであると考えられる.
3. Type IIIエフェクター複合体の活性化におけるcrRNAの役割
CRISPRシステムのエフェクター分子は,crRNAと相補的な侵入DNAあるいはRNAを分解する.ところが,crRNAをコードしている細菌自身のDNAはcrRNAに対して相補的な配列であるがエフェクター分子によって分解されない.CRISPRシステムには自身のゲノムと外部からの侵入DNA/RNAとを区別する仕組みがあるためである.たとえば,Streptococcus pyogenes株のCas9システムでは,crRNAと相補的な配列の20~30塩基上流にPAM(protospacer adjacent motif)配列:NGGを持つDNAが非自己として認識され分解される.複数の活性を誘導するType IIIシステムの場合はどうであろうか.最近,T. thermophilus株のCmr(TtCmr)を用いた解析で,Type III-Bエフェクター分子の活性化におけるcrRNAの役割が明らかになった(図2)13).
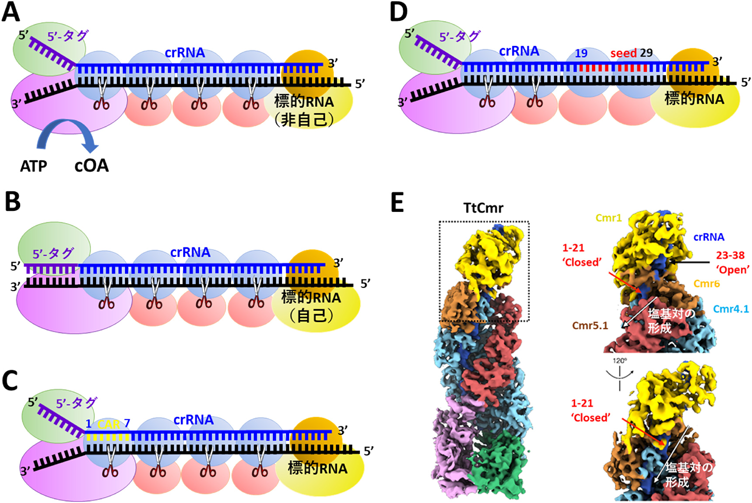
Type IIIシステムのcrRNAは,40塩基程度のCRISPRスペーサー由来の配列に加え,5′末端側にリピート由来の8塩基(5′-タグ)を含んでいる(上述).TtCmrは,スペーサー部分のみに対して相補的な配列を持つRNAと同様に,スペーサー部分と5′-タグの一部分とも相補的な配列を持つRNAをも分解した(図2A, B).すなわち,TtCmrは,非自己のRNAと同様,自己のRNAあるいはそれに類似した配列を持つRNAも分解することが強く示唆された.一方,前者の場合はcOAの合成が誘導されたが,後者の場合はそれが認められなかった(図2A, B).
crRNAのスペーサー部分と標的RNAとの塩基配列の相補性の程度もTtCmrの活性化に影響を及ぼしていた.すなわち,5′-タグの下流7塩基のうちの1塩基がミスマッチであると,6塩基目のミスマッチの場合を除いたすべての場合においてcOAの合成が抑制された(図2C).一方,それらのミスマッチRNAに対してもTtCmrのRNase活性は惹起された(図2C).すなわち,crRNAのスペーサー部分の5′側数塩基の配列と標的RNAとの相補性がcOA合成の活性化のために重要であるが,標的RNAを分解するためだけにはその厳密性は必要ではない.このcrRNAのスペーサー部分の5′側数塩基の部分はCas10-activating region(CAR)と名づけられている.crRNAの5′末端側と,その部分と塩基対を形成する標的RNAはCmr2, Cmr3,および,Cmr4サブユニットと相互作用している.CARと標的RNAとの塩基対が形成されるとそれらのサブユニットのコンホメーションがcOA合成の活性型へと変化するのかもしれない.
以上の結果,TtCmrは,わずかでも非自己である可能性があるRNA,すなわち,crRNAのスペーサー部分とある程度の相補性がある配列を持つRNAを応急的に分解するが,自己のRNAであることが確かな場合はセカンドメッセンジャーであるcOAの合成を抑制しCARFタンパク質(上述)を活性化させないことで,細胞死や休眠を回避していると考えられる.
一方,標的RNAの分解に必要な条件はcrRNAの3′末端側の部分との相補性の程度であることが強く示唆されている.すなわち,3′末端側の部分と標的RNAとの相補性が低いと標的RNAとの結合,および,RNase活性が低下する(図2D).この場合,crRNAと標的RNAとのミスマッチがcrRNAの3′末端側から数塩基以内に一つ存在していてもcOAの合成能には影響しない.ミスマッチの位置が3′末端から数塩基以上になるとcOAの産生は低下し,3′末端から15~20塩基程度(seed領域)の位置にミスマッチが存在するとcOAはほとんど産生されない.crRNA(46 nt)が結合したTtCmrの電子顕微鏡構造(図2E)をみると,crRNAの3′末端側(23~38 nt)は大きく露出しており標的RNAと結合しやすい状態にある.一方,seed領域を含む上流(5′側)の領域はCmr4とCmr5に挟まれており大部分が分子の内部に埋もれている.crRNAの3′末端側と標的RNAとが相補的に結合すると,crRNAの5′側に向かって二重鎖RNA(crRNA:標的RNA)が形成され,それがseed領域に達するとcrRNAが巻きついているTtCmr分子の中央部分のコンホメーションが変化し,分子の中央部に位置するCmr4のRNase活性が惹起されるのであろう4).
上述した理由でTtCmrにはDNase活性がない.DNaseの活性化はPyrococcus furiosus株のCmrで解析されており,標的RNAの配列のうち,crRNAと相補的な配列(protospacer)の3′側の3塩基の配列[protospacer flanking sequence(PFS),あるいは,rPAM](図3)が活性化に影響することが報告されている7, 14).一方,Type III-A Csm複合体では,標的RNAの配列がcrRNAのスペーサー由来の部分との相補性に加え5′-タグ部分との相補性がある程度保たれている場合はDNase活性を示さない(自己と認識する)ことが報告されている6).
Cmr複合体,および,Csm複合体における,crRNAの5′-タグ部分と標的RNAとの相補性によるDNase活性,および,cOA合成活性の制御機構の構造基盤は文献15)を参照されたい.
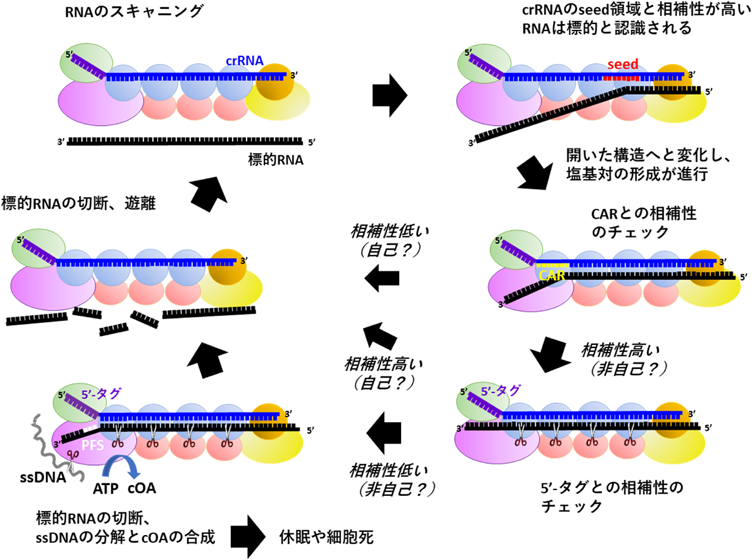
Cmrの作用機作のモデルを図3に示した.CmrはRNAの塩基配列をスキャンし,crRNAの3′末端側の領域と相補的な配列があるとそこで結合し,5′側に向かって標的RNAとの二重鎖を形成していく.二重鎖の形成がseed領域に達すると,Cmr複合体は開いた構造へとコンホメーションを変えながらcrRNAのさらに5′側(CAR)へ向かって二重鎖の形成が進行する.CARとの相補性が低いと標的RNAは3′側から順に分解され,エフェクターから遊離する.CARとの相補性が高い場合は,次に,5′-タグとの相補性をチェックする.相補性が低い標的RNAは非自己と認識され,cOA合成活性が活性化されて細胞死や休眠が誘導される.標的RNAのPFS配列に依存してDNase活性も活性化される.5′-タグとの相補性が高い場合は,標的RNAは分解されるが,自己の可能性が高いため,cOA合成,および,DNaseの活性化は起こらず,細胞死や休眠を回避している.
本稿で述べたType IIIシステムに限らず,他のTypeの場合も,生化学的・構造生物学的解析によってシステムを構成している個々の分子,特にエフェクター分子の作用機作に関する多くの知見が得られてきた.しかし,CRISPR-Casシステムの未解明な点は多く残されている.たとえば,DNA/RNAが細菌に侵入してからCRISPR-Casシステムが起動するまでの過程はほとんど明らかにされていない.複数のCRISPRシステムを持つ細菌がそれらをいかにして使い分けているのかも不明である.これらの点が細胞生物学的な観点からも明らかにされ,一つの細菌におけるCRISPRシステムの機能の全貌が解明されることを期待している.
TtCmrによるcOA合成反応と,合成されたcOAによるCARFタンパク質のRNaseの活性化(図1)を利用すると,SARS-CoV-2をPCR法と同等の感度で検出することができる13).しかも,高度好熱菌由来のタンパク質は耐熱性が高いため,高温で行う標的RNAの増幅反応からTtCmrとCARFタンパク質を用いた検出反応までを一つの試験管内で行うことができる.本システムが簡便なRNAの検出ツールとして広く利用されることを期待している.
謝辞Acknowledgments
本研究は,JSPS科研費25440013, 16K07285の助成を受け,オランダ・ワーゲニンゲン大学のRaymond Staals博士,John van der Oost博士,アメリカ・テキサス大学オースティン校のDavid Taylor博士,アメリカ・カリフォルニア大学バークレー校のJennifer Doudna博士をはじめとする多くの方々との共同で行われたものです.本研究に携わっていただいた方々に深く感謝致します.