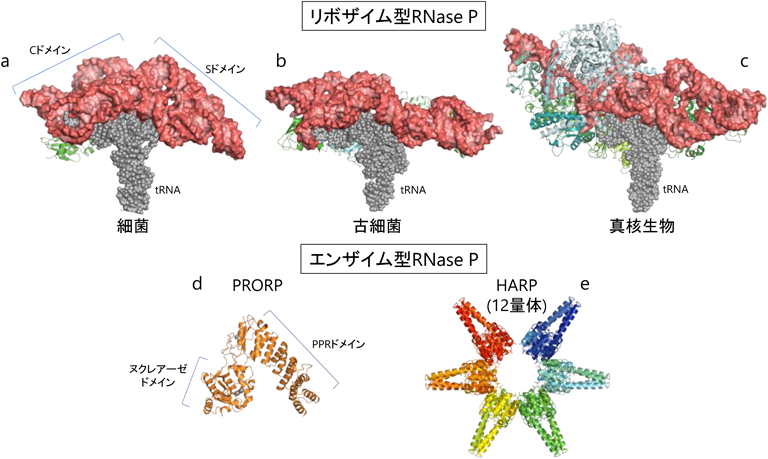
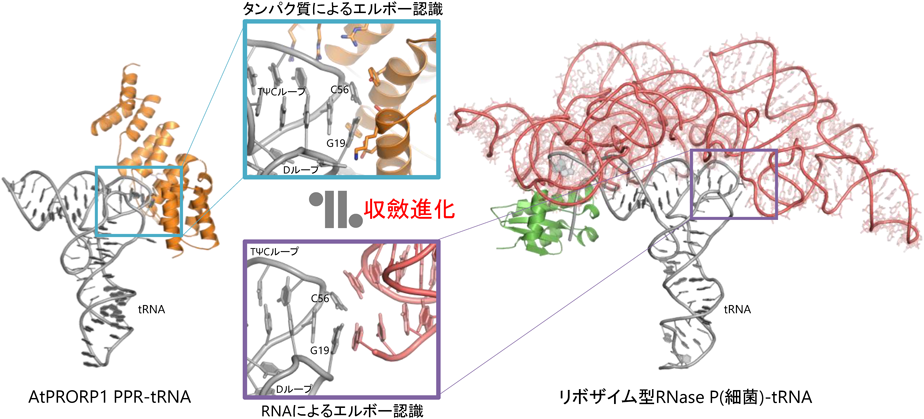
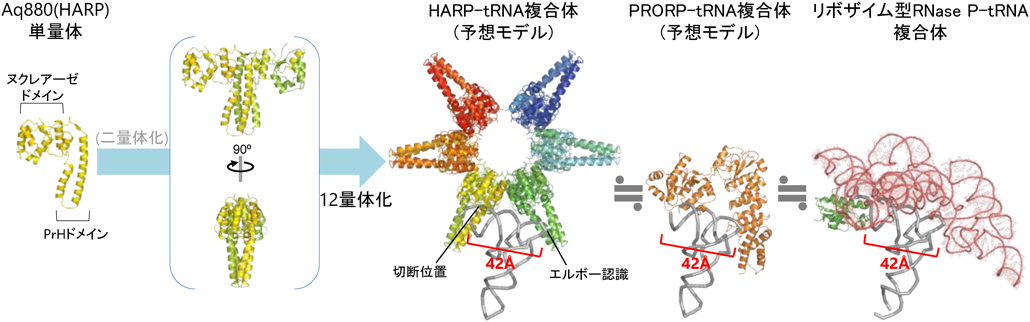
リボヌクレアーゼP(RNase P)の多様性とその構造基盤Diversity of Ribonuclease P and its structural insight
1 九州大学大学院農学研究院Faculty of Agriculture, Kyushu University ◇ 〒819–0395 福岡県福岡市西区元岡744 ◇ 744 Moto-oka, Nishi-ku, Fukuoka 819–0395, Japan
2 九州大学大学院生物資源環境科学府Faculty of Agriculture, Kyushu University ◇ 〒819–0395 福岡県福岡市西区元岡744 ◇ 744 Moto-oka, Nishi-ku, Fukuoka 819–0395, Japan