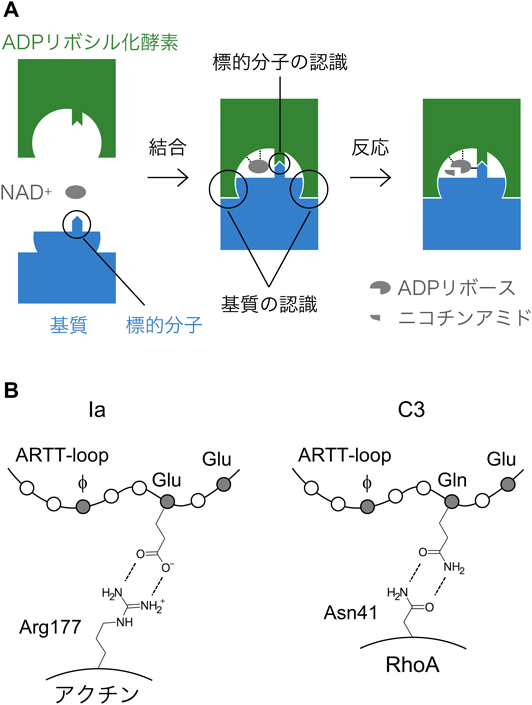
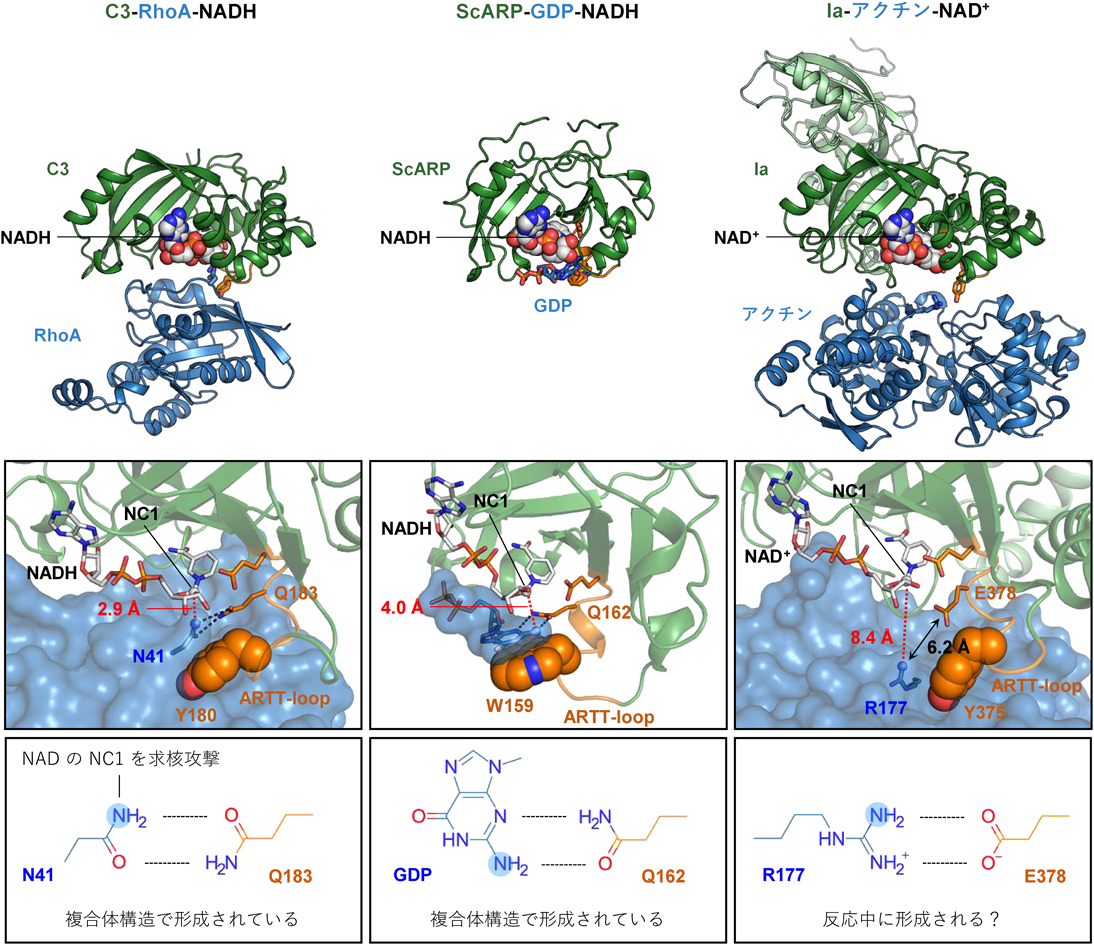
ADPリボシル化酵素がタンパク質やDNAを特異的に修飾する仕組みTarget recognition mechanism of protein- and DNA-targeting ADP-ribosyltransferases
1 日本女子大学理学部物質生物科学科Department of Chemical and Biological Sciences, Faculty of Science, Japan Women’s University ◇ 〒112–8681 東京都文京区目白台2–8–1 ◇ 2–8–1 Mejirodai, Bunkyo-ku, Tokyo 112–8681, Japan
2 京都産業大学生命科学部Faculty of Life Sciences, Kyoto Sangyo University ◇ 〒603–8555 京都市北区上賀茂本山 ◇ Kamigamo-motoyama, Kita-ku, Kyoto 603–8555, Japan