2000年前後にさまざまな生物の全ゲノム配列が次々と決定され,生体内の分子の「カタログ」がそろうようになると,従来の要素還元的な方向性だけではなく,システムレベルで生命を理解しようとする「システム生物学」的アプローチが現実的なものとして議論されるようになった1).生体システムの網羅的な生体情報をハイスループットに収集するオミクス技術は,近年の生命科学においてこのシステム生物学的アプローチを実現させた立役者である.特に,多検体の網羅的多次元情報を収集し,比較解析を行うスキームは,システム全体の要素構造と変動ダイナミクスを計測し,観察している生体機能に重要な生体要素を同定するために有効である.しかし,現行のオミクス解析技術は,transcriptome, proteome, metabolomeなど,細胞内の分子階層にある生体情報をターゲットにした技術がほとんどである.
筆者らは2010年ごろから,「個体レベルのシステム生物学」実現を目指した複数の技術開発を進めてきた.その中でもコアな技術として位置づけたのが,組織,臓器,全身の細胞や細胞ネットワークに関する構造・機能情報(細胞の種類,活動状態,空間的位置,細胞間相互作用など)をオミクススケールで収集するための網羅的解析技術である.想定される技術は,臓器や全身のすべての細胞が網羅的に検出できるだけでなく,他のオミクス技術と同様に多サンプルの比較解析が可能でなければならない.これが細胞・細胞ネットワークの階層にある生体情報をターゲットとする「セルオミクス」の基本コンセプトである.
近年は空間オミクスの技術が次々と発表され,細胞の情報を空間情報と組み合わせて収集するという考え方も技術も一般的となっているが,筆者らがこのセルオミクスのコンセプトの議論を始めた2010年ごろは,具体的な方向性も方法論も未確定の状態であった.この時期の先駆的な試みとして,理化学研究所・於保,横田らの自動シリアルセクショニング装置を応用した方法(Transcriptome Tomography)がある2).マウス脳のhorizontal, sagittal, coronalの直行する3平面で連続断面を収集し,各切片をマイクロアレイで遺伝子発現解析し,3次元的に再構築してボクセル単位で遺伝子発現プロファイル(発現密度データ)をとるという画期的なアイデアであった.一方,この技術は容易に予想されるように,多数のサンプルへの拡張性,コスト,切片厚に依存するボクセル解像度などの問題があり,セルオミクスの基本技術として利用するには難しさがあった.
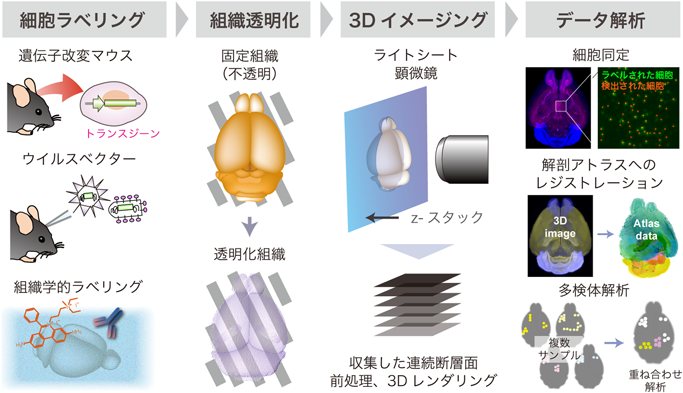
筆者らが代替的な方法を模索する中で,実現可能性の高い技術として浮上してきたのが組織透明化と高速な3次元イメージングを組み合わせて1細胞解像度の全臓器画像を取得するアプローチである.光学顕微鏡は数百nmから数十µm程度の解像度を持ち,組織中の細胞一つ一つを取得するモダリティとして最適である.問題は,生体組織が不透明で光が通らないことに起因する,光学顕微鏡での深部観察の困難さである.このため,一般的な組織学観察では,切片を作って内部の観察部位を露出させる必要がある.しかし,臓器全体を光が通るような透明化処理(組織学では古くから透徹とも呼ばれた)を行うと,3次元観察に適した光学顕微鏡を用いて全臓器・全身スケールの1細胞解像度画像が収集できると想定された.また,遺伝学的ツールや染色プローブによって生体情報を適切にラベリングしておけば,収集した3次元画像には細胞種,細胞活動,細胞ネットワークの構造などの情報が表現され,多サンプルの比較解析も実施可能だと想定された.すなわち,セルオミクスのフレームワークは①細胞ラベリング,②組織透明化,③1細胞解像度の光学3次元イメージング,④3次元画像解析,の組合わせによって実現されるという青写真が生まれた(図1).この青写真は一見荒唐無稽にもみえるかもしれない(実際,2013年ごろまでの初期開発期間中は,多くの批判的な意見にさらされた).しかし,2007年にDodtらが発表した,蛍光ライトシート顕微鏡を透明化組織と組み合わせてマウス脳全体やハエ全身を3次元撮像した論文3)は,この筆者らのアイデアが現実的に実装可能であることを強くサポートしていた.
本稿では組織透明化と3次元光学イメージングを組み合わせて実現したセルオミクスの開発経緯と代表的な応用例を紹介し,今後の展開について議論したい.各論についての詳しい情報は,近年優れた総説が複数出版されており4–6),興味のある読者は副読されたい.
組織透明化はサンプル内の光学特性(屈折率分布や光散乱体分布など)を均一にし,内部の光散乱や光吸収を抑制することで達成される.どの程度の透明度が必要かは,使用する顕微鏡に依存する.2光子顕微鏡は長波長パルスレーザーを使用して生体深部を励起可能であるため,透明度が高くなくてもmmオーダーのサンプルであれば十分観察可能である.一方,ほとんどの2光子顕微鏡はレーザースキャンニング型であるため,臓器スケールの3次元データ収集には相応の時間がかかる.このため,多数の臓器サンプルをスループットよく3次元撮影するセットアップとしては現実的でない.1光子のコンフォーカル顕微鏡では透明度をさらに上げる必要があり,また2光子顕微鏡と同様にレーザースキャンニング型のセットアップのため撮影時間の問題が生じる.
Dodtらの論文で示されたライトシート顕微鏡は,励起光をシート状に広げてサンプル中に光学断面を生成し,90度方向から2次元画像として撮影を行える顕微鏡である.サンプルをz方向に連続的に動かすことで,2次元画像の連続断層画像(zスタック画像)が容易に収集できるため,臓器全体のような大型サンプルでも現実的なタイムスケール(数十分から数時間)で3次元撮影が可能である.また撮影面だけを励起させるため,蛍光体の退色も最小限に抑えられる.一方,側方から励起光を照射し上方から撮影するというセットアップの特徴上,観察対象は光の散乱や屈折が内部で生じない(ほぼ)完全な透明体である必要がある.このため,セルオミクスを実施するための撮影画像の質を得るには,きわめて高い透明度が達成できる透明化法が必要である.
組織透明化技術は2010年代に入って急速に開発が進み,現在では数十のレシピやプロトコルが提案されている.これらの透明化試薬は大きく分けて三つのカテゴリに分類される5).1)有機溶媒を用いた試薬.透明化効率が非常に高く,短時間で透明化処理が完了する.一方,蛍光タンパク質のシグナル保持性や試薬の安全性の問題がある.また脱水を伴う処理のため,組織は収縮する.2)水溶性化合物を用いた試薬.安全性や蛍光シグナル保持性が高く,目的に応じて多様なレシピが調整できる.有機溶剤試薬に比べて透明度が低いものが多いが,組織の脱脂や脱色と組み合わせることで同等の高度な透明化も可能となった.3)組織を人工ゲルで固定する手法(hydrogel-tissue chemistry).CLARITY7)とその派生技術を含む.組織中の分子を強固に固定し,タンパク質のみならずRNAや小分子も観察可能としたり,加熱など厳しい化学的条件に耐えられる性質を利用したmultiplexラベリングを可能とする.一方で透明化の条件設定が難しく,専用のデバイス利用が推奨されている.
Dodtらの論文で使用されたBABB試薬(ベンジルアルコールと安息香酸ベンジルの混合試薬)は,1980年代に開発された古典的かつ代表的な有機溶媒透明化試薬の一つである.透明化効率が高い一方,蛍光タンパク質は数時間オーダーで急速に消退し,多サンプルの定量的な観察には不適であった.2011年,理化学研究所の濱,宮脇らは,水溶性化合物の尿素が組織透明化に寄与できることを発見し,BABBの蛍光消退の問題を回避できる水溶性透明化試薬Scaleの開発に成功した8).一方で,この当時最先端の水溶性透明化試薬でも,有機溶剤のような高度な透明化を達成することが難しく,ライトシート顕微鏡の3次元観察に適用することは難しいと考えられた.すなわち,セルオミクスの実施に必要な「高い透明度」「高いシグナル保持性」「現実的なタイムスケールで並列処理が可能」といった要件は,当時の透明化技術においてはトレードオフ関係にあった.
筆者らは,蛍光シグナル保持性や安全性が高い水溶性化合物による組織透明化技術が,セルオミクス実施要件に適合すると見立て,Scaleをベースに透明化効率を大幅に向上させることでこのトレードオフの解決を試みた.そこで,多数の候補化合物から有用な物質を選別する新規スクリーニング系を構築した.CUBICチームの岸野は,組織ブロックではなく組織をすり潰した懸濁液を用い,濁度の低下率を測定することで,各候補化合物の透明化活性を定量的に評価できる方法を考案した.1回目の開発では,Scaleに使用された3種類の化合物(界面活性剤,尿素類似体,多価アルコール)の各カテゴリに分類される合計40種類の化合物を対象とした.その結果,新たにアミノアルコールが高度な組織透明化に有効な化合物であることを発見し,Scaleのレシピを改良した透明化試薬(第1世代試薬:ScaleCUBIC-1, ScaleCUBIC-2)として報告した9).2回目の開発は,CUBICチームの田井中,村上らが中心となってさまざまなカテゴリの水溶性化合物を広く網羅する合計1600以上の化合物をスクリーニング対象として実施した10).その結果,再びアミノアルコールが最上位の組織透明化化合物として選択された.また,屈折率調整試薬(最終的に組織と溶液の光学特性を一致させ透明化を達成させる試薬)に適した化合物として,芳香族アミンが選別された.さらに,アミノアルコールは血液の色素であるヘムを除去する脱色化合物としても機能することが示された10, 11).以上のようなスケーラブルなスクリーニング方法の確立により,筆者らはノンバイアスに多数の化合物を定量的に評価し理想的な透明化試薬を開発することに成功した.現在では2回目の開発で作製した2種類の試薬(第2世代試薬:CUBIC-L, CUBIC-R)を,セルオミクス実施のための主要な透明化試薬として推奨している.
筆者らのCUBIC試薬の他,各種有機溶剤試薬,水溶性透明化試薬,人工ゲル透明化法でも,網羅的細胞検出・解析の実施例が報告されている.たとえばiDISCOは代表的な3次元染色・透明化手法で,マウス全脳のc-Fos解析に利用された(後述)12).CLARITYを用いて70個以上のマウス全脳データを収集し,ドーパミン神経回路を解析した例も報告された(後述)13).また,Ce3Dという,低分子アミドと造影剤(Histodenz)を組み合わせた水溶性透明化試薬を用いて,リンパ節内の多色染色を実施し,組織学的にサイトメトリーを実施した例も報告された14).
3. 要素技術2:臓器・全身スケールの3次元イメージング顕微鏡
高効率な組織透明化技術とライトシート顕微鏡によるハイスループットな細胞解像度の3次元イメージングは,セルオミクス実施のための最適な細胞検出系であると考えられた.実際,組織透明化技術と,透明化サンプルに最適化されたライトシート顕微鏡の開発は,各所で同時並行的に進んできた.代表的な有機溶剤透明化法の3DISCOを開発したErtürkらは,透明化組織用のライトシート顕微鏡を開発・販売するLaVision BioTecと協業し,動物全身やヒト臓器の3次元イメージングを含むアプリケーションを次々と成功させた15, 16).Hydrogel-tissue chemistryの代表的技術CLARITYを開発したDeisserothらは,CLARITYサンプルの高解像度撮影に適した専用ライトシート顕微鏡(CLARITY Optimized Light Sheet Microscopy:COLM)をリリースした17).臓器全体をカバーできるmmオーダーの広い視野範囲でz解像度を向上させる機構も提案されている.Gaoらはライトシートの最薄部をサンプル内で移動させながら擬似的にサンプル全体のz解像度を向上させるtiling light-sheet機構を提案した18).Fiolkaらはライトシートの最薄部の移動とcMOSカメラのシャッターの移動(ローリングシャッター)を同期させ,cMOSセンサー全体のシグナルがライトシート最薄部からのシグナルでカバーされるaxial sweep機構を提案した19).また,病理検体など平らな形状のサンプルを簡便に3次元撮影できるセットアップとして,上方または下方から斜めにライトシートを照射し撮像する顕微鏡(diSPIM, open-top light-sheet)も提案されている20, 21).Open-top light-sheet顕微鏡は実際にヒト病理検体の3次元観察に使用され,病理学的診断精度の向上に寄与することが示された21).
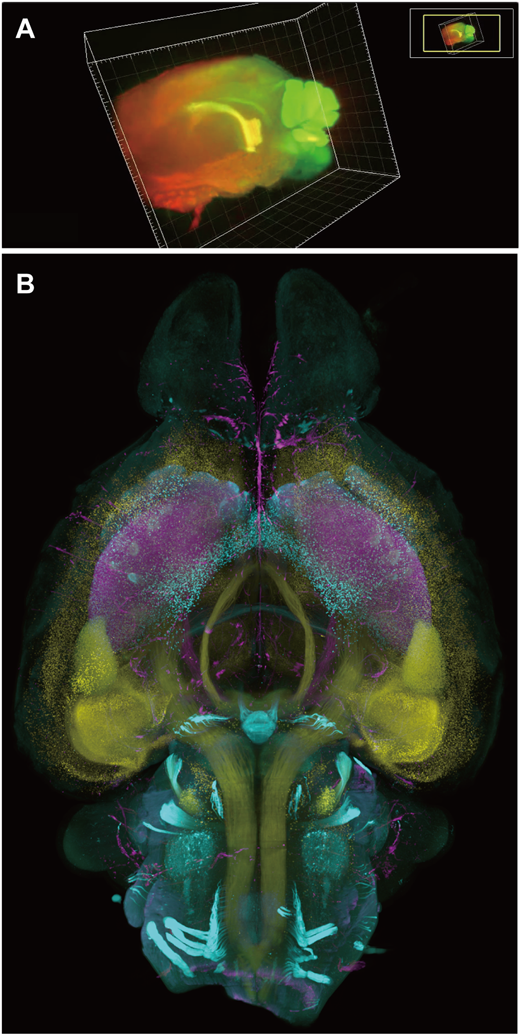
筆者らも,セルオミクス開発の経緯の中でさまざまなライトシート顕微鏡の改良や自己開発に取り組んできた.最初の組織透明化試薬が完成した2013年ごろ,筆者らは市販機として当時唯一の透明化組織用ライトシート顕微鏡だったLaVision BioTecのUltramicroscopyを日本で初めて導入し,プリミティブなマウス脳3次元データの取得に成功した(図2).しかし,メーカーのデフォルトの光学系ではサンプル全体をカバーできず,また球面収差も大きかったため,対物レンズやリレーレンズ系,サンプルホルダーなどを改良して最適化させた9).このような最初期の試行錯誤ののち,筆者らはライトシート顕微鏡を自己開発,またはメーカーと共同開発し,より最適な仕様のイメージング系を整備することに取り組んだ.大型サンプル用ライトシート顕微鏡は,透明化マウス全身を撮影できるスペックを追求した22).また,光学系を前後2軸に増築し,撮影シーケンスの最適化によりさらに高速な3次元撮影(~15分/マウス全脳)を可能としたライトシート顕微鏡も整備した23).この顕微鏡にはtiling light-sheet機構を組み込み,視野全体で高いz解像度を達成した(図2).また,CUBICチームの真野,村上が中心となり,高倍率・高開口数の対物レンズと組み合わせ,細胞が密な領域でもすべての細胞を分離して検出できる解像度を有するライトシート顕微鏡も整備された24).近年では,解像度と撮影スピードのトレードオフを解消するため,三谷,松本らがzステージを連続的に動かしながらzスタック画像を収集できるシステム(MOVIE顕微鏡)を開発した25).
使用する顕微鏡の種類にかかわらず,適切な透明化法,顕微鏡のセットアップ,対物レンズの仕様,および画像の解像度の選択が,実験ワークフロー全体の設計を最適化させるために重要である.一部の有機溶媒は揮発性が強く,また対物レンズの構造に使用されている接着剤を溶解させるため,フュームフード中に顕微鏡を設置し,有機溶剤耐性の部材を用いた顕微鏡の利用が必要である.オーバーサンプリングを避けつつ,実験・分析の目的に合致した解像度のデータを収集するため,適切な光学解像度を設定することも重要である.各種透明化試薬はそれぞれ最終的な屈折率が異なるが,おおむね1.5前後の高い屈折率を持つため,対物レンズが高屈折率サンプルに対応している必要がある.解像度を上げるほど,屈折率の不一致による球面収差は大きくなる.また,必要以上に解像度の高い高開口数の対物レンズは,動作距離が短いため3次元のボリュームイメージングが困難となり,深部での球面収差の問題も大きくなる.セルオミクスで対象とするサンプルは,アトラスなどのリソースデータを除き,比較的疎にラベルされた細胞を網羅的に検出することが要件となるため,低解像度で広視野をカバーするセットアップを用いることが多い.
4. 要素技術3:臓器・全身スケールの細胞ラベリング技術
透明化して何が観察できるかは,透明化する前に(あるいは透明化処理と組み合わせて)何をラベリングしたかに依存する.したがって,観察対象とする細胞・細胞ネットワークの構造や機能を適切なラベリングツールを用いて可視化することが,セルオミクス実施には重要である.大きく分けて1)遺伝学的ラベリング,2)組織学的ラベリングが細胞ラベリングに利用できる.
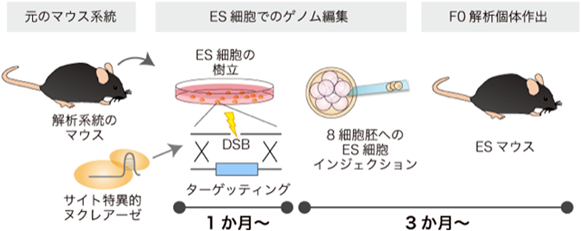
近年は発生工学やウイルスベクターツールの技術革新が進み,多様な遺伝学的ツールを短期間で生体内に導入することが可能となった.筆者らはセルオミクス実現の観点からマウス発生工学の次世代化に取り組み,遺伝子改変を施したマウスES細胞から最初の世代(従来のキメラマウスの世代)で解析個体を作出できる「交配なしの次世代マウス遺伝学」26)確立に貢献した(図3).従来のマウス発生工学による遺伝子改変マウス作出には複数のボトルネックがあった.一つ目はES細胞における遺伝子改変率,二つ目はES細胞から最初のキメラ個体を作製するときの作出率とキメラ率,三つ目は遺伝学的バックグラウンドをES細胞の系統(古典的には株化が容易な129系統など)から解析系統(C57BL/6など)に寄せるための複数回のバッククロスである.一つ目の問題は,近年のTALENやCRISPR等によるゲノム編集技術の大幅な向上により,従来数%程度だったターゲッティング率を数十%(チャンピオンケースでは100%近く)に改善することでクリアされた.二つ目と三つ目の問題は,3i培地などの新しいES細胞培養培地を用いて解析系統から直接ES細胞が作製できるようになったこと,さらには従来ES細胞のインジェクションに使用されてきた胚盤胞期の胚ではなく,より早い時期の8細胞期胚を用いることで,胚全身がES細胞由来の細胞で形成されるキメラ(100%キメラマウス,またはESマウスと呼ばれる)の作出が可能になったことにより解決された27).これらの新しい発生工学技術の組合わせにより,筆者らは次世代マウス遺伝学のワークフローを確立した.実証実験として,時計遺伝子Cry1/Cry2のダブルノックアウトマウスからES細胞を作出,さらに転写制御配列やコーディング配列にさまざまな改変や変異を加えたCry1の発現カセットをノックインするレスキュー実験を合計20系統にわたって実施し,個体レベルでの体内時計表現型を比較解析することに成功した28).この実験はダブルノックアウト+ノックインという5アレルの遺伝子改変個体を作出する必要があるため,従来のような交配ベースでの作出では実現不可能な実験であった.筆者らはさらに,セルオミクスの実証の目的で高輝度の赤色蛍光タンパク質発現マウス(3×mKate2ノックインマウス)を次世代マウス遺伝学で作出し,第1世代試薬によるマウス全脳イメージングのデモンストレーションに活用した9).現在では受精卵(1細胞胚)に直接CRISPRとターゲッティングベクターを導入して遺伝子改変する技術も開発が進んでおり,マウス遺伝学は従来のように何年もかけて解析個体を準備する状況が一変している.
発生工学による遺伝学的ツールの組込みに加え,神経科学など一部分野ではアデノ随伴ウイルス(adeno-associated virus:AAV)や狂犬病ウイルスをはじめとしたウイルスツールが充実し,発生工学に並ぶ有用な遺伝学的ラベリング技術を提供している.たとえば,GradinaruたちのCre recombination-based AAV targeted evolution(CREATE)によって改良されたAAVは,ウイルスを末梢から全身投与することで,特定の臓器全体への遺伝子導入を可能とする29).このため,古典的な遺伝子改変動物作出に類するシステミックな細胞ラベリングが実施可能である.
もう一つの重要な細胞ラベリング手法として,抗体や染色剤などで組織を染色する組織学的染色がある.一般的な組織学的染色では,染色面を切削により露出させ,切片にして施行する.しかし,セルオミクスでは臓器や全身スケールでのラベリングを実施するため,組織学的染色も3次元化が必要であった.実際,3次元組織学は透明化技術の利用や開発とほぼ並行して試みられてきた.1980年代には,Dentらがカエル胚の3次元観察のため,whole-mount免疫染色をBABB透明化試薬と組み合わせた例が報告された30).近年の透明化試薬開発でも,免疫染色への適合性が一つの重要なクライテリアとなってきた7–9, 31).しかし,3次元染色を2次元切片染色と同等に施行することは一般的に困難である.一番のボトルネックは,抗体や染色剤などの染色プローブの浸透の問題である.核染色剤などの小分子化合物でも,3次元サンプルの表面のみにトラップされ,内部が染色されないケースは頻繁に経験されるため,これは単純な分子量などの問題ではなく3次元染色系の複雑な物理化学的環境の問題であると想定された.
3次元染色の問題を解決するため,歴史的にさまざまなアプローチがテストされてきた.たとえば,脱脂,脱水,弱い固定法,プロテアーゼ処理などによる固定組織の透過処理は,ある程度の有効性が示された.現行の3次元染色技術として最も広く利用されているiDISCO12)は,DMSOやメタノールで透過処理した組織を使用して,比較的高効率な3次元染色を実施可能としている.また,尿素やSDSなどの添加剤を用いて浸透中の染色プローブと組織との間の結合親和性を調節する方法も示された.前者はScale技術を開発した濱らが,ChemScale, AbScaleとして報告した32).また後者はCLARITYを開発したChungらが,染色プローブの結合性をオフからオンに切り替えることで深部浸透を達成させるSWITCH法として報告した33).さらに,電気泳動や圧力などの物理的手法によって染色プローブの浸透性を上げる方法も試された34, 35).ChungらはSWITCHと電気泳動(電場を確率的に変動させて浸透の均一化を図るstochastic electrotransportと呼ばれる技術)を組み合わせ,最終的にeFLASHという高効率3次元染色技術を確立した36).
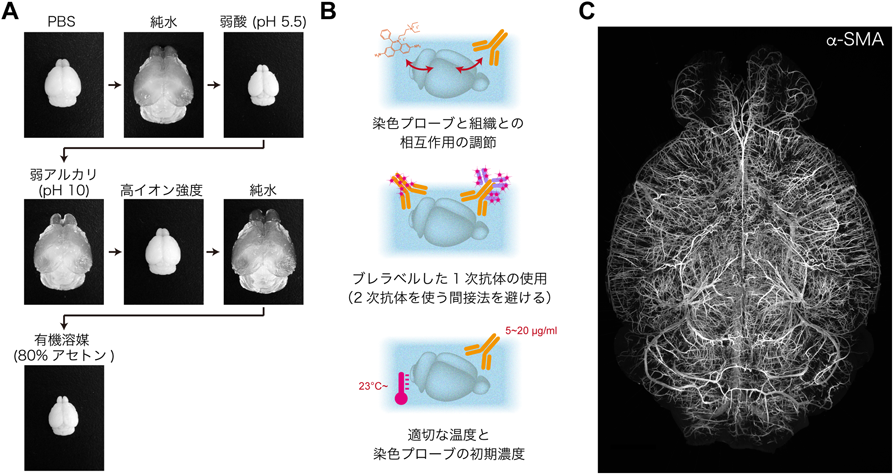
以上のような,さまざまな技術開発の経緯で浮かび上がってきたのは,1)経験的な検討が多く,設定・導入されたパラメーターが本当に3次元染色に有効なのかが定量的に評価されていない,2)有効とされるプロトコルでも効率や適用範囲が不十分,3)デバイスを使用する方法は導入ハードルが高い,などの問題点である.したがって,筆者らは従来の2次元切片染色から容易に拡張でき,多くの染色プローブに適合できる3次元染色技術の開発を目指した.その際,経験的なパラメーター設定に頼らず理想的なプロトコルをボトムアップに設計できるよう,3次元染色系の物理化学的環境,特に生体組織そのものの物理化学的物性を明らかにすることを目指した.
さまざまな生体組織の物性検討を行った結果,筆者らは,3次元観察のため固定し透明化処理を施した生体組織が,主にポリペプチドから構成される電解質ゲルの一種であることを突き止めた.この「組織ゲル」は,ある種の人工ゲル(実験では固定ゼラチンゲルを使用)と性質がきわめて似通っていたため,筆者らは人工ゲルを用いた3次元染色条件のスクリーニング系を新たに構築し,3次元染色に必須,あるいは不必要な条件を定量的に広く探索した.最終的に同定された必須条件は以下のようであった.1)組織と染色プローブとの結合性の調整(抗体染色の場合は,間接法を使用しない),2)染色プローブの初期濃度や染色温度などの物理化学的パラメーターの最適化.最終的に,筆者らはこれらの必須条件を組み合わせた3次元染色・イメージングのためのプロトコル「CUBIC-HistoVIsion(CUBIC-HV)」を構築し,マウス全脳や成体マーモセット半脳,1 cm3サイズのヒト病理脳検体など,多様なサンプルの3次元染色に成功した(図4)23).CUBIC-HVは広い用途に使用できる3次元染色技術として,30以上の抗体や染色剤への適合性を確認済みで,現在も適用範囲を広げている.また,本開発で同定された一連の必須パラメーターは,iDISCOやAbScaleなど他の3次元染色プロトコルの染色性向上にも寄与することが確認されており,一般性を持つという点は重要なポイントである.
5. 要素技術4:1細胞解像度の全臓器情報解析技術
セルオミクス実施のためには,マイクロアレイや次世代シーケンサーのインフォマティクス解析と同じような,3次元1細胞解像度画像から生体情報を取得するための解析フレームワークが必要である.筆者らが初期開発を進めていた2012~2013年ごろは,そのようなオミクススケールの網羅的細胞解析に対する解析フレームワークが存在していなかったため,自分たちでワークフローを設計し要素技術を開発する必要があった.まず,二つの脳のタンパク質発現の直接比較解析を実現するため,3次元全脳画像の重ね合わせとシグナルの差分計算を行うことを試みた.この目的のため,CUBICチームのPerrinが理化学研究所の横田らの研究室と共同で,医療画像に用いられるNIfTi形式の画像の重ね合わせと位置合わせ(レジストレーション)を行うためのソフトウェア37)を適用し,神経活動の下流でVenusレポーターが発現するトランスジェニックマウス(Arc-dVenusトランスジェニックマウス38))の全脳活動の比較解析を成功させた9, 39).この解析系の最新の適用例では,異なる10種類の抗体でそれぞれ染色した10個のマウス脳を重ね合わせ,疑似多色染色の3次元データを構築することにも成功した23).
複数の全脳データの重ね合わせからオミクス的な比較定量解析を実施するため,CUBICチームの幸永,Perrinらが中心となって,NMDA受容体阻害剤のMK-801を慢性投与したArc-dVenusトランスジェニックマウスから脳を時系列サンプリングし,全脳イメージングおよび解析を実施した40).合計8条件(薬物投与ありなし,6時間おきサンプリング,各群N=2~3)の全脳神経活動データをAllen Brain Atlasにマッピングし,脳領域ごとのラベル細胞数を定量した.この脳領域ごとの活動神経細胞量を多次元データとしてクラスタリング解析し,時間帯ごとに薬物に反応して活動した脳領域の抽出に成功した.さらなるワークフロー改良のため,CUBICチームでは領域ごとのマスクデータではなく,1細胞解像度で表現された脳アトラスの構築に取り組んだ.村上らは,撮像解像度を向上させるため組織を膨潤させつつ透明化する新しいプロトコル「CUBIC-X」を開発し,マウス全脳全細胞(約1億個)の座標情報を取得することに成功した.この座標情報にAllen Brain Atlasの解剖学的情報を付与することで,世界初の全脳1細胞解像度アトラス「CUBIC-Atlas」を構築することに成功した24).さらに近年,真野を中心にCUBIC-Atlasを基盤として多数の脳を1細胞解像度で解析するためのクラウドソフトウェア「CUBIC-Cloud」をリリースした41).CUBIC-AtlasとCUBIC-Cloudを使用することにより,ユーザーは自身で取得した複数のマウス全脳データをCUBIC-Atlas上にマッピングし,同一プラットフォーム上で1細胞解像度の定量的な比較解析を行うことが可能となる.論文中では,薬物投与による神経活動(c-Fosの全脳免疫染色)の比較解析や,視床下部ニューロペプチドの制御に関わる神経回路の雌雄差解析など,全脳3次元データでなければ実施不可能なセルオミクスの実践例を多数報告した.
上記のような全脳スケールの神経回路や神経活動解析はセルオミクスのよい適用例であり,ニーズも高い.筆者ら以外のグループからも類似のワークフローが提案されている.たとえば神経活動解析のフレームワークとして,iDISCO法を開発したRenierらが全脳c-Fos免疫染色と脳領域ごとの定量解析を実現するワークフロー(ClearMap)を報告した12).また,内田らは70個以上の狂犬病ウイルストレーサーでラベルしたマウス全脳を解析するワークフローを独自に構築し,ドーパミン神経回路を全脳解析した例を報告した13).さらに,神経活動解析と回路解析を組み合わせたフレームワークとして,DeisserothたちはCLARITYを応用し,特定の条件下で活動した神経の投射マッピングを行う手法(CAPTURE)を報告した42).近年では神経だけではなく全脳スケールの血管解析やアミロイドβ沈着などの病的プロセス解析も複数開発が進められており32, 41, 43),セルオミクス的な解析ワークフローは今後さらに適用範囲が広がると期待される.
近年,空間transcriptomicsをはじめとして,分子レベルのオミクス情報を組織中の空間的位置情報と組み合わせる技術が発展中である.発現分子の軸に加え位置情報の軸を多次元情報として収集可能であるが,3次元化に関しては100 µm程度の厚みの切片での部分的実施例にとどまるなど44),現時点ではハードルが高い.一方,現行のセルオミクスはシグナルの3次元的な空間情報取得は容易であるが,発現分子のチャンネル数についてはまだ改善の余地が大きい.しかしながら,限られた数の分子発現情報に,顕微鏡画像特有の細胞の形や大きさの情報,位置情報を加え,空間オミクス解析を実現させる試みも進行中である.水溶性化合物(フルクトース,尿素,グリセロール)から構成されるFUnGIを開発したRiosらは,8チャンネルの染色データに細胞形態情報と位置情報を加え,3次元1細胞解像度のオミクス解析を行うワークフロー(segmentation analysis by parallelization of 3D datasets:STAPL-3D)を報告した45).また,CLARITYを開発したChungらは,ヒト大脳オルガノイドの細胞種マーカー発現,微小環境,全体の構造等のマルチチャンネル情報を透明化・3次元イメージングによって収集し,発生段階やジカウイルス感染モデルにおけるマルチスケールオルガノイド解析手法(SCOUT)の開発を報告した46).これらのセルオミクス技術は現在の空間オミクス技術と融合する方向にあり,近い将来,組織中全細胞の統合的なオミクス技術が実現するだろう.
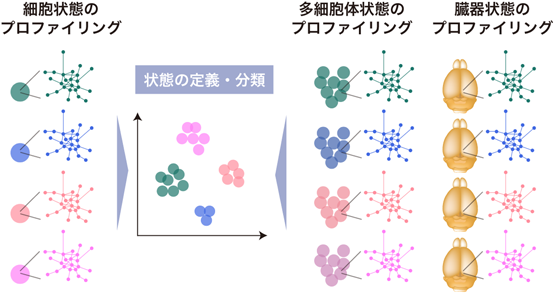
セルオミクスは一般的なシングルセル解析と同様に,各多細胞体サンプルごとに,ある種の「状態」を定義できる情報を計測し,多数の多細胞体サンプル集団のクラスタリング解析を行う方法も構築できると期待される(図5).この際必要となる生体情報は,たとえば分子のチャンネルが低次元(1~2分子)であったとしても,セルオミクスが得意とする空間位置情報を同時に取得することで,多数のサンプルについて状態クラスタリングが可能な多次元情報を与えうると考えられる.例として6種類の中枢作用性薬物を投与したマウス全脳のc-Fos免疫染色データを取得し,各脳をクラスタリング解析した例がある47).この例では,収集されている分子情報はc-Fosの1チャンネルだけだが,各薬物投与条件に対応する脳の活動領域(c-Fos発現の空間的位置)の情報が次元圧縮され独立主成分分析によってクラスタリングされうることを示した.また,筆者らは近年発表した総説の中で,オルガノイドのような小型の多細胞体ですべての細胞位置情報を取得した上で,数学的な形状解析等を適用することで,各オルガノイドの構造状態を定義・分類できる可能性を指摘した48).このような解析アプローチの実現によって,セルオミクスは現在のシングルセル解析のように,多細胞体を解析する基盤技術の一つとして展開できる可能性があると期待される.
セルオミクス技術の別の展開として,組織透明化と3次元イメージングが実現する次世代の病理学(3次元病理学)の実現も期待されている.もともと3次元的な構築を持つ生体組織を正確に客観的に評価するには,本来3次元的な情報の取得が求められる.たとえば,脈管など複雑な3次元空間上の構造や,膵臓ランゲルハンス島など分散して存在する構造体は,2次元切片の像からその正確な3次元情報を再構成することは不可能である.3次元病理学の実証例として,大阪大・野島らと筆者らの共同研究チームはヒト大腸がん患者の郭清リンパ節を3次元イメージングによって検証し,微小がん転移の検出力がどの程度向上するかを検討した.その結果,代表的な切片のみで評価する従来の病理検査では15%程度見逃しが発生するのに対し,3次元病理学のスキームでは検出感度が100%に向上した49).また,Glaser, Liuらのグループは前立腺がんの生検サンプルに対して2次元評価と3次元評価を比較した例を報告した.2名の病理医が独立に評価した結果,2次元評価では診断が一致しなかったのに対し,3次元評価では一致を見せた.この診断結果は手術適応の可否の判断にも関わるため,医療リソースや診療コスト,患者のQOLの観点からも,3次元病理学による診断能や客観性の向上は非常に重要であると考えられた.臨床病理分野では近年,機械学習と組み合わせた自動診断技術の報告も相次いでいるが,3次元病理学は教師データの作成や診断性向上にも寄与できると期待されている50).3次元病理学の近年の動向については最近の日本語総説にもまとめたので,興味がある読者はそちらも参照されたい51).
多くの研究者の貢献により,組織透明化技術や3次元細胞ラベリング技術は近年容易に利用可能となっている.一方,現在のセルオミクス実施におけるボトルネックの一つは,3次元観察を行うためのライトシート顕微鏡の普及であろう.近年の組織透明化技術の発展に後押しされ,複数のメーカーがライトシート顕微鏡の市販機をリリースするに至っている.しかし,その多くは高価(5000万円以上)で,1研究室が容易に購入できるものでないことは自明である.mesoSPIMなど,透明化組織観察用のオープンソースのライトシート顕微鏡も発表されているが(https://mesospim.org/)52),自作顕微鏡を自力で構築できる生物系研究者も少数であろう.たとえば,数百万円くらいで簡単に構築できるキット化された小型ライトシート顕微鏡は導入ハードルが比較的低く,普及に貢献できる可能性があると筆者は考えている.
また,データ解析は個別のアプリケーションに依存する部分もあり,標準化が難しい部分である.実際,現在では開発グループによって解析手法が分散している.しかしながら,CUBIC-Atlasのように1細胞解像度のデータ(あるいは1細胞解像度にたたみ込んだデータ)を前提としたワークフローなど,ある程度標準化されたワークフローが普及することにより,よりセルオミクスがさまざまなアプリケーションで利用される環境が構築されていくと期待される.また上記で議論したように,標準アトラスに依存しないサンプル間の標準化法や全細胞解析法の開発が進めば,動物組織に限らずオルガノイドや昆虫など他のモデル・非モデル生物にも展開が見込める.筆者らのCUBIC技術をはじめとして,透明化試薬,染色試薬,デバイス,解析ツールなどの多くの要素技術が製品化されたりオープンソースとして利用可能な環境となっており,これらの社会実装は技術を標準化し新規ユーザーが参入しやすい状況を醸成するのに一役買っている.
謝辞Acknowledgments
本稿で紹介したセルオミクス技術開発は東京大学・理化学研究所の上田泰己研究室を中心に実施され,医学,生物学,化学,光学,情報科学等の学際的な専門家の貢献によって達成されたものである.紙面上で全員を紹介できないことをご容赦願いたい.
引用文献References
1) Kitano, H. (2002) Systems biology: a brief overview. Science, 295, 1662–1664.
2) Okamura-Oho, Y., Shimokawa, K., Takemoto, S., Hirakiyama, A., Nakamura, S., Tsujimura, Y., Nishimura, M., Kasukawa, T., Masumoto, K.-H., Nikaido, I., et al. (2012) Transcriptome tomography for brain analysis in the web-accessible anatomical space. PLoS One, 7, e45373.
3) Dodt, H.U., Leischner, U., Schierloh, A., Jährling, N., Mauch, C.P., Deininger, K., Deussing, J.M., Eder, M., Zieglgänsberger, W., & Becker, K. (2007) Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain. Nat. Methods, 4, 331–336.
4) Yu, T., Zhu, J., Li, D., & Zhu, D. (2021) iScience 24, 102178.
5) Ueda, H.R., Erturk, A., Chung, K., Gradinaru, V., Chedotal, A., Tomancak, P., & Keller, P.J. (2020) Tissue clearing and its applications in neuroscience. Nat. Rev. Neurosci., 21, 61–79.
6) Ueda, H.R., Dodt, H.U., Osten, P., Economo, M.N., Chandrashekar, J., & Keller, P.J. (2020) Whole-brain profiling of cells and circuits in mammals by tissue clearing and light-sheet microscopy. Neuron, 106, 369–387.
7) Chung, K., Wallace, J., Kim, S.Y., Kalyanasundaram, S., Andalman, A.S., Davidson, T.J., Mirzabekov, J.J., Zalocusky, K.A., Mattis, J., Denisin, A.K., et al. (2013) Structural and molecular interrogation of intact biological systems. Nature, 497, 332–337.
8) Hama, H., Kurokawa, H., Kawano, H., Ando, R., Shimogori, T., Noda, H., Fukami, K., Sakaue-Sawano, A., & Miyawaki, A. (2011) Scale: a chemical approach for fluorescence imaging and reconstruction of transparent mouse brain. Nat. Neurosci., 14, 1481–1488.
9) Susaki, E.A., Tainaka, K., Perrin, D., Kishino, F., Tawara, T., Watanabe, T.M., Yokoyama, C., Onoe, H., Eguchi, M., Yamaguchi, S., et al. (2014) Whole-brain imaging with single-cell resolution using chemical cocktails and computational analysis. Cell, 157, 726–739.
10) Tainaka, K., Murakami, T.C., Susaki, E.A., Shimizu, C., Saito, R., Takahashi, K., Hayashi-Takagi, A., Sekiya, H., Arima, Y., Nojima, S., et al. (2018) Chemical landscape for tissue clearing based on hydrophilic reagents. Cell Rep., 24, 2196–2210.
11) Tainaka, K., Kubota, S.I., Suyama, T.Q., Susaki, E.A., Perrin, D., Ukai-Tadenuma, M., Ukai, H., & Ueda, H.R. (2014) Whole-body imaging with single-cell resolution by tissue decolorization. Cell, 159, 911–924.
12) Renier, N., Adams, E.L., Kirst, C., Wu, Z., Azevedo, R., Kohl, J., Autry, A.E., Kadiri, L., Venkataraju, K.U., & Zhou, Y. (2016) Mapping of brain activity by automated volume analysis of immediate early genes. Cell, 165, 1789–1802.
13) Menegas, W., Bergan, J.F., Ogawa, S.K., Isogai, Y., Umadevi Venkataraju, K., Osten, P., Uchida, N., & Watabe-Uchida, M. (2015) Dopamine neurons projecting to the posterior striatum form an anatomically distinct subclass. eLife, 4, e10032.
14) Li, W., Germain, R.N., & Gerner, M.Y. (2017) Multiplex, quantitative cellular analysis in large tissue volumes with clearing-enhanced 3D microscopy (Ce3D). Proc. Natl. Acad. Sci. USA, 114, E7321–E7330.
15) Cai, R., Pan, C., Ghasemigharagoz, A., Todorov, M.I., Forstera, B., Zhao, S., Bhatia, H.S., Parra-Damas, A., Mrowka, L., Theodorou, D., et al. (2019) Panoptic imaging of transparent mice reveals whole-body neuronal projections and skull-meninges connections. Nat. Neurosci., 22, 317–327.
16) Zhao, S., Todorov, M.I., Cai, R., Maskari, R.A., Steinke, H., Kemter, E., Mai, H., Rong, Z., Warmer, M., Stanic, K., et al. (2020) Cellular and molecular probing of intact human organs. Cell 180, 796–812.e19.
17) Tomer, R., Ye, L., Hsueh, B., & Deisseroth, K. (2014) Advanced CLARITY for rapid and high-resolution imaging of intact tissues. Nat. Protoc., 9, 1682–1697.
18) Gao, L. (2015) Extend the field of view of selective plan illumination microscopy by tiling the excitation light sheet. Opt. Express, 23, 6102–6111.
19) Chakraborty, T., Driscoll, M.K., Jeffery, E., Murphy, M.M., Roudot, P., Chang, B.-J., Vora, S., Wong, W.M., Nielson, C.D., Zhang, H., et al. (2019) Light-sheet microscopy of cleared tissues with isotropic, subcellular resolution. Nat. Methods, 16, 1109–1113.
20) Kumar, A., Wu, Y., Christensen, R., Chandris, P., Gandler, W., McCreedy, E., Bokinsky, A., Colón-Ramos, D.A., Bao, Z., McAuliffe, M., et al. (2014) Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nat. Protoc., 9, 2555–2573.
21) Glaser, A.K., Reder, N.P., Chen, Y., McCarty, E.F., Yin, C., Wei, L., Wang, Y., True, L.D., & Liu, J.T.C. (2017) Nat Biomed Eng 1, 0084.
22) Kubota, S.I., Takahashi, K., Nishida, J., Morishita, Y., Ehata, S., Tainaka, K., Miyazono, K., & Ueda, H.R. (2017) Whole-body profiling of cancer metastasis with single-cell resolution. Cell Rep., 20, 236–250.
23) Susaki, E.A., Shimizu, C., Kuno, A., Tainaka, K., Li, X., Nishi, K., Morishima, K., Ono, H., Ode, K.L., Saeki, Y., et al. (2020) Versatile whole-organ/body staining and imaging based on electrolyte-gel properties of biological tissues. Nat. Commun., 11, 1982.
24) Murakami, T.C., Mano, T., Saikawa, S., Horiguchi, S.A., Shigeta, D., Baba, K., Sekiya, H., Shimizu, Y., Tanaka, K.F., Kiyonari, H., et al. (2018) A three-dimensional single-cell-resolution whole-brain atlas using CUBIC-X expansion microscopy and tissue clearing. Nat. Neurosci., 21, 625–637.
25) Matsumoto, K., Mitani, T.T., Horiguchi, S.A., Kaneshiro, J., Murakami, T.C., Mano, T., Fujishima, H., Konno, A., Watanabe, T.M., Hirai, H., et al. (2019) Advanced CUBIC tissue clearing for whole-organ cell profiling. Nat. Protoc., 14, 3506–3537.
26) Susaki, E.A., Ukai, H., & Ueda, H.R. (2017) npj Systems Biology and Applications 3, 15.
27) Kiyonari, H., Kaneko, M., Abe, S., & Aizawa, S. (2010) Three inhibitors of FGF receptor, ERK, and GSK3 establishes germline-competent embryonic stem cells of C57BL/6N mouse strain with high efficiency and stability. Genesis, 48, 317–327.
28) Ode, K.L., Ukai, H., Susaki, E.A., Narumi, R., Matsumoto, K., Hara, J., Koide, N., Abe, T., Kanemaki, M.T., Kiyonari, H., et al. (2017) Knockout-rescue embryonic stem cell-derived mouse reveals circadian-period control by quality and quantity of CRY1. Mol. Cell, 65, 176–190.
29) Deverman, B.E., Pravdo, P.L., Simpson, B.P., Kumar, S.R., Chan, K.Y., Banerjee, A., Wu, W.-L., Yang, B., Huber, N., Pasca, S.P., et al. (2016) Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nat. Biotechnol., 34, 204–209.
30) Dent, J.A., Polson, A.G., & Klymkowsky, M.W. (1989) A whole-mount immunocytochemical analysis of the expression of the intermediate filament protein vimentin in Xenopus. Development, 105, 61–74.
31) Ke, M.-T., Fujimoto, S., & Imai, T. (2013) SeeDB: a simple and morphology-preserving optical clearing agent for neuronal circuit reconstruction. Nat. Neurosci., 16, 1154–1161.
32) Hama, H., Hioki, H., Namiki, K., Hoshida, T., Kurokawa, H., Ishidate, F., Kaneko, T., Akagi, T., Saito, T., Saido, T., et al. (2015) ScaleS: an optical clearing palette for biological imaging. Nat. Neurosci., 18, 1518–1529.
33) Murray, E., Cho, J.H., Goodwin, D., Ku, T., Swaney, J., Kim, S.Y., Choi, H., Park, Y.G., Park, J.Y., Hubbert, A., et al. (2015) Simple, scalable proteomic imaging for high-dimensional profiling of intact systems. Cell, 163, 1500–1514.
34) Lee, E., Choi, J., Jo, Y., Kim, J.Y., Jang, Y.J., Lee, H.M., Kim, S.Y., Lee, H.-J., Cho, K., Jung, N., et al. (2016) ACT-PRESTO: Rapid and consistent tissue clearing and labeling method for 3-dimensional (3D) imaging. Sci. Rep., 6, 18631.
35) Kim, S.Y., Cho, J.H., Murray, E., Bakh, N., Choi, H., Ohn, K., Ruelas, L., Hubbert, A., McCue, M., Vassallo, S.L., et al. (2015) Stochastic electrotransport selectively enhances the transport of highly electromobile molecules. Proc. Natl. Acad. Sci. USA, 112, E6274–E6283.
36) Yun, D.H., Park, Y.-G., Cho, J.H., Kamentsky, L., Evans, N.B., Albanese, A., Xie, K., Swaney, J., Sohn, C.H., Tian, Y., et al. (2019) Ultrafast immunostaining of organ-scale tissues for scalable proteomic phenotyping. bioRxiv.
37) Avants, B.B., Tustison, N.J., Song, G., Cook, P.A., Klein, A., & Gee, J.C. (2011) A reproducible evaluation of ANTs similarity metric performance in brain image registration. Neuroimage, 54, 2033–2044.
38) Eguchi, M. & Yamaguchi, S. (2009) In vivo and in vitro visualization of gene expression dynamics over extensive areas of the brain. Neuroimage, 44, 1274–1283.
39) Susaki, E.A., Tainaka, K., Perrin, D., Yukinaga, H., Kuno, A., & Ueda, H.R. (2015) Advanced CUBIC protocols for whole-brain and whole-body clearing and imaging. Nat. Protoc., 10, 1709–1727.
40) Tatsuki, F., Sunagawa, G.A., Shi, S., Susaki, E.A., Yukinaga, H., Perrin, D., Sumiyama, K., Ukai-Tadenuma, M., Fujishima, H., Ohno, R., et al. (2016) Involvement of Ca(2+)-dependent hyperpolarization in sleep duration in mammals. Neuron, 90, 70–85.
41) Mano, T., Murata, K., Kon, K., Shimizu, C., Ono, H., Shi, S., Yamada, R.G., Miyamichi, K., Susaki, E.A., Touhara, K., et al. (2021) CUBIC-Cloud provides an integrative computational framework toward community-driven whole-mouse-brain mapping. Cell Reports Methods, 1, 100038.
42) Ye, L., Allen, W.E., Thompson, K.R., Tian, Q., Hsueh, B., Ramakrishnan, C., Wang, A.C., Jennings, J.H., Adhikari, A., Halpern, C.H., et al. (2016) Wiring and molecular features of prefrontal ensembles representing distinct experiences. Cell, 165, 1776–1788.
43) Liebmann, T., Renier, N., Bettayeb, K., Greengard, P., Tessier-Lavigne, M., & Flajolet, M. (2016) Three-dimensional study of Alzheimer’s disease hallmarks using the iDISCO clearing method. Cell Rep., 16, 1138–1152.
44) Wang, X., Allen, W.E., Wright, M.A., Sylwestrak, E.L., Samusik, N., Vesuna, S., Evans, K., Liu, C., Ramakrishnan, C., Liu, J., et al. (2018). Three-dimensional intact-tissue sequencing of single-cell transcriptional states. Science, 361, eaat5691.
45) van Ineveld, R.L., Kleinnijenhuis, M., Alieva, M., de Blank, S., Barrera Roman, M., van Vliet, E.J., Martínez Mir, C., Johnson, H.R., Bos, F.L., Heukers, R., et al. (2021) Revealing the spatio-phenotypic patterning of cells in healthy and tumor tissues with mLSR-3D and STAPL-3D. Nat. Biotechnol., 39, 1239–1245.
46) Albanese, A., Swaney, J.M., Yun, D.H., Evans, N.B., Antonucci, J.M., Velasco, S., Sohn, C.H., Arlotta, P., Gehrke, L., & Chung, K. (2020) Multiscale 3D phenotyping of human cerebral organoids. Sci. Rep., 10, 21487.
47) Hansen, H.H., Perens, J., Roostalu, U., Skytte, J.L., Salinas, C.G., Barkholt, P., Thorbek, D.D., Rigbolt, K.T.G., Vrang, N., Jelsing, J., et al. (2021) Whole-brain activation signatures of weight-lowering drugs. Mol. Metab., 47, 101171.
48) Susaki, E.A. & Takasato, M. (2021) Perspective: extending the utility of three-dimensional organoids by tissue clearing technologies. Front. Cell Dev. Biol., 9, 679226.
49) Nojima, S., Susaki, E.A., Yoshida, K., Takemoto, H., Tsujimura, N., Iijima, S., Takachi, K., Nakahara, Y., Tahara, S., Ohshima, K., et al. (2017) CUBIC pathology: three-dimensional imaging for pathological diagnosis. Sci. Rep., 7, 9269.
50) Liu, J.T.C., Glaser, A.K., Bera, K., True, L.D., Reder, N.P., Eliceiri, K.W., & Madabhushi, A. (2021) Harnessing non-destructive 3D pathology. Nat. Biomed. Eng., 5, 203–218.
51) 洲崎悦生(2021)高精度のがん診断に向けた3次元組織病理学の現在と未来.実験医学増刊,39, 1982–1988.
52) Voigt, F.F., Kirschenbaum, D., Platonova, E., Pages, S., Campbell, R.A.A., Kastli, R., Schaettin, M., Egolf, L., van der Bourg, A., Bethge, P., et al. (2019) The mesoSPIM initiative: open-source light-sheet microscopes for imaging cleared tissue. Nat. Methods, 16, 1105–1108.
著者紹介Author Profile
 洲﨑 悦生(すさき えつお)
洲﨑 悦生(すさき えつお)順天堂大学大学院医学研究科生化学・生体システム医科学教授.博士(医学).
略歴1977年熊本県生まれ.2002年九州大学医学部卒業,07年同大学院医学系学府で博士(医学)取得後,理化学研究所,東京大学を経て21年4月より現職.
研究テーマと抱負専門は生化学,分子細胞生物学,システム生物学,遺伝学,薬理学,バイオイメージング.独自に開発した網羅的組織解析技術,次世代遺伝学技術等を駆使して,多細胞システム生物学・合成生物学的アプローチを確立させ,個体老化,がん,神経変性疾患を含む老化関連疾患研究に応用する最先端研究を総力戦で推進中.
ウェブサイトhttps://twitter.com/suishess
趣味芸術全般,アクアリウム,ローラースケート(COVID19対応中).大学時代はクラシックギターを習っていました.