カブトガニは節足動物門のうち,クモ・サソリ・ダニなどを含む鋏角亜門(Chelicerata)に属し,現存種としては北アメリカ東岸と中米ユカタン半島沿岸に1種(Limulus polyphemus),アジア東南海域沿岸に3種(Tachypleus tridentatus, Tachypleus gigas, Carcinoscorpius rotundicauda),日本では瀬戸内海と北九州の沿岸に1種(T. tridentatus)が生息している1).かつてカブトガニの祖先は古生代に繁栄した三葉虫類であるとされていた.しかし,硬質部だけでなく軟質部まで良好に保存性されていることで有名なバージェス頁岩動物群(Burgess Shale fauna)(約5億500万年前:カナダコロンビア州の古生代カンブリア紀の化石動物相)の研究が進み2),1988年にはSanctacaris uncata(鉤爪を持つ聖なるエビ)3, 4),次いで2017年にはHabelia optata(ハベル山の願い)5)が鋏角類として同定された.三葉虫類は鋏角類の祖先ではなく,カンブリア紀には,それぞれ別の亜門に属して共存していたのである.
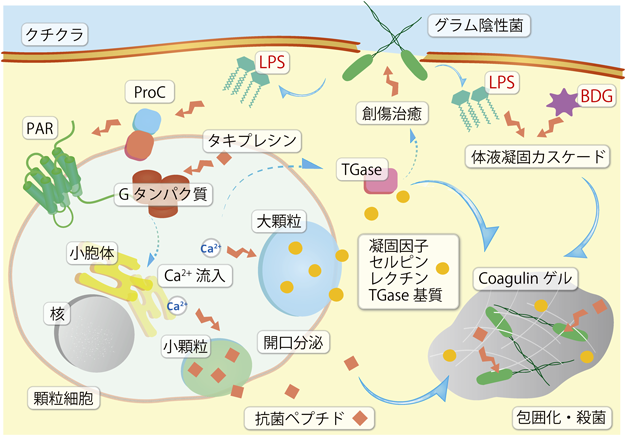
カブトガニ(T. tridentatus)は,受精卵の中で4回の胚脱皮を行い,孵化後,毎年脱皮を繰り返して少なくとも15年をかけて成熟する1).その体液は,哺乳類の血液とリンパ液に相当することから,血リンパと呼ばれる.カブトガニには,酸素運搬に働く赤血球に相当する血球はなく,ヘモグロビンと機能的に類似するヘモシアニンが高濃度(約100 mg/mL)で血リンパに溶けている1, 6, 7).ヘモシアニンは,酸素運搬だけでなく抗菌ペプチドと相互作用することで活性酸素の産生を誘導し,カブトガニの免疫系において重要な働きをしている8, 9).一方,血リンパには1種類の血球細胞が存在し,機能的には哺乳類の白血球や血小板に類似しており,顆粒細胞と呼ばれる10–12).その顆粒細胞の特徴は,グラム陰性菌の細胞壁成分であるリポ多糖(lipopolysaccharide:LPS)に鋭敏に反応し,各種の免疫関連因子を開口分泌(LPS-induced exocytosis)してさまざまな免疫応答を引き起こすことである.なかでも,分泌されたセリンプロテアーゼ前駆体がLPSや藻類や真菌の細胞壁成分であるβ-1,3-D-グルカン(β-1,3-D-glucan:BDG)と相互作用して惹起する体系凝固カスケードは,他に類をみない13, 14).このカスケードが起動すると,傷口からの体液の流出が阻止されると同時に感染微生物はレクチンにより凝集され,抗菌ペプチドで殺菌される.さらに,顆粒細胞のサイトゾルから分泌されたトランスグルタミナーゼ(transglutaminase:TGase)は,開口分泌されたTGase基質を架橋化することで創傷治癒を促進する(図1).
本稿では,カブトガニの体系凝固カスケードの分子機構を中心に紹介するが,特にLPS存在下で自己触媒的に活性化されるセリンプロテアーゼ前駆体の分子機構に迫りたい.紹介する内容は,すべて日本産カブトガニT. tridentatusを用いた研究成果である.なお,レクチン15–23),抗菌ペプチド24–35),TGase36–41)については,それぞれの原著論文や総説13, 14)を参照していただきたい.
顆粒細胞のサイトゾルには大・小の密度の異なる顆粒があって,大顆粒には凝固因子・レクチン・セリンプロテアーゼインヒビター・TGase基質,小顆粒には抗菌ペプチドなどが貯蔵されている10–14).開口分泌される凝固因子群には,セリンプロテアーゼ前駆体のProchelicerase C(ProC),Prochelicerase G(ProG),Prochelicerase B(ProB),proclotting enzyme(ProCE),および凝固タンパク質前駆体のCoagulogenが含まれる.これらのプロテアーゼ前駆体については,これまでProCEを除いてそれぞれFactor C, Factor G, Factor Bと呼んできたが,名称が哺乳類の補体系因子と重複していること,さらにはカブトガニにも補体系因子のホモログの存在が判明したことから42–44),筆者らは混乱を避けるために2018年に改名した45).
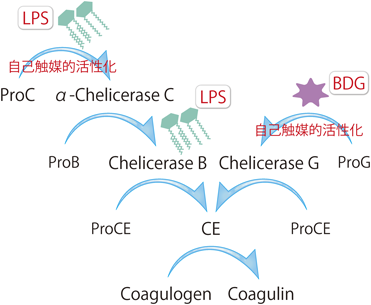
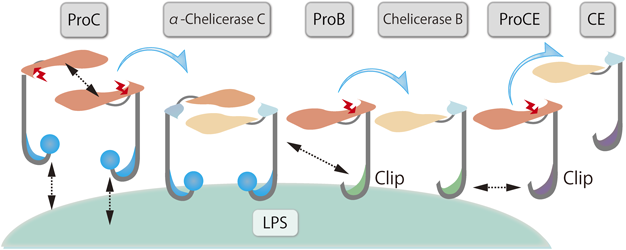
ProCはLPS存在下で自己触媒的に活性化されてα-Chelicerae Cとなり,ProBをChelicerae Bへと活性化し,次いでChelicerase BがProCEをclotting enzyme(CE)へと活性化し,最終的にはCEがCoagulogenをCoagulinに変換し,Coagulinが重合することでゲル化する(図2)13, 14, 46).一方,ProGはBDG存在下で自己触媒的にChelicerase Gに活性化されて,ProBを介さずに直接ProCEをCEへと変換する13, 47–52).また,ProCがin vitroにおいてキモトリプシンにより限定切断を介して活性化されたものは,β-Chelicerase Cとしてα-Chelicerae Cと区別した53).特筆すべきことは,β-Chelicerase Cは合成ペプチド基質に対してα-Chelicerase Cと同等のアミダーゼ活性を示すが,β-Chelicerase CのN末端領域にある–Tyr40–Cys41–間のペプチド結合はキモトリプシンにより二次的に切断されており,その結果LPS結合性を失って天然基質であるProBを活性化しないことである53).
顆粒細胞にあるLPSセンサーは,細胞表面に局在するProCであることが判明している54).ProCの特異抗体で顆粒細胞を染色すると,大顆粒だけでなくProCが細胞表面に局在していることが観察される55).開口分泌の分子機構を概説すると,ProCがLPSセンサーと応答し,活性化したα-Chelicerae Cがプロテアーゼ活性型Gタンパク質共役受容体(protease-activated G protein-coupled receptor:PAR)を限定切断する.その結果,新たに生じたPARのN末端のヘキサペプチド配列が内在性リガンドとなってPARを活性化し(この内在性ペプチド配列をtethered ligandという),三量体Gタンパク質を介した情報伝達系によりサイトゾルのCa2+濃度を上昇させ,開口分泌を誘導する(図1)54).この現象は,哺乳類の血小板のトロンビン受容体のPAR1を介した活性化と類似している56).トロンビンはカブトガニ顆粒細胞の開口分泌を誘導することはないが,PAR1やPAR2のtethered ligandであるヘキサペプチド(Ser–Phe–Leu–Leu–Arg–Asn, Ser–Leu–Ile–Gly–Arg–Leu)は,カブトガニ顆粒細胞の開口分泌を引き起こした54).また,ハチ毒の両親媒性ペプチドであるマストパランは,直接にGタンパク質と相互作用して血小板などの開口放出を誘導することが知られているが,事実,マストパランはカブトガニ顆粒細胞の開口分泌を惹起する54).
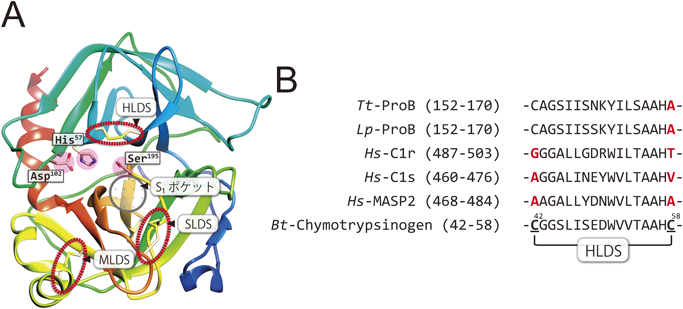
ProCの自己触媒的活性化の分子機構を解説する前に,セリンプロテアーゼ前駆体活性化の基本的な分子機構を確認しておきたい.セリンプロテアーゼ前駆体の活性化は,トリプシンによるキモトリプシノーゲンの限定切断を介した分子機構で説明される59, 60).キモトリプシノーゲンの–Arg15–Ile16–のペプチド結合がトリプシンにより限定切断されると,切断により新たに生じたNH2–Ile16–Val17–Asn18–Gly19–の配列がGly19付近で折れ曲がり,His57,Asp102,およびSer195で構成される触媒トライアドの裏側にある活性化誘導ポケット(activation pocket)と呼ばれる疎水的な穴に潜り込む.そして挿入されたNH2–Ile16–のα-アミノ基が,分子内部にあるAsp194側鎖のβ-カルボキシ基とイオン結合[塩橋(salt bridge)]を形成する.その結果,特にMet192残基周辺のコンホメーション変化を誘導し,基質結合部位(基質のP1部位側鎖を認識する部位のこと,S1ポケットと呼ばれる)の疎水的な壁が完成し,さらにオキシアニオンホール(酵素・基質の反応中間体である四面体中間体を一時的に捕捉する部位で,Gly193とSer195の主鎖アミノ基で形成される)が完成して,活性型キモトリプシンへと変換される.一方,触媒トライアドを含めた他の部位のコンホメーション変化は,ほとんど生じていない.
ところで,–Arg15–Ile16–の限定切断を受けずに活性型に変換されるセリンプロテアーゼ前駆体が報告されている.この限定切断を介さないセリンプロテアーゼ前駆体の活性化に関して,拙子の学位論文から引用させていただくと,黄色ブドウ球菌由来の単純タンパク質であるスタフィロコアグラーゼ(プロテアーゼや酵素活性はない)が,ヒトプロトロンビンに特異的に結合して,スタフィロコアグラーゼのアミノ末端配列(NH2–Ile1–Val2–Thr3–)をプロトロンビンの活性化誘導ポケットに挿入すると,プロトロンビンは限定切断を経ることなく活性型のスタフィロトロンビン(プロトロンビンとスタフィロコアグラーゼの1対1の複合体)へと変換される61–66).少なくともキモトリプシノーゲン活性化におけるNH2–Ile16–のα-アミノ基を介した塩橋形成と同じ作用をするものがあれば,プロトロンビンは限定切断を介することなく活性型プロテアーゼに変換されることが結晶構造学的にも証明されている67).したがって,セリンプロテアーゼ前駆体の活性化の本質は,–Arg15–Ile16–の限定切断そのものではなく,その切断により誘導されたコンホメーション変化によるS1ポケットとオキシアニオンホールの完成にある.
5. ProCのLPS存在下での自己触媒的活性化の分子機構
1)ProCのLPS結合部位の同定
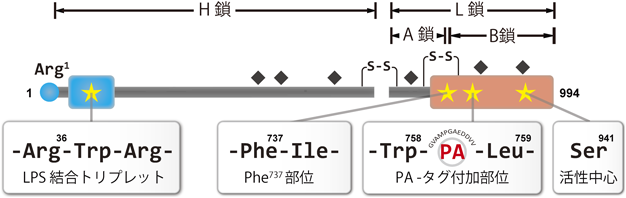
ProCは,994個のアミノ酸残基からなる一本鎖の前駆体,あるいは1か所にニックが入ったH鎖とL鎖からなる二本鎖の前駆体で開口分泌される(図3)68).顆粒細胞を用いて精製されたProCは,通常二本鎖前駆体である.筆者らは大腸菌と酵母の発現系を用いて,野生型ProC(WT-ProC)の調製法を模索したが,可溶性のWT-ProCの調製が困難で実用には至らなかった.N結合型糖鎖の存在がWT-ProCのフォールディングに不可欠であることが報告されていたので69),筆者らはHEK293細胞株を用いて,ProCのドメインごとに組換えタンパク質を調製し,ビオチン-LPSとストレプトアビジンを用いてLPS結合ドメインをスクリーニングした55).その結果,N末端ドメインのProC1-166が反応した.さらにそのドメイン内の–Arg36–Trp37–Arg38–の二つのArgをGlu残基に置換した変異体R36E/R38E-ProC,およびTrp37をAlaに置換した変異体W37A-ProCは,いずれもLPS結合性を失うことから,LPS結合にこの配列(LPS結合トリプレット)が必須であることが判明した(図3)55).
2)ProCの自己触媒的活性化におけるN末端Arg1の重要性
次に筆者らは,HEK293S変異株を用いて,一定の長さのN結合型糖鎖(Man5GlcNAc2)が付加した全長のProCを調製した70).得られた組換えProCは,LPS存在下で自己触媒的に活性化しなかった.アミノ酸配列分析の結果,N末端のArg1残基にベクター由来のトリペプチドGlu-Thr-Gly-が付加していた(+ETG-ProC)(表2).そこで,発現ベクターのArg1のN末端側にProCのプロペプチド部分の塩基配列を挿入したところ,培養細胞中でプロペプチドが正常にプロセシングされ,天然ProCに匹敵するWT-ProCの調製に成功した70).また,Arg1をLysに置換したR1K-ProCは,WT-ProCと同様にLPS依存的に活性化するが,Ala, Leu, Hisに置換したR1A-ProC, R1L-ProC, R1H-ProCについては,いずれもLPS存在下で活性化せず,N末端のArg1(あるいはLys1)が自己触媒的活性化に必須であることが判明した70).興味深いことに,SPRで測定されたこれらすべての置換体のLPSに対する結合親和性は,WT-ProCと比較してわずかに減少したのみであった(表1).したがって,WT-ProCの自己触媒的活性化においては,そのLPS結合トリプレットとLPSの結合は必要ではあるが,N末端のArg1が必須であることを示している.
表2 ProCの自己触媒的活性化に及ぼすArg1周辺のアミノ酸の欠失と付加の影響70)| WT-ProCおよび変異体 | アミノ末端側の配列 | 相対活性(%) |
|---|
| WT-ProC | Arg1–Gly2–Val3–Asp4–Leu5– | 100 |
| +ETG-ProC | Glu–Thr–Gly–Arg1–Gly2–Val3–Asp4–Leu5– | 0 |
| +RTG-ProC | Arg–Thr–Gly–Arg1–Gly2–Val3–Asp4–Leu5– | 0 |
| +R-ProC | Arg–Arg1–Gly2–Val3–Asp4–Leu5– | 30 |
| ΔGly2-ProC | Arg1–Val3–Asp4–Leu5– | 0 |
| ΔGly2-Val3-ProC | Arg1–Asp4–Leu5– | 0 |
| ΔGly2-Val3-Asp3-ProC | Arg1–Leu5– | 0 |
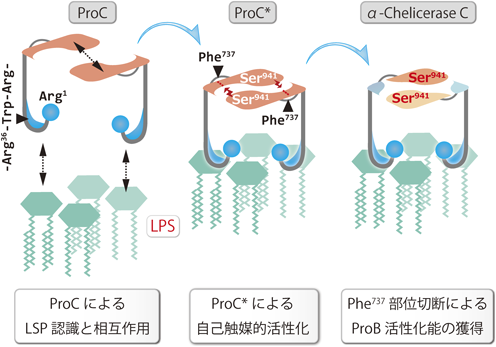
さらに,Arg1が存在しても,Arg1とLPS結合トリプレット間のアミノ酸を欠失した変異体は,LPS存在下で自己触媒的に活性化せず,また,Arg1に1残基Argを付加した変異体(+R-ProC)は,LPS存在下での自己触媒的活性化が70%も減少した(表2)70).したがって,自己触媒的活性化には,Arg1とLPS結合トリプレット配列との距離も重要である.LPS存在下でWT-ProCが自己触媒的活性化を起こす際は,WT-ProCが二量体,あるいは二量体からなる多量体を形成していることが,化学架橋剤を用いて明らかにされ,この多量体形成にN末端のArg1が必須であることも判明した70).複数のProCから提供されたArg1が新たなLPS結合部位となってProC多量体を安定化し,自己触媒的活性化を誘導している可能性も否定できない.
3)LPS存在下でのProC遷移状態の捕捉
ProCは,キモトリプシノーゲンの–Arg15–Ile16–に相当する–Phe737–Ile738–(Phe737部位)がLPS存在下で自己触媒的に限定切断され,活性型のα-Chelicerase Cへと変換される(図3)68).その際,「限定切断を受けていないが,活性型コンホメーションを有するProCの遷移状態(ProC*)」の存在を仮定する必要がある.α-Chelicerase Cは,ArgやLys残基を好むトリプシン族に属しており,事実,P1部位にArgを有する合成ペプチド基質t-butoxycarbonyl(Boc)-Val-Pro-Arg-methylcoumaryl-7-amide(MCA)を特異的に切断するが,キモトリプシンの合成ペプチド基質succinyl-Ala-Ala-Pro-Phe-MCAは切断しない53).しかし,自己触媒的活性化を介したProCからα-Chelicerase Cへの変換には,Phe737部位の限定切断が必要とされるため,さらに「ProC*はPhe737部位を切断する内在的なキモトリプシン様活性を有する」という仮定をする必要があり,また「ProC*のPhe737部位は自身の触媒活性により切断されるのか,ProC*の分子間で切断されるのか」を解明する必要があった.
前述のキモトリプシノーゲン活性化の分子機構に従うと,LPS存在下のProC*は,分子内あるいは分子間で,内在性キモトリプシン様活性を介してPhe737部位を自己触媒的に限定切断する.その結果,NH2–Ile738–Trp739–Asn740–Gly741–のペプチド配列は,Gly741付近で折れ曲がって活性化誘導ポケットに潜り込み,NH2–Ile738のα-アミノ基がAsp194に相当するAsp940の側鎖β-カルボキシ基と塩橋を形成することでS1ポケットとオキシアニオンホールが完成し,α-Chelicerase Cへと変換される.実験的には,ProCからα-Chelicerase Cへの変換は,Phe737部位の切断に伴ってL鎖からB鎖が遊離することをB鎖の抗体を用いてウェスタンブロットで判定できる(図3).また,α-Chelicerase Cのアミダーゼ活性をBoc-Val-Pro-Arg-MCAで測定することもできる.
4)ProC*の内在的キモトリプシン様活性を仮定する必要はなかった
キモトリプシノーゲンの触媒残基Ser195に相当するProCのSer941をAlaに置換した変異体S941A-ProCは,LPS依存的な自己触媒的活性化を誘導しないことから,ProC*においてもSer941が触媒部位として機能していることが確認された45).そこで,ProC*の「内在性のキモトリプシン様活性」を確認する目的で,Phe737部位をAlaやGluに置換したF737A-ProCとF737E-ProCは,予想に反してWT-ProCと同様にLPS存在下で自己触媒的活性化を引き起こした.このことは,ProC*によるPhe737部位の限定切断の際には,Phe737の側鎖は認識に重要でない,あるいは認識されていないことを示している.一方,Phe737を二次構造のブレーカーであるPro残基に置換した変異体F737P-ProCにおいては,LPS依存的な限定分解が起こらず,自己触媒的活性化も生じなかった45).LPS存在下でProC*に生じたPhe737部位周辺のコンホメーション変化が,Phe737部位の側鎖認識に依存しない限定切断に重要なのかもしれない.これらの実験結果から,Phe737部位の限定切断には,ProC*の内在的キモトリプシン様活性を仮定する必要がないことが判明した.
5)Phe737部位の限定切断はProC*分子間で生じる
変異体S941A-ProCは触媒残基であるSer941を失っているが,Phe737部位は正常である.一方,変異体F737P-ProCのSer941は正常であるが,Phe737部位は構造的に限定切断に抵抗する.二つの変異体をLPS存在下で混合した場合,F737P-ProCが遷移状態のF737P-ProC*となり,分子間で自己触媒的な限定切断が起こるとすれば,S941A-ProCのPhe737部位がF737P-ProC*により限定切断されるはずである.LPS存在下で,S941A-ProC:F737P-ProCのモル比を50:1, 50:2, 50:5で混合すると,F737P-ProCの量に比例してS941A-ProCのPhe737部位が切断され,遷移状態F737P-ProC*の存在を実証する結果となった45).
WT-ProCのN末端だけでなく,C末端に余分なアミノ酸配列,たとえばHis-タグを導入すると,LPS存在下での自己触媒的活性化が阻害される45, 70).しかし,PA-タグ(ポドプラミンに由来する12残基のペプチド)をProCのB鎖内部に導入したPA-ProC(図3)は,LPS存在下で自己触媒的に活性化することが判明した45).PA-タグは,導入したタンパク質に対して立体障害を与えにくいとされる.そこで,S941A-ProCのB鎖にPA-タグを導入したPA-S941A-ProCを調製した.LPS存在下でPA-S941A-ProCとF737P-ProCを1:1で混合すると,F737P-ProC*が生じてPA-S941A-ProCのPhe737部位が限定切断され,B鎖の遊離がPA-タグ抗体で確認された.以上の結果は,ProC*が分子間でPhe737部位を限定切断できることを証明している.しかし,本来,F737P-ProC*はPhe737部位が切断されない変異体であり,残念ながらProC*が「分子内ではPhe737部位を切断しない」という証明には至っていない.
6)ProC*どうしの分子間反応にはLPSの足場が必要である
前述のように,R36E/R38E-ProCはLPS結合活性を失い,LPS依存的な自己触媒的活性化を誘導しない55).そこで,LPS結合活性と触媒活性の両方を欠失させたR36E/R38E-S941A-ProCに対して,LPS存在下でF737P-ProCを1:1で混合したが,R36E/R38E-S941A-ProCのPhe737部位はF737P-ProC*により限定切断されなかった45).このことは,ProC*が基質であるProCを分子間で自己触媒的に切断するには,基質側ProCもLPSに結合している必要があることを示唆している.ProCがLPSと結合することで,そのPhe737部位が遷移状態のProC*により限定切断を受けやすいコンホメーションへと変換されるのであろう.現在のProCの自己触媒的活性化の分子機構のモデルは,LPSを足場としてProC*がArg1とLPS結合性トリプレットを介して二量体(あるいはその多量体)として集合し,そこでProC*どうしの分子間反応によりα-Chelicerase Cへと変換されるイメージで表現される(図4).
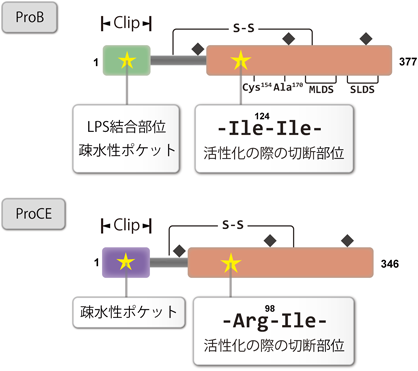
6. ProBとProCEの活性化におけるClipドメインの役割
ProBとProCEは同じドメイン構造をしており71),N末端側領域にProCやProGにみられないClipドメインを有している(図5).Clipドメインは三つのジスルフィド結合を有するユニークなドメインとして,最初にProCEに見いだされたもので72),その後,昆虫のプロフェノール酸化酵素系の構成タンパク質にもClipドメインが存在することが判明した73).最近になって,ProBはカブトガニ体液凝固系において,ProCに次いで第二のLPS結合性タンパク質であることが判明した(表1)53).また,LPS非存在下ではWT-ProBはWT-α-Chelicerase Cにより活性化されないこと,LPS存在下でもLPS結合性を欠失しているβ-Chelicerase CではWT-ProBは活性化されないことから,WT-ProBの活性化には反応の場としてLPSが必須であることも判明した53).その後,Clipドメインに含まれる5個の塩基性アミノ酸残基は,WT-ProBのLPS結合や,WT-α-Chelicerase CによるWT-ProBの活性化に重要であることが判明した74).さらに,Clipドメインには疎水性ポケットが存在するが(図5),疎水ポケットの変異体V55A-ProBのLPS結合性は失われ,WT-α-Chelicerase Cによる活性化の効率も野生型と比較して減少した74).
一方,WT-ProCEにはLPS結合性はみられなかったが,そのClipドメインの疎水性ポケットの変異体V53A-ProCEに対するWT-Chelicerase Bのkcat/Km値は,WT-ProCEと値と比較して1/7に減少していた74).一方,V55A-Chelicerase BとV53A-CEの特異的な合成ペプチド基質(Boc-Leu-Thr-Arg-MCA)に対するアミダーゼ活性は,それぞれの野生型酵素と比較して遜色はなかった.ProBのClipドメインは,LPSと結合してProBとα-Chelicerase Cの近接を助けるとともに,ProCEのClipドメインは,何らかの相互作用を介してChelicerase BがProCEを効率よく活性化する際に機能していると考えられる(図6).
7. ジスルフィド結合の導入によるWT-ProBの高機能化
セリンプロテアーゼの触媒ポケット周辺には,ヒスチジンループ(His-loop disulfide bridge:HLDS),メチオニンループ(Met-loop disulfide bridge:MLDS),セリンループ(Ser-loop disulfide bridge:SLDS)の三つのジスルフィド結合が保存されている(図7A)75, 76).ProBにおいては,HLDSを構成するCys(キモトリプシノーゲン番号ではCys58)がAlaに置換されており(図5),HLDSが進化的に欠損している.失われたHLDSの意義を解明する目的で,ProBのHLDSを復元した変異体(ProB-murasameと命名)を調製し,酵素動力学解析を行った77).その結果,活性化型のChelicerase B-murasameは,WT-Chelicerase Bと比較して,ペプチド基質や天然基質であるWT-proCEに対してkcat/Kmが約10倍ほど上昇し,熱安定性も格段に向上した.さらにカブトガニ凝固カスケードのスカベンジャーである3種のセリンプロテアーゼインヒビターSerpin-178),Serpin-279),およびSerpin-380)に対して,WT-Chelicerase Bは阻害されなかったが,Chelicerase B-murasameはSerpin-2により阻害された.
このHLDSの欠失は,アメリカ産カブトガニL. polyphemusのProB(accession No. XP_013784210)でも生じている(図7B).日本産T. tridentatusとアメリカ産L. polyphemusの分岐時期は,Coagulogenの配列比較により1億3000万年前と推定されており81),おそらくSanctacarisやHabeliaの体液凝固系においても,Chelicerase Bの触媒活性を低めに制御しておくことが何らかの生理機能や生存戦略に寄与していたのかもしれない.興味深いことに,HLDSの欠失は,ヒトの補体系に属するセリンプロテアーゼ前駆体のC1r82, 83),C1s84),MASP285)でも生じており,これらの補体因子においては,HLDSを構成する両方のCysが他のアミノ酸に置換されている(図7B).HLDSの欠失は,凝固系や補体系のように創傷局所で活性化するカスケード反応の制御に何らかの必要性があったのかもしれない.
カブトガニ体液凝固と哺乳類血液凝固のカスケードは,創傷部位の局所で惹起されるセリンプロテアーゼ前駆体活性化の連鎖反応という点では,機能的に類似した生体防御戦略である.一方,Coagulogenは,結晶構造解析によりフィブリノーゲンの相同タンパク質ではなく,神経成長因子のホモログであることが解明された86).カブトガニの体液レクチンであるタキレクチン-5が,フィブリノーゲンのホモログであることも判明している21, 23).また,ProB, ProCE, Coagulogenは,キイロショウジョウバエDrosophila melanogasterのToll経路を構成するSnake, Easter, ProSpätzleとそれぞれ相同であり,カブトガニ体液凝固系とDrosophila Toll経路は,こられの3因子カセットを保持しながら機能的に異なるカスケード反応に進化したことが推定される87).Drosophila Toll経路は,発生時の背腹軸決定に関与するだけでなく,成虫の自然免疫を誘導する経路でもあり,ProSpätzleから変換されたSpätzle二量体がToll受容体の内在性リガンドとして機能する88).すなわち,感染微生物が侵入すると,Toll受容体を介して細胞内にシグナルが伝達され,転写因子NF-κBが核内へ移行し,各種抗菌ペプチドが産生される.カブトガニにもToll様受容体89),さらにはNF-κBやその抑制因子であるIκBが存在しており,グラム陰性菌の感染はIκBの分解を引き起こし,NF-κBが核内へ移行した後,NO合成酵素やProCの転写を誘導する90).しかし,カブトガニのToll様受容体の自然免疫系への関与は不明のままである.
鋏角亜門に属する動物のゲノム解析が進むにつれて,カブトガニのProC, ProB, ProCEは,サソリ・クモ・ダニにもそれらのホモログが存在することが判明した.たとえば,ダニのProCホモログは補体系の因子として機能しており91),カブトガニProCも血リンパでは補体C3因子と複合体を形成して補体系でも機能している43, 44).おそらく,カブトガニの祖先は,鋏角亜門で共通の補体因子であったProCを借用して,独自の体液凝固カスケードを構築したのであろう.一方,ProGのホモログは,他の鋏角亜門に属する種からは見つかっていない92).ProGのBDG認識ドメインは,土壌細菌Cellvibrio mixtusのエンドグルカナーゼ5Aに含まれるセルロース結合ドメインと立体構造がぴったりと重なることが判明している52).したがって,ProGは,カブトガニの祖先がサソリ・クモ・ダニとの共通祖先から分岐した後に,土壌細菌から水平伝播した遺伝子と融合して誕生したセリンプロテアーゼ前駆体であると推定される.
本稿で解説したように,LPS存在下での自己触媒的活性化における遷移状態のProC*の存在を証明できたことは大きな成果であった.要約すると,i)自己触媒的なPhe737部位の限定切断は,ProC*因子どうしの分子間反応である.ii)Phe737部位の限定切断に対して,ProC*の内在的キモトリプシン様活性を仮定する必要はない.iii)LPSを足場としたProCの多量体形成は,ProC*のS1ポケットやオキシアニオンホールを完成させるだけでなく,限定切断されるPhe737部位周辺のコンホメーション変化を誘導する.iv)ProCの多量体形成には,Arg1およびLPS結合トリプレットが必須である.多量体の複数のArg1が新たなLPS結合部位を形成して,自己触媒的活性化を誘導しているのかもしれない.v)α-Chelicerase-CがProBを活性化する際には,ProBがそのClipドメインを介してLPSに結合している必要がある.vi)ProCEのClipドメインにはLPS結合性はないが,Chelicerase BがProCEを活性化する際にはClipドメインが重要な働きをする.
最近になって,ProC, ProB, ProCEの組換え凝固因子を用いてカブトガニ体液凝固カスケードが再構成され,天然材料に頼ることのない次世代のLPS検査試薬として上市されるに至った93).また,上述したように機能性を高めたProB-murasameも調製され77),さらなる高品質のLPS検査試薬が製品化されるであろう.
筆者らは,カブトガニの体液や顆粒細胞から免疫関連分子を精製し,それらの組換え体を用いて,昭和,平成,令和と中断することなく自然免疫の分子機構を研究してきた.しかし,免疫関連分子の生理機能解明のために,遺伝子ノックアウトやノックダウンの手法をカブトガニに適応することはできなかった.一方では,カブトガニTGaseやその基質群は,無脊椎動物で最初に報告されたものであるが36–41),キイロショウジョウバエをモデルとしてTGaseの生理機能の解明を推進してきた.その結果,TGaseは,外骨格形成,腸管の囲食膜形成,腸管での常在細菌の恒常性維持に重要な役割を担っていることが明らかになってきた94–99).
引用文献References
1) 山崎柄根(1999)カブトガニの生物学増補版(関口晃一編),制作同人社.
2) Collins, D. (2009) Misadventures in the Burgess Shale. Nature, 460, 952–953.
3) Briggs, D.E.G. & Collins, D. (1988) A middle Cambrian chelicerate from mount Stephen, British Columbia. Palaeontology, 31, 779–798.
4) Legg, D.A. (2014) Sanctacaris uncata: The oldest chelicerate (Arthropoda). Naturwissenshaften, 101, 1065–1073.
5) Aria, C. & Caron, J.-B. (2017) Mandibulate convergence in an armoured Cambrian stem chelicerate. BMC Evol. Biol., 17, 261.
6) Takagi, T. & Nemoto, T. (1980) Tachypleus tridentatus hemocyanin: Separation and characterization of monomer subunits and studies on sulfhydryl groups. J. Biochem., 87, 1785–1793.
7) van Holde, K.E., Miller, K.I., & Decker, H. (2001) Hemocyanins and invertebrate evolution. J. Biol. Chem., 276, 15563–15556.
8) Nagai, T. & Kawabata, S. (2000) A link between blood coagulation and prophenol oxidase activation in arthropod host defense. J. Biol. Chem., 275, 29264–29267.
9) Nagai, T., Osaki, T., & Kawabata, S. (2001) Functional conversion of hemocyanin to phenoloxidase by horseshoe crab antimicrobial peptides. J. Biol. Chem., 276, 27166–27170.
10) Copeland, D.E. & Levin, J. (1985) The fine structure of the amebocyte in the blood of Limulus polyphemus. I. Morphology of the normal cell. Biol. Bull., 169, 449–457.
11) Toh, Y., Mizutani, A., Tokunaga, F., Muta, T., & Iwanaga, S. (1991) Morphology of the granular hemocytes of the horseshoe crab Tachypleus tridentatus and immunocytochemical localization of clotting factors and antimicrobial substances. Cell Tissue Res., 266, 137–147.
12) Shigenaga, T., Takayenoki, Y., Kawasaki, S., Seki, N., Muta, T., Toh, Y., Ito, A., & Iwanaga, S. (1993) Separation of large and small granules from horseshoe crab (Tachypleus tridentatus) hemocytes and characterization of their components. J. Biochem., 114, 307–316.
13) Kawabata, S. & Muta, T. (2010) Sadaaki Iwanaga: Discovery of the lipopolysaccharide- and β-1,3-D-glucan-mediated proteolytic cascade and unique proteins in invertebrate immunity. J. Biochem., 147, 611–618.
14) Kawabata, S. (2010) Immunocompetent molecules and their response network in horseshoe crabs. In Invertebrate Immunity (Söderhäll, K., ed.), pp. 122–136, Springer Science+Business Media, New York.
15) Saito, T., Kawabata, S., Hirata, M., & Iwanaga, S. (1995) A novel type of limulus lectin-L6. Purification, primary structure and antibacterial activity. J. Biol. Chem., 270, 14493–14499.
16) Okino, N., Kawabata, S., Saito, T., Hirata, M., Takagi, T., & Iwanaga, S. (1995) Purification, characterization, and cDNA cloning of a 27-kDa lectin (L10) from horseshoe crab hemocytes. J. Biol. Chem., 270, 31008–31015.
17) Saito, T., Hatada, M., Iwanaga, S., & Kawabata, S. (1997) A newly identified horseshoe crab lectin with binding specificity to O-antigen of bacterial lipopolysaccharides. J. Biol. Chem., 272, 30703–30708.
18) Inamori, K., Saito, T., Iwaki, D., Nagira, T., Iwanaga, S., Arisaka, F., & Kawabata, S. (1999) A newly identified horseshoe crab lectin with specificity for blood group A antigen recognizes specific O-antigens of bacterial lipopolysaccharides. J. Biol. Chem., 274, 3272–3278.
19) Beisel, H.-G., Kawabata, S., Iwanaga, S., Huber, R., & Bode, W. (1999) Tachylectin-2: Crystal structure of a specific GlcNAc/GalNAc-binding lectin involved in the innate immunity host defense of the Japanese horseshoe crab Tachypleus tridentatus. EMBO J., 18, 2313–2322.
20) Iwaki, D., Osaki, T., Yoshimitsu, M., Wai, S.N., Iwanaga, S., & Kawabata, S. (1999) Functional and structural diversities of C-reactive proteins present in horseshoe crab hemolymph plasma. Eur. J. Biochem., 264, 314–326.
21) Gokudan, S., Muta, T., Tsuda, R., Koori, K., Kawahara, T., Seki, N., Mizunoe, Y., Wai, S.N., Iwanaga, S., & Kawabata, S. (1999) Horseshoe crab acetyl group-recognizing lectins involved in innate immunity are structurally related to fibrinogen. Proc. Natl. Acad. Sci. USA, 96, 10086–10091.
22) Nagai, T., Kawabata, S., Shishikura, F., & Sugita, H. (1999) Purification, characterization, and amino acid sequence of an embryonic lectin in perivitelline fluid of the horseshoe crab. J. Biol. Chem., 274, 37673–37678.
23) Kairies, N., Beisel, H.-G., Fuentes-Prior, P., Tsuda, R., Muta, T., Iwanaga, S., Bode, W., Huber, R., & Kawabata, S. (2001) The 2.0-Å crystal structure of tachylectin 5 provides evidence for the common origin of the innate immunity and the blood coagulation systems. Proc. Natl. Acad. Sci. USA, 98, 13519–13524.
24) Nakamura, T., Furunaka, H., Miyata, T., Tokunaga, F., Muta, T., Iwanaga, S., Niwa, M., Takao, T., & Shimonishi, Y. (1988) Tachyplesin, a class of antimicrobial peptide from the hemocytes of the horseshoe crab (Tachypleus tridentatus). J. Biol. Chem., 263, 16709–16713.
25) Kawano, K., Yoneya, T., Miyata, T., Yoshikawa, K., Tokunaga, F., Terada, Y., & Iwanaga, S. (1990) Antimicrobial peptide, tachyplesin I, isolated from hemocytes of the horseshoe crab (Tachypleus tridentatus). J. Biol. Chem., 265, 15365–15367.
26) Shigenaga, T., Muta, T., Toh, Y., Tokunaga, F., & Iwanaga, S. (1990) Antimicrobial tachyplesin peptide precursor. J. Biol. Chem., 265, 21350–21354.
27) Saito, T., Kawabata, S., Shigenaga, T., Cho, J., Nakajima, H., Hirata, M., & Iwanaga, S. (1995) A novel big defensin identified in horseshoe crab hemocytes: Isolation, amino acid sequence and antibacterial activity. J. Biochem., 117, 1131–1137.
28) Kawabata, S., Nagayama, R., Hirata, M., Shigenaga, T., Agarwala, K.L., Saito, T., Cho, J., Nakajima, H., & Iwanaga, S. (1996) Tachycitin, a small granular component in horseshoe crab hemocytes, is an antimicrobial protein with chitin-binding activity. J. Biochem., 120, 1253–1260.
29) Kawabata, S., Tokunaga, F., Kugi, Y., Motoyama, S., Miura, Y., Hirata, M., & Iwanaga, S. (1996) Limulus factor D, a 43-kDa protein isolated from horseshoe crab hemocytes, is a serine protease homologue with antimicrobial activity. FEBS Lett., 398, 146–150.
30) Kawabata, S., Saito, T., Saeki, K., Okino, N., Mizutani, A., Toh, Y., & Iwanaga, S. (1997) cDNA cloning, tissue distribution, and subcellular localization of horseshoe crab big defensin. Biol. Chem., 378, 289–292.
31) Osaki, T., Omotezako, M., Nagayama, R., Hirata, M., Iwanaga, S., Kasahara, J., Hattori, J., Ito, I., Sugiyama, H., & Kawabata, S. (1999) Horseshoe crab hemocyte-derived antimicrobial polypeptides, tachystatins, with sequence similarity to spider neurotoxins. J. Biol. Chem., 274, 26172–26178.
32) Suetake, T., Tsuda, S., Kawabata, S., Miura, K., Iwanaga, S., Hikichi, K., Nitta, K., & Kawano, K. (2000) Chitin-binding proteins in invertebrates and plants comprise a common chitin-binding structural motif. J. Biol. Chem., 275, 17929–17932.
33) Fujitani, N., Kawabata, S., Osaki, T., Kumaki, Y., Demura, M., Nitta, K., & Kawano, K. (2002) Structure of the antimicrobial peptide tachystatin A. J. Biol. Chem., 277, 23651–23657.
34) Suetake, T., Aizawa, T., Koganesawa, N., Osaki, T., Kobashigawa, Y., Demura, T., Kawabata, S., Kawano, K., Tsuda, S., & Nitta, K. (2002) Production and characterization of recombinant tachycitin, the chitin-binding protein. Protein Eng., 15, 763–769.
35) Kouno, T., Fujitani, N., Mizuguchi, M., Osaki, T., Nishimura, S., Kawabata, S., Aizawa, T., Demura, M., Nitta, K., & Kawano, K. (2008) A novel beta-defensin structure: A potential strategy of big defensin to overcome resistance by Gram-positive bacteria. Biochemistry, 47, 10611–10619.
36) Tokunaga, T., Yamada, M., Miyata, T., Ding, Y.-L., Hiranaga-Kawabata, M., Muta, T., Iwanaga, S., Ichinose, A., & Davie, E.W. (1993) Limulus hemocyte transglutaminase: Its purification and characterization, and identification of the intracellular substrates. J. Biol. Chem., 268, 252–261.
37) Tokunaga, T., Muta, T., Iwanaga, S., Ichinose, A., Davie, E.W., Kuma, K., & Miyata, T. (1993) Limulus hemocyte transglutaminase: cDNA cloning, amino acid sequence, and tissue localization. J. Biol. Chem., 268, 262–268.
38) Osaki, T., Okino, N., Tokunaga, F., Iwanaga, S., & Kawabata, S. (2002) Proline-rich cell surface antigens of horseshoe crab hemocytes are substrates for protein cross-linking with a clotting protein coagulin. J. Biol. Chem., 277, 40084–40090.
39) Iijima, M., Hashimoto, T., Matsuda, Y., Nagai, T., Yamano, Y., Ichi, T., Osaki, T., & Kawabata, S. (2005) Comprehensive sequence analysis of horseshoe crab cuticular proteins and their involvement in transglutaminase-dependent cross-linking. FEBS J., 272, 4774–4786.
40) Matsuda, Y., Osaki, T., Hashii, T., Koshiba, T., & Kawabata, S. (2007) A cysteine-rich protein from an arthropod stabilizes clotting mesh and immobilizes bacteria at injured sites. J. Biol. Chem., 282, 33545–33552.
41) Matsuda, Y., Koshiba, T., Osaki, T., Suyama, H., Arisaka, F., Toh, Y., & Kawabata, S. (2007) An arthropod cuticular chitin-binding protein endows injured sites with transglutaminase-dependent mesh. J. Biol. Chem., 282, 37316–37324.
42) Zhu, Y., Thangamani, S., Ho, B., & Ding, J.L. (2005) The ancient origin of the complement system. EMBO J., 24, 382–394.
43) Ariki, S., Takahara, S., Shibata, T., Fukuoka, T., Ozaki, A., Endo, Y., Fujita, T., Koshiba, T., & Kawabata, S. (2008) Factor C acts as a lipopolysaccharide-responsive C3 convertase in horseshoe crab complement activation. J. Immunol., 181, 7794–8001.
44) Tagawa, K., Yoshihara, Y., Shibata, T., Kitazaki, K., Endo, Y., Fujita, T., Koshiba, T., & Kawabata, S. (2012) Microbe-specific C3b deposition in the horseshoe crab complement system in a C2/factor B-dependent or -independent manner. PLoS One, 7, e36783.
45) Shibata, T., Kobayashi, Y., Ikeda, Y., & Kawabata, S. (2018) Intermolecular autocatalytic activation of serine protease zymogen factor C through an active transition state responding to lipopolysaccharide. J. Biol. Chem., 293, 11589–11599.
46) Kawasaki, H., Nose, T., Muta, T., Iwanaga, S., Shimoshigashi, Y., & Kawabata, S. (2000) Head-to-tail polymerization of coagulogen, a clottable protein of the horseshoe crab. J. Biol. Chem., 275, 35297–35301.
47) Kakinuma, A., Asano, T., Torii, H., & Sugino, Y. (1981) Gelation of Limulus amoebocyte lysate by an antitumor 1,3-β-D-glucan. Biochem. Biophys. Res. Commun., 101, 434–439.
48) Morita, T., Tanaka, S., Nakamura, T., & Iwanaga, S. (1981) A new1,3-β-D-glucan-mediated coagulation pathway found in Limulus amebocytes. FEBS Lett., 129, 318–321.
49) Seki, N., Muta, T., Oda, T., Iwaki, D., Kuma, K., Miyata, T., & Iwanaga, S. (1994) Horseshoe crab 1,3-β-D-glucan-sensitive coagulation factor G: A serine protease zymogen heterodimer with similarities to β-glucan-binding proteins. J. Biol. Chem., 269, 1370–1374.
50) Muta, T., Seki, N., Takaki, Y., Hashimoto, R., Oda, T., Iwanaga, A., Tokunaga, F., & Iwanaga, S. (1995) Purified horseshoe crab factor G: Reconstitution and characterization of the 1,3-β-D-glucan-sensitive serine protease cascade. J. Biol. Chem., 270, 892–897.
51) Takaki, Y., Seki, N., Kawabata, S., Iwanaga, S., & Muta, T. (2002) Duplicated Binding Sites for 1,3-β-D-glucan in the horseshoe crab coagulation factor G: Implications for a molecular basis of the pattern recognition in innate immunity. J. Biol. Chem., 277, 14281–14287.
52) Ueda, Y., Ohwada, S., Abe, Y., Shibata, T., Iijima, M., Yoshimitsu, Y., Koshiba, T., Nakata, M., Ueda, T., & Kawabata, S. (2009) Factor G utilizes a carbohydrate-binding cleft that is conserved between horseshoe crab and bacteria for the recognition of β-1,3-D-glucans. J. Immunol., 183, 3810–3818.
53) Kobayashi, Y., Takahashi, T., Shibata, T., Ikeda, S., Koshiba, T., Mizumura, H., Oda, T., & Kawabata, S. (2015) Factor B is the second lipopolysaccharide-binding protease zymogen in the horseshoe crab coagulation cascade. J. Biol. Chem., 290, 19379–19386.
54) Ariki, S., Koori, K., Osaki, T., Motoyama, K., Inamori, K., & Kawabata, S. (2004) A serine protease zymogen functions as a pattern-recognition receptor for lipopolysaccharides. Proc. Natl. Acad. Sci. USA, 101, 953–958.
55) Koshiba, T., Hashii, T., & Kawabata, S. (2007) A structural perspective on the interaction between lipopolysaccharide and Factor C, a receptor involved in recognition of Gram-negative bacteria. J. Biol. Chem., 282, 3962–3967.
56) Coughlin, S.R. (1999) How the protease thrombin talks to cells. Proc. Natl. Acad. Sci. USA, 96, 11023–11027.
57) Ozaki, A., Ariki, S., & Kawabata, S. (2005) An antimicrobial peptide tachyplesin acts as a secondary secretagogue and amplifies lipopolysaccharide-induced hemocyte exocytosis. FEBS J., 272, 3863–3871.
58) Nakamura, T., Tokunaga, F., Morita, T., Iwanaga, S., Kusumoto, S., Shiba, T., Kobayashi, T., & Inoue, K. (1988) Intercellular serine-protease zymogen, factor C, from horseshoe crab hemocytes: Its activation by synthetic lipid A analogues and acid phospholipids. Eur. J. Biochem., 176, 89–94.
59) Freer, S.T., Kraut, J., Robertus, J.D., Wright, H.T., & Xuong, N. (1970) Chymotrypsinogen: 2.5-Å crystal structure, comparison with α-chymotrypsin, and implications for zymogen activation. Biochemistry, 9, 1997–2009.
60) Wright, H.T. (1973) Activation of chymotrypsinogen-A: A hypothesis based upon comparison of the crystal structures of chymotrypsinogen-A and α-chymotrypsin. J. Mol. Biol., 79, 13–23.
61) Kawabata, S., Morita, T., Iwanaga, S., & Igarashi, H. (1985) Staphylocoagulase-binding region in human prothrombin. J. Biochem., 97, 325–331.
62) Kawabata, S., Morita, T., Iwanaga, S., & Igarashi, H. (1985) Difference in enzymatic properties between α-thrombin and staphylocoagulase complex and free α-thrombin. J. Biochem., 97, 1073–1078.
63) Kawabata, S., Morita, T., Iwanaga, S., & Igarashi, H. (1985) Enzymatic properties of staphylothrombin, an active molecular complex formed between staphylocoagulase and prothrombin. J. Biochem., 98, 1603–1614.
64) Kawabata, S., Miyata, T., Morita, T., Miyata, T., Iwanaga, S., & Igarashi, H. (1986) The amino acid sequence of the procoagulant- and prothrombin-binding domain isolated from staphylocoagulase. J. Biol. Chem., 261, 527–531.
65) Kawabata, S., Morita, T., Miyata, T., Iwanaga, S., & Igarashi, H. (1986) Isolation and characterization of staphylocoagulase chymotryptic fragment: Localization of the procoagulant- and prothrombin-binding domain of this protein. J. Biol. Chem., 261, 1427–1433.
66) Kaida, S., Miyata, T., Yoshizawa, Y., Kawabata, S., Morita, T., Igarashi, H., & Iwanaga, S. (1987) Nucleotide sequence of the staphylocoagulase gene: Its unique COOH-terminal 8 tandem repeats. J. Biochem., 102, 1177–1186.
67) Friedrich, R., Panizzi, P., Fuentes-Prior, P., Richer, K., Verhmme, I., Anderson, P.J., Kawabata, S., Huber, R., Bode, W., & Bock, P.E. (2003) Staphylocoagulase is a prototype for the mechanism of cofactor-induced zymogen activation. Nature, 425, 535–539.
68) Muta, T., Miyata, T., Misumi, Y., Tokunaga, F., Nakamura, T., Toh, Y., Ikehara, Y., & Iwanaga, S. (1991) Limulus factor C: An endotoxin-sensitive serine protease zymogen with a mosaic structure of complement-like, epidermal growth factor-like and lectin-like domains. J. Biol. Chem., 266, 6554–6561.
69) Wang, J., Tan, N.S., Ho, B., & Ding, J.L. (2002) Modular arrangement and secretion of a multidomain serine protease: Evidence for involvement of proline-rich region and N-glycans in the secretion pathway. J. Biol. Chem., 277, 36363–36372.
70) Kobayashi, Y., Shiga, T., Shibata, T., Sako, M., Maenaka, K., Koshiba, T., Mizumura, H., Oda, T., & Kawabata, S. (2014) The N-terminal Arg residue is essential for autocatalytic activation of a lipopolysaccharide-responsive protease zymogen. J. Biol. Chem., 289, 25987–25995.
71) Muta, T., Oda, T., & Iwanaga, S. (1993) Horseshoe crab coagulation factor B: A unique serine protease zymogen activated by cleavage of an Ile-Ile bond. J. Biol. Chem., 268, 21384–21388.
72) Muta, T., Hashimoto, R., Miyata, T., Nishimura, H., Toh, Y., & Iwanaga, S. (1990) Proclotting enzyme from horseshoe crab hemocytes: cDNA cloning, disulfide locations, and subcellular localization. J. Biol. Chem., 265, 22426–22433.
73) Jiang, H. & Kanost, M.R. (2000) The clip-domain family of serine proteinases in arthropods. Insect Biochem. Mol. Biol., 30, 95–105.
74) Yamashita, K., Shibata, T., Takahashi, T., Kobayashi, Y., & Kawabata, S. (2020) Roles of the clip domains of two protease zymogens in the coagulation cascade in horseshoe crabs. J. Biol. Chem., 295, 8857–8866.
75) Hartley, B.S. (1970) Homologies in serine proteinase. Philos. Trans. R. Soc. Lond., B, 257, 77–87.
76) Mihalyi, E. (1978) Proteolytic enzymes in Application of Proteolytic Enzymes to Protein Structure Studies, 2nd Edition, pp. 43–127, CRC Press, Inc., West Palm Beach, Florida.
77) Yamashita, K., Takeshita, N., Arita, A., Shibata, T., Kobayashi, Y., & Kawabata, S. (2021) A mutant equipped with a regenerated disulfide for the missing His loop of a serine protease zymogen in the horseshoe crab coagulation cascade. J. Biochem., 170, 489–500.
78) Miura, Y., Kawabata, S., & Iwanaga, S. (1994) A limulus intracellular coagulation inhibitor with characteristics of the serpin superfamily, purification, characterization, and cDNA cloning. J. Biol. Chem., 269, 542–547.
79) Miura, Y., Kawabata, S., Wakamiya, Y., Nakamura, T., & Iwanaga, S. (1995) A limulus intracellular coagulation inhibitor type 2: Purification, characterization, cDNA cloning, and tissue localization. J. Biol. Chem., 270, 558–565.
80) Lal Agarwala, K., Kawabata, S., Miura, Y., Kuroki, Y., & Iwanaga, S. (1996) Limulus intracellular coagulation inhibitor type 3: Purification, characterization, cDNA cloning, and tissue localization. J. Biol. Chem., 271, 23768–23774.
81) Srimal, S., Miyata, T., Kawabata, S., Miyata, T., & Iwanaga, S. (1985) The complete amino acid sequence of coagulogen isolated from Southeast Asian horseshoe crab, Carcinoscorpius rotundicauda. J. Biochem., 98, 305–318.
82) Leytus, S.P., Kurachi, K., Sakariassen, K.S., & Davie, E.W. (1986) Nucleotide sequence of the cDNA coding for human complement C1r. Biochemistry, 25, 4855–4863.
83) Journet, A. & Tosi, M. (1986) Cloning and sequencing of full-length cDNA encoding the precursor of human complement C1r. Biochem. J., 240, 783–787.
84) Mackinnon, C.M., Carter, P.E., Smyth, S.J., Dunbar, B., & Fothergill, J.E. (1987) Molecular cloning of cDNA for human complement component C1s: The complete amino acid sequence. Eur. J. Biochem., 169, 547–553.
85) Endo, Y., Takahashi, M., Nakao, M., Saiga, H., Sekine, H., Matsushita, M., Nonaka, M., & Fujita, T. (1998) Two lineage of mannose-binding lectin-associated serine protease (MASP) in vertebrates. J. Immunol., 161, 4924–4930.
86) Bergner, A., Oganessyan, V., Muta, T., Iwanaga, S., Typke, D., Huber, R., & Bode, W. (1996) Crystal structure of coagulogen, the clottable protein from horseshoe crab: A structural homologue of nerve growth factor. EMBO J., 15, 6789–6797.
87) Bergner, A., Muta, T., Iwanaga, S., Beisel, H.-G., Delotto, R., & Bode, W. (1997) Horseshoe crab coagulogen is an invertebrate protein with a nerve growth factor-like domain. Biol. Chem., 378, 283–287.
88) Lemaitre, B. & Hoffmann, J. (2007) The host defense of Drosophila melanogaster. Annu. Rev. Immunol., 25, 697–743.
89) Inamori, K., Ariki, S., & Kawabata, S. (2004) A Toll-like receptor in horseshoe crabs. Immunol. Rev., 198, 106–115.
90) Wang, X.W., Tan, N.S., Ho, B., & Ding, J.L. (2006) Evidence for the ancient origin of the NF-κB/IκB cascade: Its archaic role in pathogen infection and immunity. Proc. Natl. Acad. Sci. USA, 103, 4204–4420.
91) Urbanová, V., Hartmann, D., Grunclová, L., Šíma, R., Flemming, T., Hajdušek, O., & Kopáček, P. (2014) IrFC, an Ixodes ricinus injury-responsive molecular related to Limulus Factor C. Dev. Com. Immunol, 46, 439–447.
92) Zhou, Y., Liang, Y., Yan, Q., Zhang, L., Chen, D., Ruan, L., Kong, Y., Shi, H., Chen, M., & Chen, J. (2020) The draft genome of horseshoe crab Tachypleus tridentatus reveals its evolutionary scenario and well-developed innate immunity. BMC Genomics, 21, 137.
93) Mizumura, H., Ogura, N., Aketagawa, J., Aizawa, M., Kobayashi, Y., Kawabata, S., & Oda, T. (2017) Genetic engineering approach to develop next-generation reagents for endotoxin quantification. Innate Immun., 23, 136–146.
94) Shibata, T., Ariki, S., Shinzawa, N., Miyaji, R., Suyama, H., Sako, M., Inomata, N., Koshiba, T., Kanuka, H., & Kawabata, S. (2010) Protein crosslinking by transglutaminase controls cuticle morphogenesis in Drosophila. PLoS One, 5, e13477.
95) Shibata, T., Sekihara, S., Fujikawa, T., Miyaji, R., Maki, K., Ishihara, T., Koshiba, T., & Kawabata, S. (2013) Transglutaminase-catalyzed protein-protein cross-linking suppresses the activity of the NF-κB-like transcription factor Relish. Sci. Signal., 6, ra61.
96) Shibata, T., Maki, K., Hadano, J., Fujikawa, T., Kitazaki, K., Koshiba, T., & Kawabata, S. (2015) Crosslinking of a peritrophic matrix protein protects gut epithelia from bacterial exotoxins. PLoS Pathog., 11, e1005244.
97) Sekihara, S., Shibata, T., Hyakkendani, M., & Kawabata, S. (2016) RNA interference directed against the transglutaminase gene triggers dysbiosis of gut microbiota in Drosophila. J. Biol. Chem., 291, 25077–25087.
98) Maki, K., Shibata, T., & Kawabata, S. (2017) Transglutaminase-catalyzed incorporation of polyamines masks the DNA-binding region of the transcription factor relish. J. Biol. Chem., 292, 6369–6380.
99) Shibata, T., Hadano, J., Kawasaki, D., Dong, X., & Kawabata, S. (2017) Drosophila TG-A transglutaminase is secreted via an unconventional Golgi-independent mechanism involving exosomes and two types of fatty acylations. J. Biol. Chem., 292, 10723–10734.