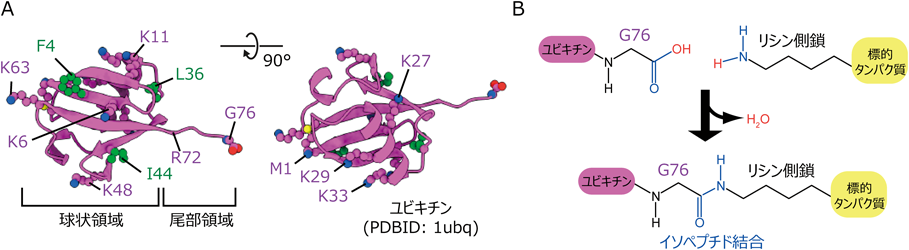
ユビキチン修飾は真核生物では必須の翻訳後修飾系であり,タンパク質の分解,DNA修復,免疫・炎症応答,細胞内輸送,細胞周期の制御,などきわめて多岐にわたる細胞機能を制御する.ユビキチン活性化酵素(E1),ユビキチン連結酵素(E2),ユビキチンリガーゼ(E3)が順番に働くことで,ユビキチンは基質と共有結合を形成し,その機能や運命をさまざまに制御する.一方,ユビキチン修飾は可逆的であり,脱ユビキチン化酵素(deubiquitinase:DUB)により除去される.
ユビキチン修飾では,主に標的タンパク質のリシン側鎖のアミノ基に対し,ユビキチンのC末端である76番目のグリシン(G76)がイソペプチド結合を介して結合する(図1)1).標的と結合したユビキチンは単独での修飾因子(モノユビキチン)としても機能するが,多くの場合ユビキチン自身に七つ存在するリシンやN末端メチオニンのアミノ基を介して数珠つなぎに連結し,8種類の結合型(K6, K11, K27, K29, K33, K48, K63, M1)のユビキチン鎖(ポリユビキチン)を形成する.これら,モノユビキチン修飾と8種類のユビキチン鎖修飾は,それぞれ異なる構造と機能を有すると考えられている.たとえば細胞内で最も豊富に存在するK48を介して連結したユビキチン鎖(K48鎖)は,標的タンパク質のプロテアソーム依存性分解シグナルとして機能する.一方,K63を介して結合したユビキチン鎖(K63鎖)は免疫・炎症応答,DNA修復,膜輸送といったさまざまな役割を持つ.このように,ユビキチン鎖の結合型に由来する高次構造はそれぞれが異なる機能情報を有しているため,ユビキチンコードと称されている2).
細胞機能を正常に維持するためにはユビキチンコードを制御することが必要である.ユビキチンコードの制御因子として,ユビキチン鎖を連結してコードを形成するライター,ユビキチン鎖と結合してコードを解読するデコーダー,ユビキチン鎖を除去してコードを消去するイレーサーが存在する.ライターを構成する酵素であるE1, E2, E3は,それぞれ2種類,約40種類,600種類以上,とユビキチン連結反応の後半になるほど種類が豊富になり,E2とE3によって基質の種類やユビキチン鎖の結合型を決定する.デコーダーは多様なファミリーから構成された,ユビキチン結合ドメイン(ubiquitin binding domain:UBD)を有している.特定の結合型のユビキチン鎖のみと結合するUBDを有するデコーダーが細胞内で対応するユビキチン鎖と結合することで,デコーダーごとに異なるシグナルを下流に伝達する.イレーサーはDUBにより担われており,約100種類存在していて,それぞれ異なる標的からユビキチン鎖を除去し,ユビキチンコードによるシグナル伝達を負に制御する.ライター,デコーダー,イレーサーはいずれも,ユビキチン鎖の結合型に対して高い特異性を持つものから,ほとんど特異性を持たないものまで,多様な種類が存在する.
近年,質量分析などの解析技術の発達に伴い,複数種類の結合型を有する混合鎖や,枝分かれした分岐鎖,などが実際に細胞内に存在し,機能していることが発見された他,ユビキチンがリン酸化やアセチル化といった翻訳後修飾を受けることも明らかとなり,ユビキチンコードの複雑性は当初考えられていたものよりも,はるかに複雑であることがわかってきた.本稿では,特定の結合型を有するユビキチン鎖が,どのようにしてUBDやイレーサーによって認識,切断されるのか,構造生物学の手法を用いて明らかにされたその特異的認識機構を著者の研究成果を交えて紹介する.
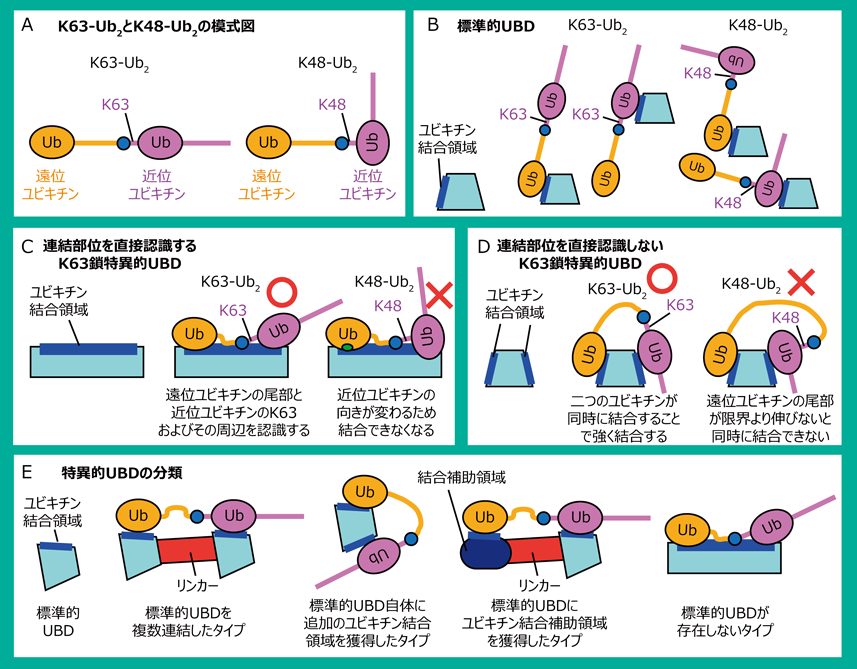
UBDにはユビキチン鎖の結合型を識別しないものと,識別するものが存在する(本稿ではそれぞれ標準的UBD,特異的UBDと呼ぶ).標準的UBDは,ユビキチンと相互作用するための領域を1か所だけ持ち,ユビキチン鎖の種類にかかわらず一つのユビキチンと相互作用するため,特異性がない(図2A, B).一方,著者が特異的UBDの構造解析を始めた当初は,特異的UBDはユビキチン鎖連結部位,すなわち,遠位ユビキチンの尾部領域から近位ユビキチンの特定のリシン(またはN末端メチオニン),およびその周辺構造を認識することで,特定の結合型を直接認識すると予測していた(図2C).しかし,特異的UBDとユビキチン鎖との複合体の立体構造が次々と解明されるにつれ,著者の予想は外れ,多くの特異的UBDは複数のユビキチンを同時に認識することで特異性を獲得していることがわかった.このとき,複数のユビキチン結合領域に対し,ある特定の結合型のユビキチン鎖であれば遠位と近位のユビキチンが同時に結合できるが,それ以外のユビキチン鎖の場合,遠位ユビキチンの尾部が限界まで伸びた構造をとっても近位ユビキチンのリシン(またはN末端)に届かないため同時に結合できない,という位置関係になることが重要である(図2D).ユビキチン結合領域がこのような位置関係である場合,特定のユビキチン鎖のみ複数か所の結合をすることができ,親和性が向上する.本節では,特異的UBDによる鎖特異的な結合機構を,実際の例をあげファミリーごとに紹介する(図2E,図3).なお,Ub2ではC末端が連結に使われていない側から近位,遠位ユビキチン,Ub3ではC末端が連結に使われていない側から,近位,中位,遠位ユビキチンと表記する.
1)UIM
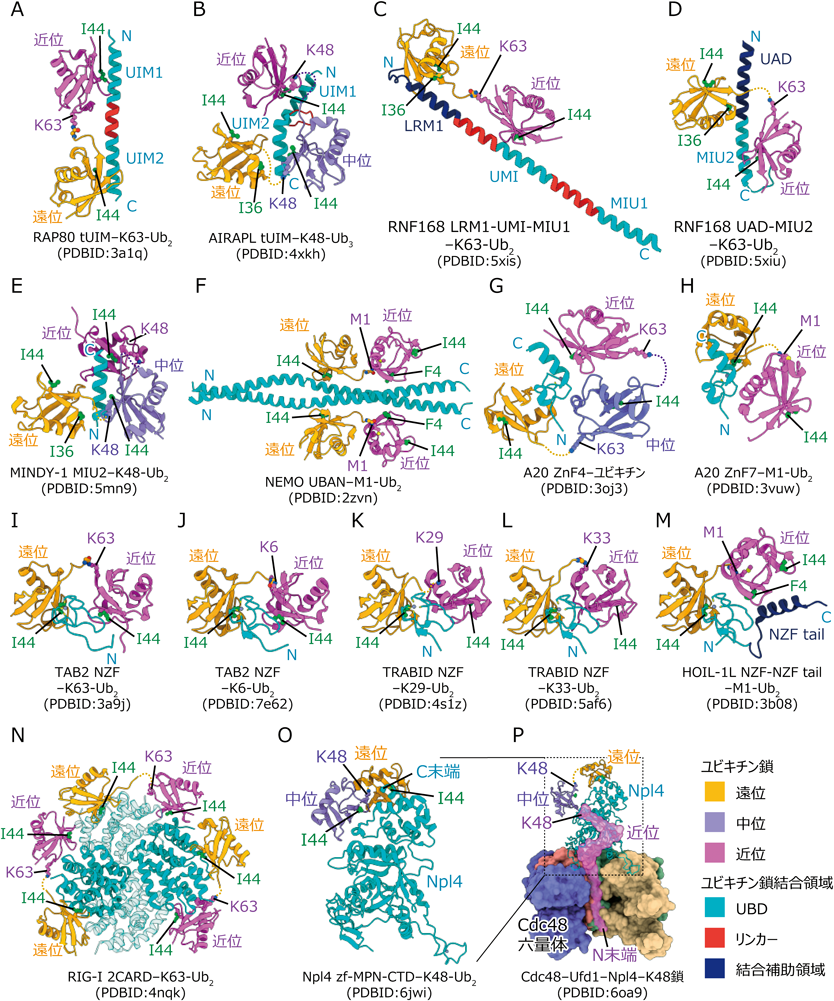
ubiquitin interacting motif(UIM)は1本のヘリックスのみからなるUBDでユビキチンのI44疎水性パッチと結合する.RAP80はUIMが二つ連結したタンデムUIM(tUIM)を持つことでK63鎖特異的に結合する.我々が決定した結晶構造から,RAP80の1番目のUIM(UIM1)と2番目のUIM(UIM2)がそれぞれユビキチンと結合した際,近位ユビキチンのK63のみが遠位ユビキチンのC末端と連結できる位置にあり,その他のアミノ基はK63に最も近いM1ですら結合できない距離に配置されることがわかった(図3A)4).つまり,K63鎖のみ,隣り合った二つのユビキチンがRAP80 tUIMと同時に結合することで,高い親和性を得ることができる.
tUIMを持つデコーダー分子としてはAIRAPLとK48-Ub3の複合体構造も報告されているが,RAP80とは鎖特異性獲得の機構は異なる5).AIRAPLの二つのUIMのうちUIM2は特殊で,標準的なUIMが持つユビキチン結合領域の他にもう一つユビキチン結合領域を持ち,ここで遠位ユビキチンのI36疎水性パッチと結合する(図3B).AIRAPL tUIMとK48-Ub3との複合体の構造をみると,UIM2と相互作用している遠位と中位のユビキチンはK48で連結されていないと同時に結合できない位置関係にあり,UIM2がAIRAPLのK48鎖特異性を決定していることがわかる.一方,AIRAPLのUIM1は標準的なUIMであり,ユビキチン結合領域を一つだけ持ち,K48-Ub3の近位ユビキチンと相互作用する.AIRAPLはUIM2のみでK48鎖特異性を獲得し,UIM1はユビキチンが三つ以上連結された長いユビキチン鎖への親和性を増強するために存在するのだろう.
2)MIU, UMI
motif interacting with ubiquitin(MIU)とUIM and MIU related UBD(UMI)もUIM同様1本のヘリックスのみからなるUBDでユビキチンのI44疎水性パッチと相互作用する.しかし,ユビキチンと結合した際のヘリックスの方向が逆で,UIMはヘリックスのN末端側がユビキチンのC末端側を向くが,MIUとUMIはヘリックスのC末端側がユビキチンのC末端側を向く.
RNF168は損傷DNAにユビキチンを付加するライターだが,DNA損傷箇所のK63鎖と特異的に結合し集積するというデコーダーとしての面も持つ.RNF168はN末端側からUMI, 1番目のMIU(MIU1),2番目のMIU(MIU2)の三つのUBDを持つが,UMIとMIU1の距離が15残基程度と近いため,UMIとMIU1がRAP80 tUIMのようにK63鎖と結合すると考えられていた.しかし,我々のグループで構造解析した結果,UMIとMIU1の距離はK63鎖特異的に結合するには遠すぎるため,RNF168の鎖特異性には関与しないことがわかった.一方,当初ユビキチンとの結合に関与しないと考えられていたLR motif 1(LRM1)が遠位ユビキチンと,UMIが近位ユビキチンと相互作用することでK63鎖特異的な結合をすることが明らかとなった(図3C)6).しかし,RNF168のホモログであるRNF169もLRM1-UMIを持つが,K63鎖への結合能を持たない.これは,LRM1とUMIの距離がRNF169ではRNF168と比べて5残基短いため,K63-Ub2の二つのユビキチンが同時に結合するための適切な距離を保てないためだと考えられる.
我々の詳細な解析によってRNF168はLRM1-UMIとは別に,MIU2もK63鎖特異的に結合することがわかった.ただし,MIU2だけでは特異性はほとんどなく,MIU2のN末端側に存在する結合補助領域ubiquitin-associated domain(UAD)が必要である.UADとMIU2が,それぞれ遠位,近位ユビキチンと同時に結合することで,UAD-MIU2はK63鎖特異的に結合する(図3D)6).
その他の鎖特異性のあるMIUとして,K48鎖特異的なイレーサーであるMINDY-1が二つ持つMIUのうち,MIU2がK48鎖特異的に結合することが知られている.MINDY-1 MIU2とK48-Ub2との複合体の結晶構造から,MINDY-1 MIU2の表面には追加のユビキチン結合領域が2か所存在することがわかった(図3E)7).つまり,MIUが本来持つ結合領域と合わせて3か所のユビキチン結合領域を持つため,補助領域なしでK48-Ub3の三つのユビキチンと同時に結合でき,K48鎖に特異性がある.なお,構造解析はK48-Ub2で行われたが,結晶学的対象操作により3か所すべてのユビキチンのモデルが置かれている.ところで,AIRAPL UIM2とMINDY-1 MIU2のK48鎖との複合体の構造をよく観察すると,遠位,中位ユビキチン,そしてUIM2とMIU2がほとんど同じ位置関係になる.図3B, EではAIRAPLとMINDY-1に関してこれらの分子が同一方向から観察できるよう描写したが,よく似ていることがわかる.UIMとMIUではヘリックスの方向が逆であるにもかかわらず,このような構造上の類似性が観察できるのは非常に面白い.
3)UBAN
ubiquitin binding in ABIN and NEMO(UBAN)モチーフは,ABIN-1, NEMO, OPTNなどに存在するM1鎖特異的なUBDである.UBANには他の特異的UBDとは異なる特徴をいくつか持つ.第一に,特異的UBDの多くは標準的UBDが同一のファミリーに存在するが,UBANは類似の標準的UBDが存在せず,いずれのUBANもM1鎖特異的に結合する.第二に,UBANはユビキチン鎖との結合の有無にかかわらずコイルドコイル構造を持つホモ二量体で,2分子のM1-Ub2と結合することができる(図3F)8).第三に,UBANはユビキチン鎖の連結部位を直接認識する.本稿で紹介する特異的UBDとユビキチン鎖複合体の結晶構造中では,UBANを除いたすべてで遠位ユビキチンの尾部領域の温度因子が他の領域と比べて高い,もしくはディスオーダーしており,UBDによってその位置が固定されていないことがわかる.つまり,いずれもが連結部位を認識していなかった(図2D,図3).一方,UBANとM1-Ub2の結晶構造中では遠位ユビキチンの尾部領域の温度因子は他の領域と比べて低くなっており,その構造が固定されていることがわかる.さらに,近位ユビキチンのM1は直接認識していないものの,M1周辺のQ2, E16, E64と相互作用することで,M1鎖の連結部分の構造を認識する.
4)A20 ZnF
A20 zinc finger(A20 ZnF)は四つのシステイン残基で亜鉛と結合するジンクフィンガーである.ライターとイレーサーの働きをするA20で見つかったためこのような名称がついているが,A20以外にもいくつかのユビキチン修飾系に関与するタンパク質から見つかっている.鎖特異性がないA20 ZnFは標準的UBDとしては珍しくユビキチンのD58を中心とする親水性パッチと結合する.A20はC末端側に七つのA20 ZnFを持つが,そのうちのN末端側から4番目のZnF4がK63鎖に,7番目のZnF7がM1鎖と特異的に結合する.A20 ZnF4はユビキチンとの複合体として結晶構造が報告されているが,結晶中でA20 ZnF4は3分子のユビキチンと結合しており,この3分子がK63鎖であっても問題ない位置関係にあるため,この構造はK63-Ub3との複合体を模したものだと考えられている(図3G)9).一方,A20 ZnF7はM1-Ub2との複合体の結晶構造が決定されている10).A20 ZnF7は遠位ユビキチンのI44疎水性パッチと,近位ユビキチンのD32を中心とする親水性のパッチと相互作用していた(図3H).A20 ZnF4, ZnF7ともに補助領域は持たず,A20 ZnFの表面にユビキチン結合領域を複数持つことで鎖特異性を獲得しているが,A20 ZnF7は奇妙な特徴がある.すでに説明したとおり,ほとんどの特異的UBDは標準的UBDに何らかの方法でユビキチン結合領域を追加することで特異性を獲得する(図2E).A20 ZnF4の場合は中位ユビキチンの位置が標準的なA20 ZnFのユビキチン結合位置に相当し,認識機構も標準的なA20 ZnFと等しい.しかし,A20 ZnF7のこの位置に結合するユビキチンは,M1-Ub2の近位ユビキチンであるが,A20 ZnF4の中位ユビキチンと方向が180°近く回転しており,まるで一致しない(図3G, H, A20 ZnF4とA20 ZnF7の向きは同一になるよう描写してある).どのような過程を経て,A20 ZnF7が標準的なA20 ZnFとは異なるユビキチン認識機構を獲得したのか,興味深い点である.
5)NZF
Npl4 zinc finger(NZF)はA20 ZnFと同様,四つのシステイン残基で亜鉛と結合するジンクフィンガーである.動物のNpl4のC末端で最初に見つかったためこの名前がついているが,やはり多くのユビキチン修飾系に関与するタンパク質にも存在する.その多くは鎖特異性を持たないが,TAB2, TRABID, HOIL-1LのNZFドメインは鎖特異性を有する11–15).特筆すべき点として,TAB2とTRABIDのNZFドメインは,それぞれK6鎖とK63鎖,K29鎖とK33鎖という2種類の結合型に特異的に結合するという性質を持つ.遠位ユビキチンの認識機構はどのNZFでもほぼ同様であるが,近位のユビキチンの認識機構はTAB2 NZF, TRABID NZF, HOIL-1L NZFで大きく異なる(図3I~M, NZFの向きはすべて同一になるように描写してある).しかし,2種類の結合型に対する特異性の機構はTAB2 NZFとTRABID NZFでよく似ているため,我々が構造決定したTAB2 NZFを例に説明する11, 15).TAB2 NZFは標準的なNZFと同様の様式で遠位ユビキチンと,もう一つのユビキチン結合領域で近位ユビキチンと結合する.この際,TAB2 NZFと近位ユビキチンの結合様式は,K6-Ub2とK63-Ub2で変わりはない.結合型が異なるのに,近位ユビキチンが同じようにTAB2 NZFと結合できるのは,遠位ユビキチンの球状領域のC末端(L71)から近位ユビキチンのK6とK63の側鎖アミノ基との距離が,それぞれ14 Åと16 Åであり,連結するのに適切だからである.一方,その次に距離が近い近位ユビキチンのK48の側鎖アミノ基でも,遠位ユビキチンのL71からの距離は23 Åと遠く離れており,連結することはできない.したがって,K6-Ub2とK63-Ub2のみが2か所同時にTAB2 NZFと結合することができ,TAB2 NZFはK6鎖とK63鎖に特異性を持つ(図3I, J).
HOIL-1LのNZFもNZFの表面にユビキチン結合領域を獲得することで近位ユビキチンと結合し,M1鎖特異的なUBDとして機能する(図3M)14).しかしHOIL-1Lはそれに加えて,C末端側にM1鎖との結合を増強するための結合補助領域を有している.我々がNZF tailと名づけたこの領域はヘリックス構造を持ち,近位ユビキチンと結合することでM1鎖に対する結合のみを補助し,HOIL-1L NZFのM1鎖特異性を増強する.
6)2CARD
RIG-Iのtandem caspase activation and recruitment domain(2CARD)は二つのCARDが並んだ構造を持つ,K63鎖特異的なUBDである.ウイルス由来の二本鎖RNAが細胞内に侵入すると,RIG-I 2CARDはK63鎖と結合し活性型の四量体となり,下流にシグナルを伝えることでインターフェロンの産生を誘導する.2CARDとK63-Ub2との複合体の結晶構造からは,1分子のK63-Ub2が2分子の2CARDにまたがるように結合することでRIG-I 2CARDの四量体化を誘導し,最終的に3分子のK63-Ub2が2CARD四量体を取り囲むことが解明された(図3N)16).多くの場合,K63鎖はデコーダー分子を集積させるための標識や足場として機能するが,直接的にタンパク質の多量体化を誘導することもあるという事実は大変興味深い.
7)Npl4
Npl4とUfd1はヘテロ二量体を形成し,Cdc48六量体がUfd1-Npl4ヘテロ二量体と結合する.K48鎖修飾を受けた基質は多くの場合そのままではプロテアソームで分解をすることができず,事前にCdc48-Ufd1-Npl4複合体がK48鎖修飾を受けた基質を認識し,Cdc48六量体の中心にある孔に通すことで解きほぐす必要がある.K48鎖特異的な結合には酵母の場合Npl4が必要であることがわかっていたため,我々はNpl4とK48-Ub2の複合体の結晶構造を決定した.その結果,Npl4のC末端ヘリックスがUMIと似た配列を持っており遠位ユビキチンと結合すること,そしてC末端ヘリックス近傍のループ領域が近位ユビキチン(図3Oでは中位ユビキチンと表記)を認識することで,K48鎖特異的な結合をすることがわかった17).
このように酵母Npl4のK48鎖特異的結合機構は解明できたが,ヒトNpl4はC末端ヘリックスのユビキチンとの結合に必要な残基が変異している.しかし,ヒトNpl4はC末端に鎖特異性のないNZFドメインを持つため,あらゆる種類のユビキチン鎖と結合することができる.ところが,Cdc48-Ufd1-Npl4複合体はヒト由来,酵母由来ともにK48鎖修飾を受けた基質しか解きほぐすことができない.また,酵母Npl4のC末端ヘリックスに変異を導入したうえでNZFドメインを付加し,酵母Npl4をあらゆる種類のユビキチン鎖と結合するように改変した場合でも,酵母Cdc48-Ufd1-Npl4複合体はK48鎖のみを標的とする17).この結果はNpl4のC末端以外にCdc48-Ufd1-Npl4複合体がK48鎖を特異的に認識する仕組みがあることを意味する.我々の結晶構造からはその位置は解明できなかったが,酵母Cdc48-Ufd1-Npl4がK48鎖修飾を受けた基質を解きほぐしている最中の構造が,Rapoportらによりクライオ電子顕微鏡を用いた単粒子解析により決定されたことから,その仕組みが解明された18).得られた構造ではUfd1のモデルは構築されていなかったが,Cdc48, Npl4に加えK48鎖の三つのユビキチンが観察できる(図3P).遠位,中位ユビキチンの位置は我々が構造決定した酵母Npl4-K48-Ub2複合体と似ていたが,驚くべきことに近位ユビキチンは解きほぐされた状態でNpl4に存在する溝に収まっており,そのN末端側からCdc48六量体の孔へと挿入されることがわかった.解きほぐされた近位ユビキチンの構造はNpl4の溝に認識されているため,中位ユビキチンのC末端とK48で連結していないと溝に収まることができない.このため,Cdc48-Npl4-Ufd1複合体はK48鎖のみを解きほぐすことができると考えられる.また,この溝を構成するアミノ酸はヒトと酵母でよく似ているため,同じ機構でK48鎖を認識するのだろう.
最後に,話は変わるがM1鎖が細胞内で機能していることが知られるまでは,本節でも紹介したM1鎖特異的なUBDであるNEMO UBANは,K63鎖特異的に結合すると考えられていた.しかし,その後の解析により,実際にはNEMO UBANのK63鎖への親和性は他のユビキチン鎖への親和性と大きな違いはないことがわかった.UBDの鎖特異性を測定する際は,プルダウンアッセイだけでなく,ユビキチン鎖の長さを統一した上で解離定数を測定するなど,細心の注意を払う必要があるだろう.
4. 特定の結合型のユビキチン鎖を切断するイレーサー
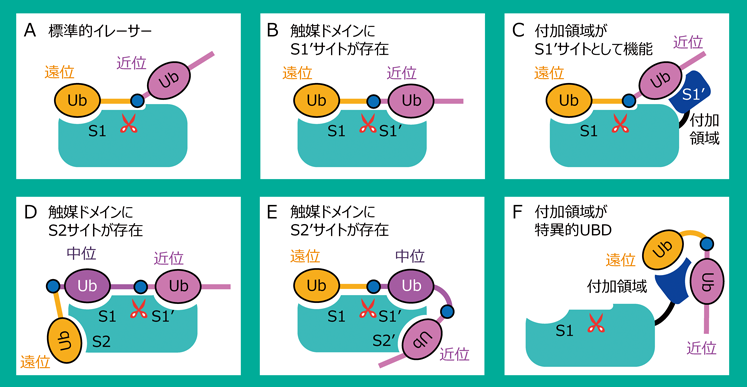
イレーサーもUBD同様,ユビキチン鎖の結合型を識別しないものと,識別するものが存在する(本稿ではそれぞれ標準的イレーサー,特異的イレーサーと呼ぶ).イレーサーによって切断を受けるユビキチン鎖の連結部分を中心として,遠位ユビキチンが結合する領域をS1,近位ユビキチンが結合する領域をS1′と呼ぶ(図4).また,S1に結合するユビキチンよりさらに遠位側のユビキチンが結合する領域をS2, S1′に結合するユビキチンよりさらに近位側のユビキチンが結合する領域をS2′,S3′……と呼ぶ.イレーサーはUBDとは異なり,ユビキチンのC末端を切断する必要があるため,イレーサーのS1はユビキチンの尾部領域と結合することで位置を固定し,C末端G76が活性中心の近傍に配置される.標準的イレーサーでは,S1と遠位ユビキチンとの結合のみで切断することができるため,結合型はどのようなものでも構わない(図4A).一方,特異的イレーサーではS1だけの結合では不十分で他の結合領域を必要とする,または特定の種類の結合型の場合のみ近位ユビキチンが結合することができる,などの方法で特異性を獲得している.ほとんどの場合,ある特定の結合型の場合のみ,近位ユビキチンがS1′と結合し,切断部位が活性中心の近傍に配置されるため,特異的に切断が起こる.真核生物のイレーサーは触媒ドメインの構造が異なる七つのファミリーから構成されるが,本節では鎖特異的な認識機構が解明された,五つのファミリーについて紹介する.
1)JAMM
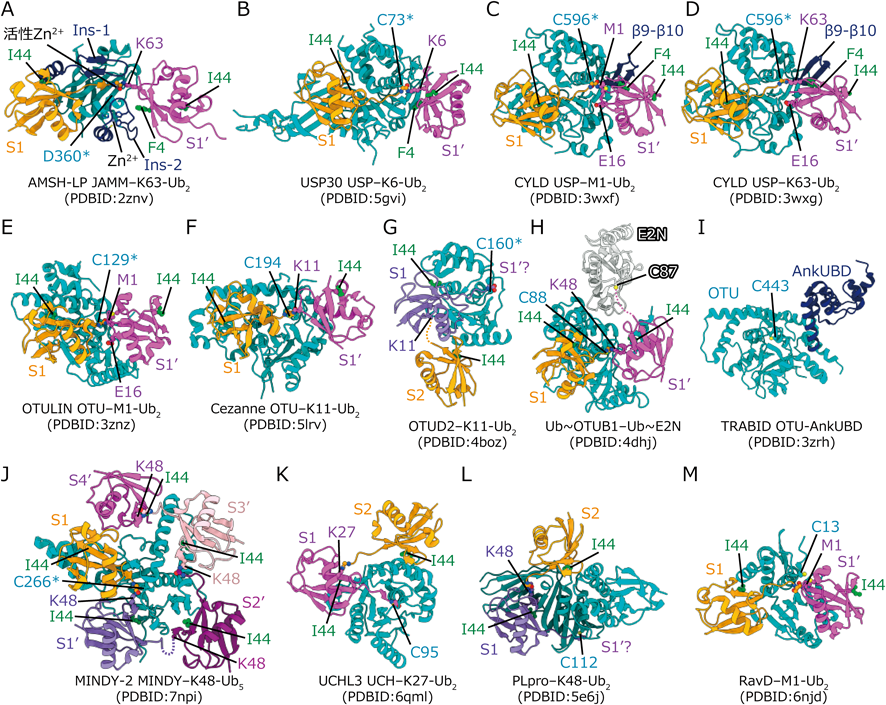
ほとんどのイレーサーは触媒三残基を持ったシステインプロテアーゼであるが,JAB1/MPN/Mov34 metalloenzyme(JAMM)ファミリーに属するイレーサーのみ,亜鉛プロテアーゼである.JAMMファミリーに属するAMSH, AMSH-LP, BRCC36はK63鎖に対する高い特異性を持つ.ここでは,著者が構造決定したAMSH-LPによるK63鎖切断機構を紹介する19).
JAMMモチーフはイレーサーに属さないいくつかのタンパク質でもみられるが,脱ユビキチン化活性を持つJAMMには二つの挿入領域,Insertion-1(Ins-1)とIns-2が存在する.Ins-1とIns-2の構造はイレーサーに属するJAMMでも構造が異なり,その基質特異性に影響を及ぼす.AMSH-LPの場合も,Ins-2がS1′として機能して近位ユビキチンのF4疎水性パッチと結合する(図4C,図5A).また,Ins-2だけでなく,JAMMモチーフ自体にもS1′が存在し,近位ユビキチンのK63の前後,Q62とE64と水素結合を形成していた(図4B).これらの相互作用により,近位ユビキチンがK63でつながっていた場合のみAMSH-LPは切断する.また,AMSH-LPのIns-2は亜鉛と結合することで構造を維持しているが,この亜鉛と結合できなくなるような変異を導入すると,K63鎖への切断活性は減弱するがK48鎖への切断活性はわずかに上昇する19).これは,Ins-2が,K63鎖以外の近位ユビキチンが活性中心に接近することを防ぐことを意味する.これら複数の機構により,AMSH-LPは高いK63鎖特異性を獲得している.
2)USP
ubiquitin-specific protease(USP)に属するイレーサーは約50種類存在し,イレーサーの中で最も種類が多い.現在までにUSP30とCYLDが鎖特異性を有することが発見されているが,それ以外のUSPファミリーについてはM1鎖に対して活性があるか否かの違いはあるものの,鎖特異性が報告されていない.ここでは,著者が構造決定したゼブラフィッシュ由来のUSP30とCYLDの構造を紹介する.
USP30はK6鎖特異性を持つが,その特異性は高くなく,K11, K48, K63鎖に対してもある程度活性がある.USP30とK6-Ub2との複合体の構造解析の結果,USP30はS1′によってK6-Ub2の近位ユビキチンのF4疎水性パッチを認識することで,K6鎖への結合を強めていることがわかった(図5B)20).一方K6鎖以外では,近位ユビキチンのF4疎水性パッチは同じ位置にこないため,強く結合することができないのだろう.しかし,USP30のS1は特異性のないUSPとよく似ており,遠位ユビキチンとの結合だけでもある程度切断はできる.そのため,USP30の特異性は完全ではないのだと考えられる.
CYLDはM1鎖とK63鎖特異的なUSPであるが,その特異性はUSP30より高く,他のユビキチン鎖はK11鎖とK48鎖をごくわずかに切断する程度である.CYLDはUSP30と比較してS1が小さく,遠位ユビキチンとの親和性が低いと予想される(図5C, D)21).このため,近位ユビキチンとの結合がないとUb2と強く結合することができず,USP30ほどにはゆるい特異性ではないのだろう.ではなぜCYLDは2種類のユビキチン鎖を認識することができるのか.これはCYLDのS1′が柔軟で,M1鎖でもK63鎖でも近位ユビキチンと結合することができるためである.図5C, DのCYLDとM1-Ub2およびK63-Ub2複合体の結晶構造では,CYLDの向きが同一となるよう描写してあるが,近位ユビキチンの向きがわずかに異なる.このような近位ユビキチンの向きの違いを許容するため,CYLDは他のUSPには存在しない逆平行βシートβ9–β10を付加領域として持つ.β9–β10はわずかに構造を変えてM1-Ub2やK63-Ub2の近位ユビキチンのF4疎水性パッチと結合することで,この二つのユビキチン鎖に対する結合を増強する.また,近位ユビキチンのE16はCYLD特有なループ構造と水素結合を形成するが,E16は近位ユビキチンが回転してもあまりずれない位置に配置されているため,近位ユビキチンがわずかに回転しても,CYLDと水素結合することができる.このように,CYLDのS1′は近位ユビキチンのわずかな回転であれば許容し,M1, K63鎖という2種類のユビキチン鎖を切断できる.
3)OTU
ovarian tumor domain(OTU)に属するイレーサーは約20種類存在し,Komanderらによって網羅的な鎖特異性が解析された結果,多くのOTUが鎖特異性を持つことがわかった22).ここでは,主にKomanderらによって決定されたOTUとUb2の複合体の結晶構造を紹介する.
OTULINはM1鎖に対して高い特異性を持つOTUで,S1′サイトで近位ユビキチンを認識することでM1鎖への特異性を獲得している(図5E)23).重要な点としてOTULINの触媒三残基は,M1鎖が結合していない状態では正しく並んでおらず,切断活性を持たない.しかし,M1鎖の近位ユビキチンのE16がCys/His/Asnで構成される触媒三残基のAsnと水素結合を形成すると,触媒三残基が正しく並び,M1鎖を切断することになる.
CezanneはK11鎖特異的なOTUで,やはりそのS1′サイトで近位ユビキチンと相互作用することで,K11鎖特異的に切断することがわかっている(図5F)24).Cezanneの触媒三残基もK11鎖が結合していない状態では正しく並んでおらず,K11鎖が結合することでS1′が大きな構造変化を受け,それに伴い触媒三残基が正しく配置され,K11鎖を切断する.ただし,CezanneはOTULINと比べると特異性は低く,高濃度ではM1, K29, K33鎖以外のユビキチン鎖を切断する.OTUD2もK11鎖特異的なOTUであるが,CezanneとはK11鎖認識機構が異なる.報告されたOTUD2 OTUドメインとK11-Ub2複合体の結晶構造から,OTUD2 OTUドメインにはS2が存在し,S1とS2の2か所で二つのユビキチンと結合することでK11鎖に対する特異性を獲得していることがわかった(図4D,図5G)22).S1とS2の結合だけでは活性中心にユビキチン間の結合が配置されないため切断できないが,活性測定の結果から,OTUD2は結晶構造からは解明されていないS1′が存在し,S2, S1, S1′の3か所同時にユビキチンが結合することでK11-Ub3の中位と近位のユビキチン間を切断すると考えられている.このように,イレーサーによってはUb2よりも長いユビキチン鎖に対してより強い特異性を持つものも存在するため,注意が必要である.
特殊な機能を持つOTUB1によるK48鎖切断機構も解明されている.OTUB1は脱ユビキチン化活性以外に,E1からユビキチンを受け渡されたE2(Ub~E2)と結合することで,ユビキチン鎖伸長を阻害するという機能を持つ.ユビキチンのC末端を活性中心に共有結合させたOTUB1(Ub~OTUB1)とUb~E2の複合体の構造からユビキチン鎖伸長反応の阻害機構が解明された(図5H)25).さらに,驚くべきことにこの構造中に存在する二つのユビキチンは,S1とS1′に結合していたため,OTUB1のK48鎖特異的切断機構も明らかとなった.
その他,K29, K33鎖特異的切断活性を持つTRABIDのOTUは,ユビキチン鎖との複合体の構造は決定されていないが,C末端にS1′として機能するankyrin-repeat UBD(AnkUBD)という付加領域が存在し,K29, K33鎖特異性を向上させることがわかっている(図4C,図5I)26).また,以前の節でも紹介したTRABID OTUのN末端側に存在するNZFドメインはK29, K33鎖特異的な結合能を有する(図3K, L, 図4F).NZFドメインはTRABIDのUb2に対する活性には影響を及ぼさないが,長いユビキチン鎖であった場合活性を増強する.長いユビキチン鎖がNZFとOTUを同時に結合することで,TRABIDは長いユビキチン鎖を効率よく切断すると考えられている26).
4)MINDY
MIU-containing novel DUB family(MINDY)に属するMINDY-1とMINDY-2はよく似た触媒ドメインを持ち,どちらも長いK48鎖の遠位側のユビキチンを切断する.最近報告されたMINDY-2とK48-Ub5の結晶構造から,MINDY-1とMINDY-2は5か所のユビキチン結合領域を持つことが明らかとなった(図5J)27).その結合領域はS1とS1′~4′となっており,K48-Ub5が切断される際は最も遠位のユビキチンが切断を受けるということが構造から示された(図4E).また,活性測定の結果から,K48-Ub4よりも短い場合はMINDY-1/2の活性は大きく減少することや,K48-Ub6より長い場合は近位側から5番目以降のユビキチン鎖を優先して切断することも解明された.また,以前の節で紹介したようにMINDY-1はC末端側にK48鎖特異的に結合するMIUを持つ(図3E).このMIUもMINDY-1の長いユビキチン鎖に対する活性を向上させるようだ(図4F).
5)UCH
ubiquitin C-terminal hydrolase(UCH)に属するUCHL3はK27鎖と特異的に結合するが,ユビキチン鎖は切断しない.報告されているK27-Ub2との複合体の結晶構造から,UCHL3のS1とS2に二つのユビキチンが結合することで,K27鎖特異性を獲得していることがわかった(図5K)28).しかし,おそらく立体障害のためにS1′にユビキチンが入り込むことはできないため,UCHL3はユビキチン鎖を切断しない.UCHL3が切断する基質ははっきりしていないが,K27鎖が結合することでUCHL3の活性を阻害していると考えられている.
紙面の都合で割愛するが,一部のウイルスや細菌は特異的イレーサーを持ち,SARSコロナウイルスのPLproとK48-Ub2との複合体や,レジオネラ・ニューモフィラのRavDとM1-Ub2との複合体については構造も報告されている(図5L, M)29, 30).ウイルスや細菌のイレーサーは,感染過程においてヒトの免疫系を破壊し,感染を促進する.つまり,その阻害剤は治療薬としての効果が期待できるため,今後のさらなる研究が期待される.