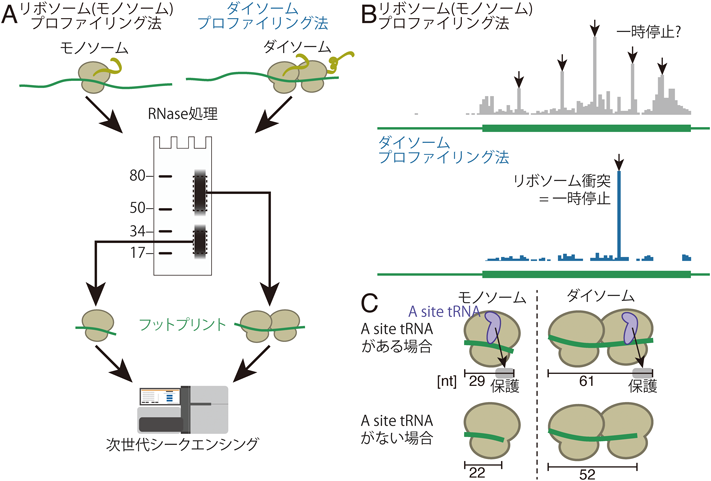
リボソームプロファイリング法はまさにこの問題を解決することのできる技術として登場した3).リボソームは非常に大きい複合体であり,mRNAの一部分を内部に取り込む形で結合する.そのために,RNase処理を施してもリボソーム内部に含まれるmRNA領域は分解から免れる.この残った約30塩基程度のRNA断片(リボソームフットプリントと呼ぶ)の配列を次世代シークエンサーによって網羅的に決定することがリボソームプロファイリング法の骨子である(図1A左).解析された核酸配列はmRNA上におけるリボソームの位置情報を反映する.リボソームプロファイリング法は細胞の中で,1)どのmRNAの,2)mRNA上のどの領域で,3)どのコドンが,4)どの程度,翻訳されていたかという情報を得ることのできる非常に強力な手法である(図1B).
mRNAのORF中の特定のコドン上でリボソームが一時停止していた場合,リボソームプロファイリングでは該当するコドンからより多くのフットプリントが出現することになり,ピークとして算出されるはずである(図1B).しかしながら,リボソームプロファイリング法では通常に翻訳伸長しているリボソームと一時停止しているリボソームを見分けることができないため,感度高く翻訳停止位置を決定することが難しかった.
それに対して近年開発されたダイソームプロファイリング法はこれまで不明瞭だったリボソーム停止位置を高感度,高分解能かつ網羅的に解析することができる4–14).ある特定のコドン上でリボソームが十分な時間一時停止すると,さらに次のリボソームが追いつき一時停止していたリボソームに衝突することになり,二つのリボソームが連なった複合体がmRNA上で形成されることになる(図1A右).この複合体をダイソーム(di-ribosome:disome)と呼ぶ.通常のリボソームプロファイリング法[ダイソームプロファイリング法と特に区別し,モノソーム(mono-ribosome:monosome)プロファイリング法と呼ぶ]では,リボソーム一つ分が残すフットプリントとして約20~30塩基長のRNA断片を回収し解析するのに対し,ダイソームプロファイリング法ではそのおおよそ倍の長さである約50~60塩基長のRNA断片を回収し実験に用いる(図1A).ダイソームプロファイリング法では一時停止したリボソーム由来のフットプリントがより濃縮されることになる(図1B).実際にこの手法がこれまでに,ヒト培養細胞8),マウス肝臓7),マウスES細胞10),ゼブラフィッシュ胚8),シロイヌナズナ12),酵母4, 6, 9, 13),大腸菌5, 14),SARS-CoV-2ウイルス11)に応用されている.
予想されるとおり,ダイソームフットプリントの長さはおおよそモノソームプリント長の2倍程度,約50~60塩基長程度になる(図1C).特に51~54塩基長,58~61塩基長の2種類の長さのフットプリントが得られる8, 9).これらは前方の一時停止位置しているリボソームの状態を反映することがわかっている.
通常のモノソームプロファイリングではリボソームのA site上のtRNAの有無に従って長いフットプリント(28~30塩基長,A siteにtRNAがある場合)と短いフットプリント(21~22塩基長,A siteにtRNAがない場合)が生じる(図1C)15).これはA siteのtRNAによって,mRNAがRNaseによって3′末端側から刈り込まれるのを防ぐためである.ダイソームの前方のリボソームがA siteにtRNAを保持しているかに従って2種類の長さのダイソームフットプリントが生じると考えられる(図1C).
リボソームの衝突が生じる過程として,二つのシナリオが考えられる.たとえば,mRNAの翻訳の効率が高く,より多くのリボソームが結合するため,確率論的に衝突するリボソームが多い,ということが考えられる(stochasticモデル).この場合,リボソーム衝突が生じるmRNA上の位置はランダムである.実際にマウスの肝臓などではこの傾向が高い7).
その一方で,特定のmRNA上でリボソームの一時停止が生じやすい配列がある場合,決まったコドン上に特にダイソームが形成されやすいことが考えられる(deterministicモデル).特にダイソームフットプリントが蓄積しやすいコドン位置には以下で列挙するような特徴があることがわかっている.注目すべきはこれらの特徴は必ずしもすべての生物種や組織に共通するわけではなく,それぞれの生物種や組織,細胞種によって異なる点である.これは翻訳伸長,翻訳終結,リボソームのリサイクルの速度がダイナミックに変化しうることを示している.
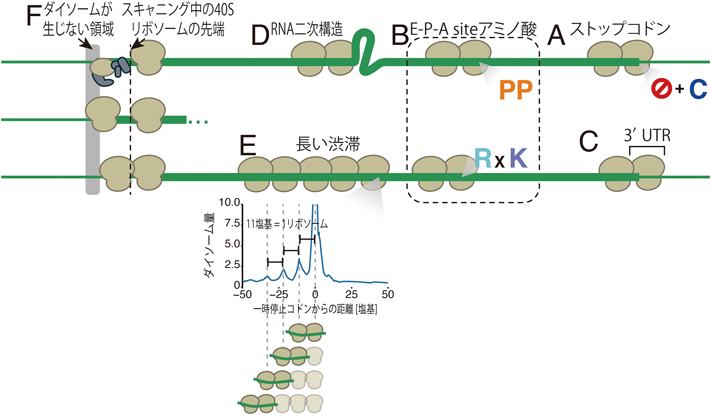
1)ストップコドン
ストップコドンでは非常に多くダイソームが形成されることが報告されている(図2A)8, 9, 13).このような傾向はヒト培養細胞,ゼブラフィッシュ胚,酵母でみられる.ストップコドンはeRF1と呼ばれる因子によって認識されるが,eRF1によるストップコドンの認識は,ストップコドンのさらに下流1塩基目の配列によっても影響されることがわかっており,ピリミジン塩基(U/C)よりもプリン塩基(A/G)が好まれることが知られている16).ヒト培養細胞ではストップコドン下流にC塩基が存在するとよりダイソームが形成されやすいことがわかっており,翻訳終結反応の効率が悪いストップコドンでリボソームの衝突が生じることが示唆されている8).それに対して,マウス肝臓ではストップコドン上でダイソームが特段蓄積する様子はみられていない7).
2)E, P, A siteのコドンおよびアミノ酸配列
ダイソームはストップコドンだけでなくORF中にも多数生じる.ダイソーム中の二つリボソームのうち,先端で一時停止しているリボソームが読み取っているコドンやアミノ酸,特に三つのtRNA結合サイト(E site, P site, A site),に強い傾向が現れる(図2B).たとえば,プロリンの連続配列やアルギニン-X-リシン配列はリボソームによるペプチジル転移反応が非効率な配列として知られており17, 18),実際にそのような配列でダイソームが形成される8, 9, 13).特にプロリンはペプチジル転移反応の供与体としても受容体としても,活性が低いことが知られている.また,アミノ酸ではなくコドンの配列による効果も観察されることから,tRNAの読み取り段階での制御も示唆されている7).
3)3′ UTR
通常リボソームはストップコドン上で翻訳を終結した後にmRNAから解離するが,ダイソームが3′ UTR上で形成される場合がある.特にORF中のリボソームがストップコドン上で停滞していたリボソームを3′ UTRに押し出すような形でダイソームフットプリントが取得される(図2C)8, 9).ダイソームの前方のリボソームはもともとストップコドン上で翻訳終結反応が完了し,tRNAと新生ペプチドが切断された後,80Sリボソーム自体がリサイクルされず依然としてmRNAに結合した状態の分子種であることが示唆されている.これはリボソームリサイクルの速度が,翻訳の伸長や終結よりも遅いことを示している.
4)二次構造
非常に強いRNAの二次構造がORF中に形成されると翻訳伸長反応の立体障害となることが知られている.酵母ではRNAの二次構造の上流にダイソームが観測される(図2D)13).その一方でヒト培養細胞ではそのような傾向は高くない8)が,これは特殊な因子(AVEN)によって二次構造が巻き戻されているためである可能性が示唆されている10).
また,特殊なRNAの二次構造によるダイソームの形成が報告されている.SARS-CoV-2ウイルスのORF1aとORF1bの間にはフレームシフト位置が存在し,下流のシュードノット(pseudoknot)構造によってリボソームを停滞させ,フレームシフトを促進することが知られている.ダイソームプロファイリングによってまさにこのフレームシフト直前にリボソームの衝突が生じることが明らかになっている11).
5)長いリボソーム渋滞
ダイソームプロファイリング法では衝突した二つのリボソームを観測しているが,そのデータ解析から実際の細胞ではさらに長い3個以上のリボソームの渋滞が生じることがわかる.ダイソームの上流にはさらにリボソーム一つ分(33塩基=11コドン)程度の間隔でダイソームが蓄積するのが観察される(図2E)8).特に,XBP1uと呼ばれる遺伝子上にあるリボソーム一時停止位置では最大六つのリボソームが連続して渋滞を起こしていることが観察される8).また,酵母では三つのリボソームの衝突(tri-ribosome:trisome)によるフットプリントも観測することができる9).
6)ダイソームが生じない領域
逆にリボソーム渋滞が生じにくい位置も存在する.たとえば,後方のリボソームは開始コドンから4コドンほどは先端のリボソームと衝突できない(図2F)4, 7–9).これは,スキャニング中の40Sリボソームに開始因子等が結合しており,開始コドン上に80Sを形成するための余分なスペース(4コドン程度)が必要だからではないかと考察されている.
7)モノソームプロファイリングとの比較
リボソームの衝突が生じるためには前方のリボソームの一時停止が必要であることから,ダイソームプロファイリングで観察される衝突位置にはモノソームが常に蓄積すると考えられる.しかしながらこの直感的な予想と相反して,ダイソームが生じていてもモノソームが蓄積しない位置というのが多く観察される7–9, 13).これは上記のタイプに加えて,リボソームの停滞が強固すぎ,多くのリボソームがダイソームになってしまった結果,モノソームプロファイリングではリードが得られない,という状況を反映していると考えられる.今後分子動力学等を利用した解析により,ダイソーム形成のダイナミクスに対する理解がさらに進むことが期待される.
以上のように多くのmRNA,多くのコドン位置でリボソームが一時停止し,ダイソームが形成されることが明らかになってきた.リボソームの一時停止に関しては1)タンパク質フォールディング促進と2)翻訳異常センサーとしての役割の2通りが報告されている.
1)タンパク質フォールディング促進
複数の生物種間(酵母–ヒト,ヒト–ゼブラフィッシュ)におけるホモログで,全く同じ遺伝子の同じコドン上でダイソームが形成されることが示されており,進化的な重要な機能の存在を示している8, 13).また,ダイソームプロファイリングから見つかるリボソーム一時停止位置はαヘリックスをコードするmRNA配列間に多く見つかることから7),リボソームが一時停止することで,合成中のアミノ酸鎖がフォールディングする時間的猶予を与えるのではないか,と推察されている.実際に,ダイソーム上にはフォールディングを助ける分子シャペロンがより多くリクルートされるということが報告されている13).
2)翻訳異常センサー
その一方でダイソームの形成が細胞にとって有害なmRNAやタンパク質の産生を示すシグナルとして利用されることもわかってきている.たとえば,ribosome-associated quality control(RQC),ribosome stress response(RSR)2),アミノ酸飢餓応答のようなストレス応答のシグナルがあげられる.このような経路はダイソームに特異的な構造・分子インターフェイスを認識する因子が重要な役割を果たす.たとえばRQCでは,Hel2と呼ばれる因子がダイソームを認識するが,この因子を酵母においてノックアウトすると,RQCの標的となることがわかっている配列(リシンやアルギニンの連続配列)上のダイソームが不安定化し減少する9).また,ヒトにおいてもダイソームを多く蓄積させるXBP1u mRNAから産生されるタンパク質がRQCのシステムによって分解されていることがわかってきた8).
以上のような,細胞にとって許容してもよいダイソームと翻訳異常時に形成されるダイソームがどのように見分けられているか,その分子機構の理解が今後期待される.