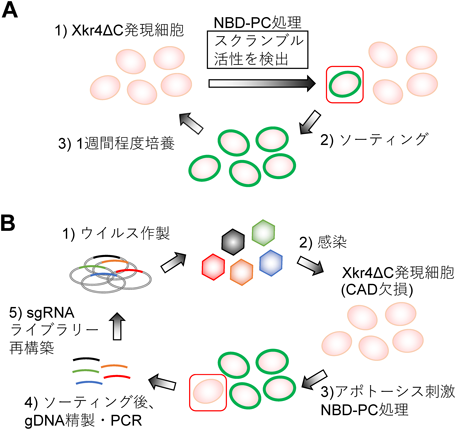
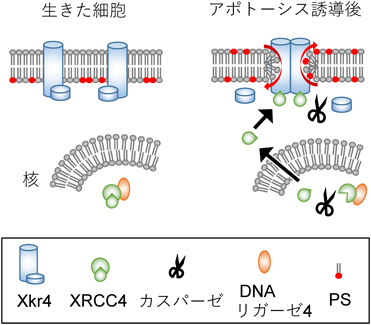
核内タンパク質によるスクランブラーゼの活性制御Regulatory mechanism of scramblase activity by nuclear protein
京都大学高等研究院物質-細胞統合システム拠点(iCeMS)Institute for Integrated Cell-Material Sciences (WPI-iCeMS), Kyoto University ◇ 〒606–8501 京都市左京区吉田本町 ◇ Yoshida-Honmachi, Sakyoku, Kyoto 606–8501, Japan