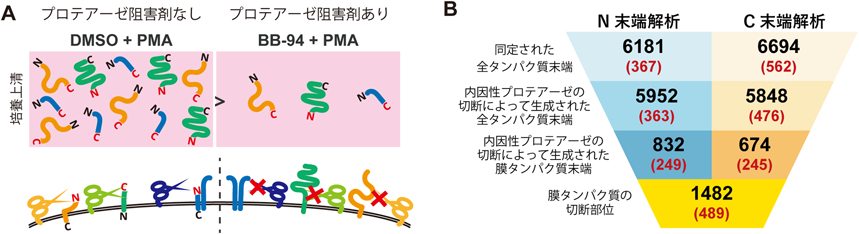
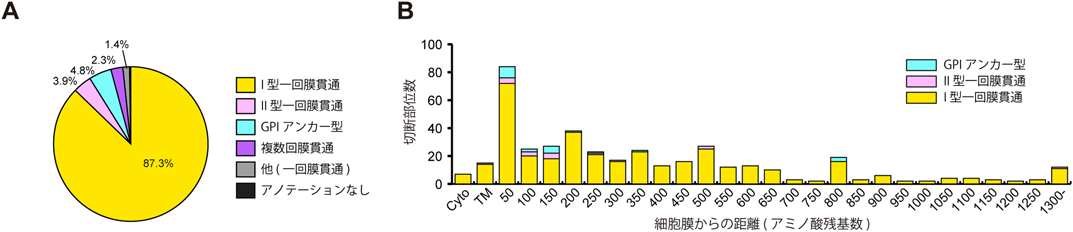
定量的末端プロテオミクスによるエクトドメインシェディング基質切断部位の大規模解析Large-scale identification of substrate cleavage sites of ectodomain shedding by quantitative protein terminomics
京都大学大学院薬学研究科生体分子計測学分野Department of Molecular Systems Bioanalysis, Graduate School of Pharmaceutical Sciences, Kyoto University ◇ 〒606–8501 京都市左京区吉田下阿達町46–29 ◇ 46–29 Yoshida-shimoadachi-cho, Sakyo-ku, Kyoto 606–8501, Japan