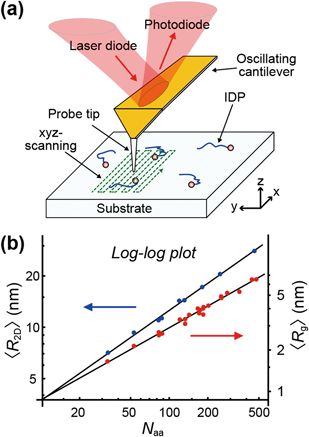
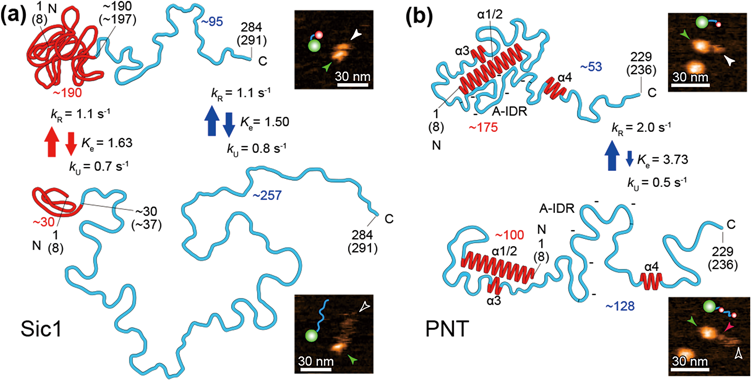
高速原子間力顕微鏡による天然変性タンパク質の動態観察Structural dynamics of intrinsically disordered proteins observed by high-speed atomic force microscopy
金沢大学ナノ生命科学研究所Nano Life Science Institute, Kanazawa University ◇ 〒920–1192 石川県金沢市角間町 ◇ Kakuma-machi, Kanazawa 920–1192, Japan