細胞核内にはゲノムDNA, RNA,タンパク質,その他,小分子などのさまざまな分子が存在し,非常に混雑している.多種の分子が混ざった環境で遺伝子発現などの核内事象がどのように起こるのかを理解することは,単純な試験管内の化学反応と複雑な細胞内反応ネットワークの間にあるギャップを埋めることにつながる.これまで核内構造体が分子を区画化することで,単一な素反応を超えた連続かつ複雑な化学反応が可能となると想像されてきたが,その実態は不明であった.近年,その一端が液–液相分離によって担われていることが明らかになりつつある1).液–液相分離とは,溶液が均一に混じり合わず,二相に分離する現象で,相分離して球状になった集合体は液滴(ドロプレットまたはコンデンセート)と呼ばれる.液滴は特定の分子を濃縮したり,別の分子を排除したりすることで,核内の転写,スプライシング(またはプロセシング),RNA修飾などの生化学反応を時空間的に制御できるポテンシャルを秘めている.
本稿では,最も大きな核内構造体である核小体と,特定の遺伝子の読み出しに伴い形成される「転写コンデンセート」に焦点を当てる.両者が液滴であることがわかってきた歴史的経緯から最新の研究知見を含めて,相分離が細胞の機能にどのように寄与するかを紹介する2, 3).細胞内で生じる液–液相分離の発見を通して,現在,分子生物学,生化学,細胞生物学,生物物理学は新たな潮流を迎えている.
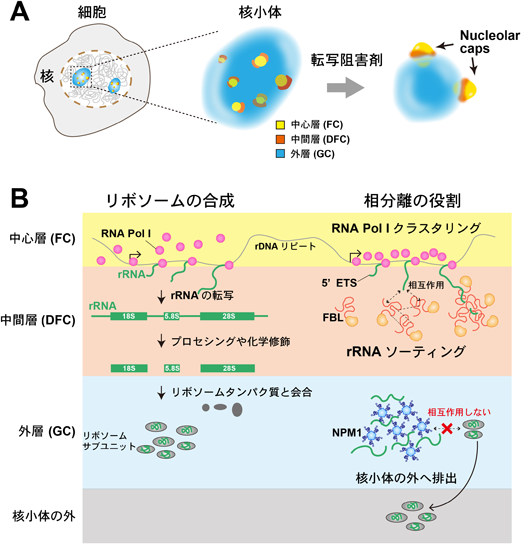
核小体はタンパク質翻訳装置であるリボソームを合成する場で,光学顕微鏡によって容易に観察できる膜のない核内構造体である.電子顕微鏡で核小体を観察すると,三つの層があらわれる(図1A中央).中心層はFC(fibrillar center)でリボソームRNA遺伝子(rDNA)とRNAポリメラーゼIが局在する.その周りを中間層であるDFC(dense fibrillar center)が囲む.これらが外層のGC(granules component)の中に散らばっている(図1A中央)4).この層構造は,リボソームの合成の工程と密接に連携している.リボソームの合成はFC/DFCの境界でRNAポリメラーゼIがrDNAを転写することによって始まる(図1B左上段).転写産物のリボソームRNA(rRNA)はDFCでプロセシングや修飾を受け(図1B左中段),GCでリボソームタンパク質などが取りつけられる(図1B左下段).rRNAの転写を薬剤などで阻害すると,この構造が大きく変化し,GC内に散らばっていたFCおよびDFCが融合しnucleolar capと呼ばれる大きな塊として核小体の縁に局在することが古くから知られていた.そのため,これらの特徴的な三層構造が核小体の機能に直結していると考えられてきた(図1A右)5).
次に核小体の物理的性質について考える.この際,重要となる一つの指標は核小体に超音波を照射しても壊れないことであろう6, 7).このため核小体は核骨格のように比較的に硬いものであると信じられてきた.実際,この性質を利用して超音波によって核膜を壊し,核から核小体を単離することができる.そして核小体を構成するタンパク質が質量分析計で網羅的に同定されたり,核小体内部やその縁に結合するゲノム領域(nucleolus-associated domain:NAD)が決定された8, 9).一方,蛍光タンパク質を用いたイメージング技術で生きている細胞の核小体を観察すると,ダイナミックな姿が浮かび上がってきた.光退色後蛍光回復法(fluorescence recovery after photobleaching:FRAP)により核小体の分子動態を調べると,内と外の分子がすばやく入れ替わるようすが観察された10–12).また細胞周期を通じて,二つの核小体が近接した場合,融合することからもその柔軟性は示唆されている13, 14).核小体は,超音波による物理的な衝撃に耐えるほど頑強であると同時に,互いが衝突すると融合するくらい柔らかいという相反する二面性を持っている.
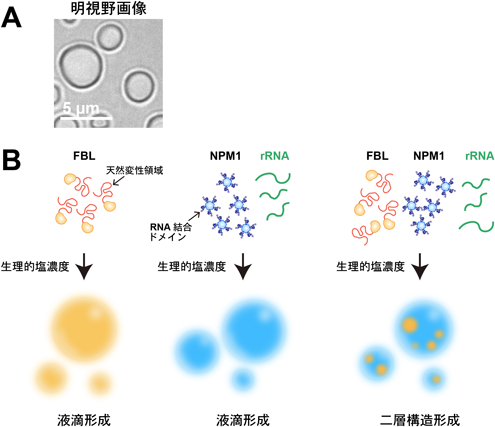
細胞内の膜のない構造体が液–液相分離によって形成されることは,生殖系の細胞内にあるP顆粒で初めて示された15, 16).P顆粒には,RNAとRNA結合タンパク質が濃縮されており,これらの分子どうしがつながること(多価の相互作用)により,形が維持されている17).また,特定の立体構造をとらない天然変性タンパク質もRNAなどとともに静電的または疎水性相互作用等,弱い多価結合によって,相分離することがわかってきた18).核小体のタンパク質の多くも天然変性領域をコードしている.Brangwynneらはその中から中間層DFCのタンパク質fibrillarinと外層GCのタンパク質nucleophosmin(NPM1)に着目し,これらの組換えタンパク質をrRNAとともに試験管内で混ぜることで,層構造を再構成することに成功した(図2)19).fibrillarinはN末端に天然変性領域をコードしており,NPM1はホモ五量体を形成し,それぞれのサブユニットでrRNAと結合する20, 21).生理的な条件でfibrillarinと,rRNAを加えたNPM1はそれぞれ独立に液滴を形成するが(図2B左と中央),2種類の液滴を混ぜるとより疎水的なfibrillarinの液滴がNPM1の液滴に包埋される(図2B右).興味深いことに,この試験管内で再構成された液滴は,時間経過とともにそのダイナミックな性質は失われ,より粘性の高いゲル状に変わっていく.液滴は周りの環境によって液体から固体状態へ遷移することから,核小体は核内の環境や人為的に精製するときの条件に応じてその物性が変わりうることを示唆する.確かにこれまで観察されてきた核小体が頑強性と柔軟性の二面性を持つことは相分離モデルによってうまく説明できる.
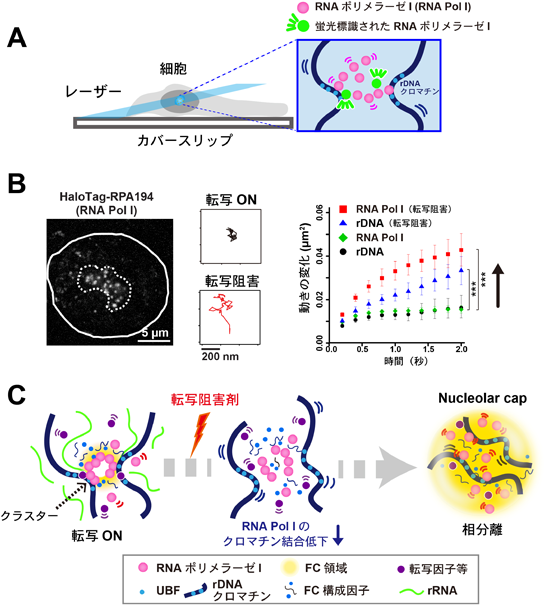
核小体の中間層と外層が相分離した液滴であることが明らかになる一方,rRNAの転写を担う分子からなる中心層FCが液滴かどうかはわかっていなかった.筆者らは超解像蛍光顕微鏡を用いて,生きた細胞のなかでrDNAやRNAポリメラーゼIの振る舞いを分子レベルで詳しく観察した(図3A,方法の詳細についてはキャプション参照)22–24).核小体内に存在するRNAポリメラーゼIは,転写が行われている状態ではrDNA上に集まって塊(クラスター)を作り,rDNAが動かないように抑えていた(図3B右の黒点と緑点,図3C左).一方で,RNAポリメラーゼIの阻害剤を加えると,RNAポリメラーゼIの塊が崩壊し,rDNAとともに流動的になり,核小体の縁に新たな液滴として存在していた(nucleolar cap,図3B右の青点と赤点,図3C右).つまり,リボソームの合成が行われ,三層構造が形成されているとき,中心層FCは液滴というよりむしろ固いゲルのような状態になる.一方,転写の阻害によりFCが融合してnucleolar capになると,液滴としての性質を示すことがわかった.このことは,中心層FCが球状ではなくいびつなリング状の形をしているという,超解像顕微鏡の一つである構造化照明顕微鏡(structured illumination microscopy:SIM)による観察と一致する25, 26).リボソームの合成時に何がFCの性質を変化させるかは重要な課題である.今後,FCの試験管内再構成実験などによる検証が待たれる.
相分離はどのように核小体の機能に寄与しているのだろうか? ここでは三つの可能性を議論する.一つ目として,相分離によって,リボソームの合成時,各層で必要な分子を区画化・濃縮することで,転写やプロセシングなどの生化学反応効率を高めていることが考えられる27).二つ目として中心層FC辺縁部で転写されたrRNAをリボソームの組立て工程と共役しながら,中間層,外層,そして核小体外へと運ぶ流れを生み出している.具体的には,転写されたばかりのrRNA前駆体はその5′側のexternal transcribed spacer(5′ ETS)領域がfibrillarinと結合するため,優先的に中間層に運ばれ(ソーティング),プロセシングを受けることが示されている(図1B右中段)26).また,外層で組み立て終えたリボソームサブユニットはNPM1との相互作用を失い,外層にとどまることができずに核小体の外へ排除される(図1B右下段)28).これは自動車のライン生産方式のような仕組みで,rRNAをベルトコンベアに乗せて順序よくプロセシングやタンパク質の取付作業をしているようにみえる.三つ目として,相分離がリボソームの合成とは別の核小体の機能にも関わることがあげられる.ヒートショックストレスによりフォールディングができなくなったタンパク質が核小体に濃縮することはよく知られている.これらが外層のNPM1に相互作用することにより核小体にトラップされ,核内全体に広がらないようにしていると考えられている29).
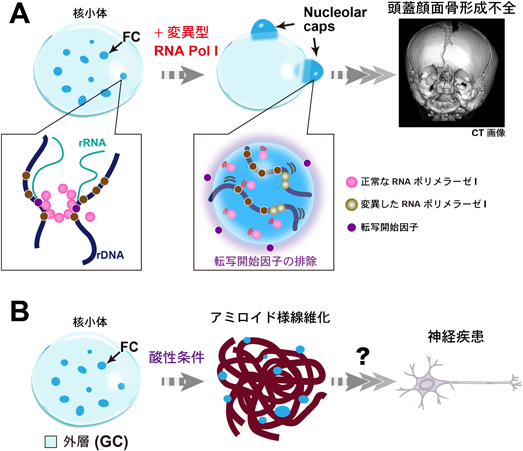
一方,核小体が液滴であるため,本来の役割とは別に細胞の機能を阻害することもある.筆者らは,ヒトの骨形成疾患(具体的には,頭蓋顔面骨形成不全症)の原因となる変異したRNAポリメラーゼIについて調べた24, 30).変異タンパク質を発現させると,阻害剤処理したときと同様に転写が停止してFCがnucleolar capのような液滴になることを見いだした(図4A)24).興味深いことに,このとき,転写開始に必要なタンパク質がnucleolar capから追い出されるため,転写が完全に止まっていた(図4A中央).以上のことから,相分離を介したリボソーム合成異常がヒト遺伝性疾患の原因になることが示唆された24).また別の例として,細胞内のpHが変化すると液滴である核小体の外層がアミロイド線維に似た状態に相転移を起こし,凝集体を形成することがあげられる(図4B)31).アミロイド様繊維は液滴やゲルと異なり不可逆で細胞の機能を阻害することから,神経疾患などへの関与が予想される.
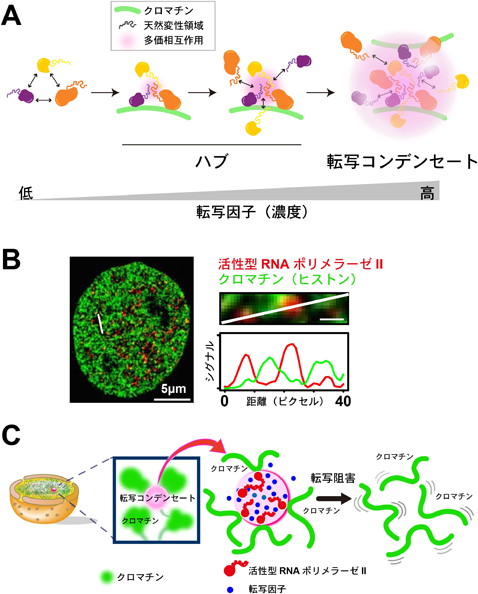
これまでリボソーム合成を担う核小体と相分離の関係について着目してきたが,次に遺伝子の読み出し,すなわちメッセンジャーRNAの転写に伴い形成されると考えられている液滴について紹介していく.遺伝子の転写は「転写ファクトリー」と呼ばれる核内の特定の場所で行われることは古くから提唱されてきた32–34).転写される一群の遺伝子が1か所に集められることで,転写の開始から伸長,終了まで,一連の反応過程が効率よく進められると考えられていたが,その実態は不明のままであった.遺伝子の転写は,DNA配列特異的に結合する転写因子が,プロモーターやエンハンサーといった転写調節領域に結合することで活性化する.多くの転写因子はDNA結合ドメインとは別に天然変性領域を持っている.最近,Chongらは高い時空間分解能を備えた顕微鏡を用い,細胞内での転写因子の振る舞いを4次元的に捉えた35).その結果,転写因子どうしが天然変性領域を介して相互作用し,濃度が低いときにはハブ(小さな集合体)を形成し,濃度が高くなると液–液相分離を引き起こして液滴(転写コンデンセート)を形成することを示した(図5A).この転写コンデンセートには天然変性領域をC末端ドメインに持つRNAポリメラーゼIIも濃縮されていることから,転写関連因子群の局所濃度を高める役割を担っていると考えられる36).この最たる例が多数のエンハンサーで構成されているスーパーエンハンサーである.転写因子とRNAポリメラーゼIIを橋渡しするメディエーター(mediator)やBRD4などの転写のコアクティベーターが,天然変性領域を介して弱く相互作用し,これらがスーパーエンハンサー領域に結合することで液–液相分離を誘導し,巨大な転写コンデンセートを形成する37).このような転写の活性化に伴う転写コンデンセートが従来考えられてきた転写ファクトリーの実態であることが推察される.最近,生きた細胞の核の中で遺伝子が転写される場所をリアルタイムに可視化することにより,転写の「開始」と「伸長」が別々の場所で行われていることが報告された38).転写コンデンセートも転写開始複合体と転写伸張因子などを別々に区分けして核小体のような多層構造を形成しているのかもしれない.メッセンジャーRNAの転写に伴い形成されると考えられている転写コンデンセートは機能的にもとても興味深い一方,核小体と異なり,これが液–液相分離による本当の液滴なのか?という強い反論が存在することは指摘しておく必要があるだろう3).
核小体や転写コンデンセートが相分離により形成された液滴であるとした場合,液滴がゲノムの配置や構造にどのような影響を与えるのかは興味深い.核小体や転写コンデンセートができると,その周りにあるクロマチンは押し出され空間的に排除されると考えられる.核小体の領域はDNAを染色したときに染まらず,空洞のように抜けてみえる.同様に,転写コンデンセートもDNA(もしくはヒストン)とは必ずしもオーバーラップせずに局在することが,筆者ら複数のグループから報告されている(図5B)23, 39).さらに,最近,光遺伝学手法を用いて核内に人工的な液滴を誘発した場合,その周りのクロマチンが押し出されることが示されている40).このことから長いポリマーであるクロマチンは相分離により効率的にフィルタリングされることが推察される.相分離は標的遺伝子以外のゲノム領域の混在を防ぐことで,誤って転写が行われたり,本来のプロセスの妨げになることを抑制していると考えられる.
一方,核小体や転写コンデンセートには親和性が高い特定クロマチン領域が集約されるので,核内のゲノム構造を形成するための要として機能しているのだろう.筆者らは,薬剤や遺伝学的トリックを使い,RNAポリメラーゼIIを含む転写コンデンセートを消失させると,ゲノムワイドにクロマチンの動きが上昇することを,一分子ヌクレオソームイメージング法によって見いだした(図5C)41).このことから,転写に伴う転写コンデンセートは,クロマチンを物理的に束縛しており,ゲノムDNAの収納様式やその機能において重要であることが示唆された.このことから,核内にはクロマチンによる緩いネットワークがあり,転写の場はネットワークの中核,文字どおり「ハブ」の役割を担っていることが推察される.また核小体もその外層の縁に転写の不活性なヘテロクロマチン領域NADを内包する.筆者らは,この領域のヌクレオソームの動きが周りに比べて物理的に束縛されていることを見いだしており,転写コンデンセートと同じようにゲノムDNAを収納するためのハブとして役割を果たしている可能性が考えられる23).
液–液相分離は細胞内の膜のない構造体を形成する基本原理として提唱された.相分離は核小体や転写コンデンセートのみならず,その他の多くのゲノムの制御に関わる核内構造体の形成にも関与している42).この原理により,生化学反応の効率が上昇し,異なる二つの反応を共役させることが可能となり,さまざまな核内プロセスが促進される.相分離モデルを基軸とし,生体分子が混雑した環境で起こる複雑な化学反応系がより深く理解されることを期待したい.
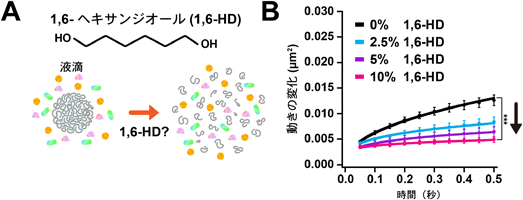
相分離の研究のさらなる進展のためには,今後イメージング技術の役割がより重要になる.一般的によく使用されるFRAP法は観察領域の内と外の分子の入れ替わりのようすを定量する方法なので,細胞内の構造体が液滴か否かを証明する手段にはならないと指摘されている43).生細胞内で直接分子の動きを計測する一分子イメージング技術は液滴の有無やその性質を検証するのに必須になる.その一例として,筆者らは一分子ヌクレオソームイメージング法を用いて,液滴の溶解に広く用いられている1,6-ヘキサンジオール(1,6-HD)(図6A)が,生きた細胞内で速やかにクロマチンを固定化・凝縮することを見いだしている(図6B)44).注目すべきは,この1,6-HDの作用は,液滴を溶解する活性(図6A)とはまったく異なることから,1,6-HDを用いて得られたクロマチンを含んだ液滴の評価は慎重に解釈するか再考する必要がある.細胞内相分離の仕組みの理解には,さらなる高速かつ高解像で汎用性の高いイメージング技術の開発が鍵となってくるだろう45).
謝辞Acknowledgments
本稿執筆にあたりご協力いただいた国立遺伝学研究所・ゲノムダイナミクス研究室の伊藤優志さん,田村佐知子さんに感謝いたします.
引用文献References
1) Hyman, A.A., Weber, C.A., & Julicher, F. (2014) Liquid–liquid phase separation in biology. Annu. Rev. Cell Dev. Biol., 30, 39–58.
2) Lafontaine, D.L.J., Riback, J.A., Bascetin, R., & Brangwynne, C.P. (2021) The nucleolus as a multiphase liquid condensate. Nat. Rev. Mol. Cell Biol., 22, 165–182.
3) Narlikar, G.J., Myong, S., Larson, D., Maeshima, K., Francis, N., Rippe, K., Sabari, B., Strader, L., & Tjian, R. (2021) Is transcriptional regulation just going through a phase? Mol. Cell, 81, 1579–1585.
4) Thiry, M. & Lafontaine, D.L. (2005) Birth of a nucleolus: the evolution of nucleolar compartments. Trends Cell Biol., 15, 194–199.
5) Reynolds, R.C., Montgomery, P.O., & Hughes, B. (1964) Nucleolar “caps” produced by actinomycin D. Cancer Res., 24, 1269–1277.
6) Busch, H., Muramatsu, M., Adams, H., Steele, W.J., Liau, M.C., & Smetana, K. (1963) Isolation of nucleoli*1. Exp. Cell Res., 24(suppl9), 150–163.
7) Muramatsu, M. & Onishi, T. (1978) Isolation and purification of nucleoli and nucleolar chromatin from mammalian cells. Methods Cell Biol., 17, 141–161.
8) Andersen, J.S., Lam, Y.W., Leung, A.K., Ong, S.E., Lyon, C.E., Lamond, A.I., & Mann, M. (2005) Nucleolar proteome dynamics. Nature, 433, 77–83.
9) van Koningsbruggen, S., Gierlinski, M., Schofield, P., Martin, D., Barton, G.J., Ariyurek, Y., den Dunnen, J.T., & Lamond, A.I. (2010) High-resolution whole-genome sequencing reveals that specific chromatin domains from most human chromosomes associate with nucleoli. Mol. Biol. Cell, 21, 3735–3748.
10) Dundr, M., Hoffmann-Rohrer, U., Hu, Q., Grummt, I., Rothblum, L.I., Phair, R.D., & Misteli, T. (2002) A kinetic framework for a mammalian RNA polymerase in vivo. Science, 298, 1623–1626.
11) Phair, R.D. & Misteli, T. (2000) High mobility of proteins in the mammalian cell nucleus. Nature, 404, 604–609.
12) Chen, D. & Huang, S. (2001) Nucleolar components involved in ribosome biogenesis cycle between the nucleolus and nucleoplasm in interphase cells. J. Cell Biol., 153, 169–176.
13) Brangwynne, C.P., Mitchison, T.J., & Hyman, A.A. (2011) Active liquid-like behavior of nucleoli determines their size and shape in Xenopus laevis oocytes. Proc. Natl. Acad. Sci. USA, 108, 4334–4339.
14) Caragine, C.M., Haley, S.C., & Zidovska, A. (2018) Surface Fluctuations and Coalescence of Nucleolar Droplets in the Human Cell Nucleus. Phys. Rev. Lett., 121, 148101.
15) Brangwynne, C.P., Eckmann, C.R., Courson, D.S., Rybarska, A., Hoege, C., Gharakhani, J., Julicher, F., & Hyman, A.A. (2009) Germline P granules are liquid droplets that localize by controlled dissolution/condensation. Science, 324, 1729–1732.
16) Elbaum-Garfinkle, S., Kim, Y., Szczepaniak, K., Chen, C.C., Eckmann, C.R., Myong, S., & Brangwynne, C.P. (2015) The disordered P granule protein LAF-1 drives phase separation into droplets with tunable viscosity and dynamics. Proc. Natl. Acad. Sci. USA, 112, 7189–7194.
17) Anderson, P. & Kedersha, N. (2006) RNA granules. J. Cell Biol., 172, 803–808.
18) Kato, M., Han, T.W., Xie, S., Shi, K., Du, X., Wu, L.C., Mirzaei, H., Goldsmith, E.J., Longgood, J., Pei, J., et al. (2012) Cell-free formation of RNA granules: low complexity sequence domains form dynamic fibers within hydrogels. Cell, 149, 753–767.
19) Feric, M., Vaidya, N., Harmon, T.S., Mitrea, D.M., Zhu, L., Richardson, T.M., Kriwacki, R.W., Pappu, R.V., & Brangwynne, C.P. (2016) Coexisting liquid phases underlie nucleolar subcompartments. Cell, 165, 1686–1697.
20) Berry, J., Weber, S.C., Vaidya, N., Haataja, M., & Brangwynne, C.P. (2015) RNA transcription modulates phase transition-driven nuclear body assembly. Proc. Natl. Acad. Sci. USA, 112, E5237–E5245.
21) Mitrea, D.M., Cika, J.A., Guy, C.S., Ban, D., Banerjee, P.R., Stanley, C.B., Nourse, A., Deniz, A.A., & Kriwacki, R.W. (2016) Nucleophosmin integrates within the nucleolus via multi-modal interactions with proteins displaying R-rich linear motifs and rRNA. eLife, 5, e13571.
22) Hihara, S., Pack, C.G., Kaizu, K., Tani, T., Hanafusa, T., Nozaki, T., Takemoto, S., Yoshimi, T., Yokota, H., Imamoto, N., et al. (2012) Local nucleosome dynamics facilitate chromatin accessibility in living mammalian cells. Cell Rep., 2, 1645–1656.
23) Nozaki, T., Imai, R., Tanbo, M., Nagashima, R., Tamura, S., Tani, T., Joti, Y., Tomita, M., Hibino, K., Kanemaki, M.T., et al. (2017) Dynamic organization of chromatin domains revealed by super-resolution live-cell imaging. Mol. Cell, 67, 282–293 e287.
24) Ide, S., Imai, R., Ochi, H., & Maeshima, K. (2020) Transcriptional suppression of ribosomal DNA with phase separation. Sci. Adv., 6, abb5953.
25) Maiser, A., Dillinger, S., Langst, G., Schermelleh, L., Leonhardt, H., & Nemeth, A. (2020) Super-resolution in situ analysis of active ribosomal DNA chromatin organization in the nucleolus. Sci. Rep., 10, 7462.
26) Yao, R.W., Xu, G., Wang, Y., Shan, L., Luan, P.F., Wang, Y., Wu, M., Yang, L.Z., Xing, Y.H., Yang, L., et al. (2019) Nascent Pre-rRNA sorting via phase separation drives the assembly of dense fibrillar components in the human nucleolus. Mol. Cell, 76, 767–783 e711.
27) Shin, Y. & Brangwynne, C.P. (2017) Liquid phase condensation in cell physiology and disease. Science, 357, eaaf4382.
28) Riback, J.A., Zhu, L., Ferrolino, M.C., Tolbert, M., Mitrea, D.M., Sanders, D.W., Wei, M.T., Kriwacki, R.W., & Brangwynne, C.P. (2020) Composition-dependent thermodynamics of intracellular phase separation. Nature, 581, 209–214.
29) Frottin, F., Schueder, F., Tiwary, S., Gupta, R., Korner, R., Schlichthaerle, T., Cox, J., Jungmann, R., Hartl, F.U., & Hipp, M.S. (2019) The nucleolus functions as a phase-separated protein quality control compartment. Science, 365, 342–347.
30) Weaver, K.N., Watt, K.E., Hufnagel, R.B., Navajas Acedo, J., Linscott, L.L., Sund, K.L., Bender, P.L., Konig, R., Lourenco, C.M., Hehr, U., et al. (2015) Acrofacial dysostosis, cincinnati type, a mandibulofacial dysostosis syndrome with limb anomalies, is caused by POLR1A dysfunction. Am. J. Hum. Genet., 96, 765–774.
31) Wang, M., Bokros, M., Theodoridis, P.R., & Lee, S. (2019) Nucleolar sequestration: remodeling nucleoli into amyloid bodies. Front. Genet., 10, 1179.
32) Cook, P.R. (1999) The organization of replication and transcription. Science, 284, 1790–1795.
33) Feuerborn, A. & Cook, P.R. (2015) Trends in genetics. TIG, 31, 483–490.
34) Edelman, L.B. & Fraser, P. (2012) Transcription factories: genetic programming in three dimensions. Curr. Opin. Genet. Dev., 22, 110–114.
35) Chong, S., Dugast-Darzacq, C., Liu, Z., Dong, P., Dailey, G.M., Cattoglio, C., Heckert, A., Banala, S., Lavis, L., Darzacq, X., et al. (2018) Imaging dynamic and selective low-complexity domain interactions that control gene transcription. Science, 361, eaar2555.
36) Kwon, I., Kato, M., Xiang, S., Wu, L., Theodoropoulos, P., Mirzaei, H., Han, T., Xie, S., Corden, J.L., & McKnight, S.L. (2013) Phosphorylation-regulated binding of RNA polymerase II to fibrous polymers of low-complexity domains. Cell, 155, 1049–1060.
37) Sabari, B.R., Dall’Agnese, A., Boija, A., Klein, I.A., Coffey, E.L., Shrinivas, K., Abraham, B.J., Hannett, N.M., Zamudio, A.V., Manteiga, J.C., et al.Cisse, II, Roeder, R.G., Sharp, P.A., Chakraborty, A.K., & Young, R.A. (2018) Science, 361, eaar3958.
38) Uchino, S., Ito, Y., Sato, Y., Handa, T., Ohkawa, Y., Tokunaga, M., & Kimura, H. (2022). J. Cell Biol., 221, e202104134.
39) Markaki, Y., Gunkel, M., Schermelleh, L., Beichmanis, S., Neumann, J., Heidemann, M., Leonhardt, H., Eick, D., Cremer, C., & Cremer, T. (2010) Functional nuclear organization of transcription and DNA replication: a topographical marriage between chromatin domains and the interchromatin compartment. Cold Spring Harb. Symp. Quant. Biol., 75, 475–492.
40) Shin, Y., Chang, Y.C., Lee, D.S.W., Berry, J., Sanders, D.W., Ronceray, P., Wingreen, N.S., Haataja, M., & Brangwynne, C.P. (2018) Liquid nuclear condensates mechanically sense and restructure the genome. Cell, 175, 1481–1491.
41) Nagashima, R., Hibino, K., Ashwin, S.S., Babokhov, M., Fujishiro, S., Imai, R., Nozaki, T., Tamura, S., Tani, T., Kimura, H., et al. (2019) Single nucleosome imaging reveals loose genome chromatin networks via active RNA polymerase II. J. Cell Biol., 218, 1511–1530.
42) Strom, A.R. & Brangwynne, C.P. (2019) The liquid nucleome-phase transitions in the nucleus at a glance. J. Cell Sci., 132, jcs235093.
43) McSwiggen, D.T., Mir, M., Darzacq, X., & Tjian, R. (2019) Evaluating phase separation in live cells: diagnosis, caveats, and functional consequences. Genes Dev., 33, 1619–1634.
44) Itoh, Y., Iida, S., Tamura, S., Nagashima, R., Shiraki, K., Goto, T., Hibino, K., Ide, S., & Maeshima, K. (2021) 1,6-hexanediol rapidly immobilizes and condenses chromatin in living human cells. Life Sci. Alliance, 4, e202001005.
45) Ide, S., Tamura, S., & Maeshima, K. (2022) Chromatin behavior in living cells: Lessons from single-nucleosome imaging and tracking. BioEssays, 2200043.
著者紹介Author Profile
 井手 聖(いで さとる)
井手 聖(いで さとる)国立遺伝学研究所ゲノムダイナミクス研究室助教.博士(バイオサイエンス).
略歴2006年奈良先端科学技術大学院大学バイオサイエンス研究科博士後期課程研究指導認定退学.国立遺伝学研究所細胞遺伝研究部門(小林武彦教授主宰)博士研究員,フランスCNRS人類遺伝学研究所DeJardin研究室JSPS海外特別研究員を経て,14年から現職.
研究テーマと抱負核小体と相分離の研究を通して,生命と非生命の間にあるギャップを埋める法則を知りたい.
ウェブサイトhttps://researchmap.jp/satide
趣味ボールルームダンス.
 前島 一博(まえしま かずひろ)
前島 一博(まえしま かずひろ)国立遺伝学研究所ゲノムダイナミクス研究室教授.博士(医学).
(Twitter @kazu_maeshima)
略歴1993年筑波大学第二学群生物学類卒業.99年大阪大学大学院修了,スイス・ジュネーブ大学Ulrich Laemmli研究室博士研究員,2005年理化学研究所今本細胞核機能研究室研究員,専任研究員を経て,09年より現職.
研究テーマと抱負生きた細胞の中でゲノムDNAにコードされている遺伝情報が検索され,読み出される原理を追求中.一緒に興味を持って追求してくれる方,募集中.
ウェブサイトhttp://maeshima-lab.sakura.ne.jp/index.html
趣味野球観戦,バッティングセンター通い.