生物は遺伝子発現の強度やタイミングを巧みに調節することで,発生や分化などの高次な生命現象を緻密に制御している.ワトソンとクリックによる二重らせんモデルの提唱以来1),細胞がどのようにDNAにコードされた情報を読み取るのかという謎は,生命科学における中心的な問いである.DNAを鋳型としてRNAを合成する「転写」は遺伝子発現の第一段階であり,生物が備える根源的な仕組みである.しかし一方で,転写の基本的な分子機構には依然として数多くの重要な未解決問題が存在する.真核生物において,転写活性は主にエンハンサーと呼ばれるゲノム中の調節領域を介して制御されている2).最新の網羅的なゲノム解析から,ヒトゲノム中には90万以上ものエンハンサー領域が存在すると見積もられている3).重要なことに,エンハンサーは標的遺伝子の上流・下流を問わず,数十~数百kbも離れた遠位から標的遺伝子の転写活性を制御できるという特筆すべき性質を持つ.しかし,エンハンサーがどのように遠く離れた位置に存在する標的遺伝子と特異的に相互作用し,転写を誘導するのかという基本的な作用機序は依然として未解明である.また,近年の定量的なライブイメージング解析から,個々の細胞における転写活性は数分から数時間単位でON・OFFを繰り返しながら不連続状に揺らいでいることが報告されている4–6).こうした不連続な転写反応は「転写バースト」と呼ばれ,細菌から高等真核生物にまで共通にみられる4, 7–9).しかし,転写バーストが引き起こされる仕組みについても,十分に理解されていない.
興味深いことに近年,多様な生命現象がタンパク質の液–液相分離とそれに伴う局在変化を介して動的に制御されることが相次いで報告されている10–13).転写制御に関しても,現在までに多くの核内因子が相分離活性を示すことが実験的に示されている.一般的に液–液相分離の誘導には,タンパク質内部の天然変性領域(intrinsically disordered region:IDR)と呼ばれる柔軟で特定の立体構造をとりにくい領域が重要な役割を担うことが示されている14–16).多くの配列特異的な転写因子やコアクティベーターなどのエンハンサー結合因子に加えて,RNA polymerase II(Pol II)自身もIDRを介して相分離を誘導し,局所的に濃縮された液滴を核内に形成することが報告されている17–19).そして,このように形成された液滴が,転写を促進する微小環境として働くという新たなモデルが最近大きな注目を集めている.相分離を介した転写制御モデルは,エンハンサーの作用機序や転写動態の制御機構を理解する上で重要な手がかりになる可能性を秘めている.しかし現時点では,転写装置の相分離活性が生体内においてどれほど遺伝子発現に寄与しているのかといった機能的な側面からの理解は十分ではないことから,現在提唱されているモデルについて,今後実験的に注意深く検証していく必要がある.
本稿では,最近報告された転写装置が示す液–液相分離現象を,因子の機能ごとに大別して紹介する.後半では,RNAを介した相分離活性の制御や,形成された液滴がどのように遺伝子発現に影響を及ぼすのかについて,転写活性化の「ハブ」という新たな観点から説明する.最後に,転写因子の相分離活性とtopologically associating domain(TAD)などの立体的なゲノム構造との関連性について,最新の知見を紹介する.
1)転写因子
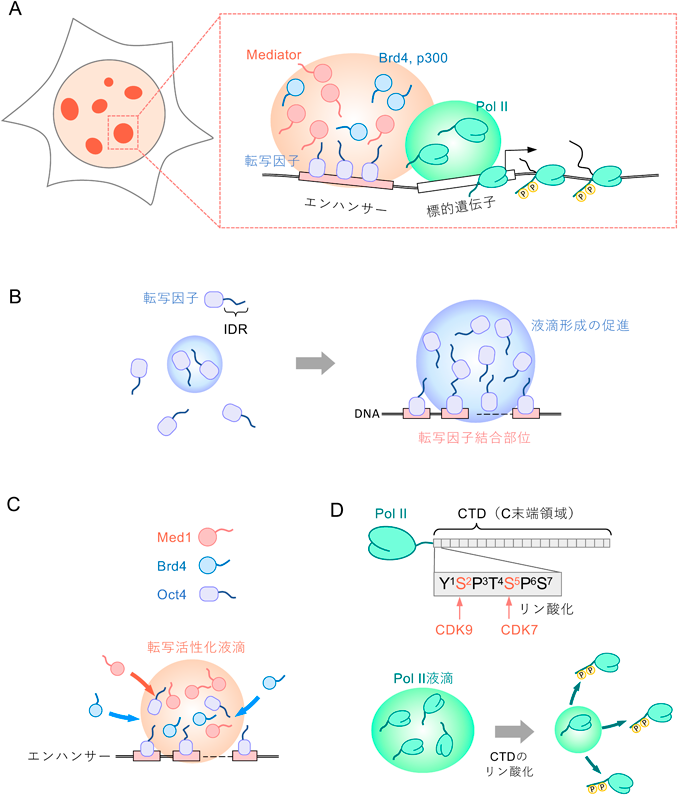
転写活性の強さやタイミングは,ゲノム中のエンハンサーと呼ばれる調節領域によって緻密に制御される.試験管内で再構成された転写反応系とは異なり20, 21),細胞内ではPol IIや基本転写因子のみでは転写反応を高レベルに誘導することはできない.配列特異的にDNAに結合する転写因子がエンハンサー上に呼び込まれることで,標的遺伝子からの転写活性が大きく促進される.興味深いことに,超解像度顕微鏡を用いて転写因子を直接可視化すると,それらは必ずしも核内に均一に存在しているわけではなく,局所的に濃縮されたクラスターを形成するようすが頻繁にみられる22–29).特に,転写因子を介してエンハンサー上に形成される数十から数百nmスケールの相分離を介して形成される液滴はtranscription hubやtranscriptional condensateと呼ばれ30–34),近年大きな注目を集めている(図1A).転写因子の多くはIDRを有しており35, 36),IDRどうしの多価性のタンパク質間相互作用を介した相分離が液滴の形成を促進すると考えられている.ある転写因子が形成する液滴は,複数の異なるタンパク質と同時に相互作用することで,他の転写因子やコアクティベーターなどを含んだ反応区画を形成することが示唆されている.またFRAP解析などから,液滴内部ではその構成因子が動的に移動・拡散することも示されている12, 37–39).実際に試験管内において,Myc, Nanog, Sox2, RARa, GATA2やエストロゲン受容体といったさまざまな転写因子が液–液相分離を起こし,タンパク質が局所的に濃縮された液滴を形成する24).ヒトの四肢形成を制御する転写因子であるHoxD13も同様に,アラニンの連続したリピート配列によって構成されるIDRをN末端側に含んでおり,試験管内において相分離活性を示す28).人為的にアラニンのリピート配列を伸長させると,IDRを介した多価性相互作用が異常亢進し,結果として液滴を形成する活性が異常に高まる.四肢形成異常がみられる患者では,HoxD13のアラニンリピートの伸長が頻繁にみられることを考えると,液滴形成効率の変化が疾患発症の原因となる可能性が考えられる28, 40, 41).
重要なことに,単独のリコンビナントタンパク質として精製された転写因子の挙動は,必ずしも細胞内の転写調節機能を十分に反映していない可能性もある.たとえば,パイオニア転写因子で,多能性幹細胞の誘導に必要な山中因子の一つとしても知られるOct4の精製タンパク質は単独では試験管内で液滴を形成しないが,Mediatorと呼ばれるコアクティベーターのサブユニットであるMed1と混合した場合において,2種類のタンパク質が共局在する液滴を形成する24).酵母の転写因子GCN4の液滴形成も同様に,MediatorサブユニットであるMed15の存在によって大きく促進される24).加えて,DNAが反応系に存在することで,転写因子の液滴形成がさらに促進されることも報告されている.Oct4とMed1の混合系では,Oct4結合部位を含むDNAを試験管内に加えることで,液滴形成能が非線形的に急激に上昇する31).Oct4と同様に,パイオニア転写因子であるKlf4を用いた実験系においても,結合配列を含むDNAによってKlf4の液滴形成が大きく促進される42).これらの現象は,転写因子がDNA中の結合配列を足場として局所的にタンパク質を濃縮させ,IDRを介したタンパク質間相互作用を促進し,相分離活性を上昇させていることを強く示唆している(図1B).転写因子以外にも,ヘテロクロマチンタンパク質HP1の相分離活性がDNAを介して制御されることが示されている43).DNA結合タンパク質と同様に,RNA結合タンパク質の液滴形成が基質となるRNA分子の存在によって大きく促進されることも相次いで報告されている44, 45).このように核酸を足場にして相分離を誘導する活性は,細胞内のさまざまな場面でみられる.試験管内における結果と同様に,マウスES細胞においてもOct4はMed1と共局在しながら核内に顆粒状の液滴を形成する.Oct4–Med1の液滴は,特にスーパーエンハンサーと呼ばれる巨大なエンハンサー領域で顕著に形成されており,標的遺伝子からの高レベルでのRNA産生を誘導している24).重要なことに,Oct4のIDRに変異を導入すると,試験管内だけでなく細胞内においてもMed1と共局在する活性が失われ,標的遺伝子の転写を誘導する能力も大きく低下する.これらの結果は,転写因子はIDRを介して転写活性化に働く液滴をエンハンサー上に作り出し,標的遺伝子を制御しているという考えと一致している.興味深いことに,活性化に働く転写因子だけでなく,転写リプレッサーやポリコーム群タンパク質などの抑制因子も,試験管内や細胞内において相分離を介した液滴を形成することが報告されている46–48).特に転写リプレッサーの液滴は,標的遺伝子近傍から転写活性化因子群を排除する抑制的な核内区画として機能することが示唆されている48).このように,転写の活性化因子や抑制因子が互いに物性の異なる相互排他的な液滴を形成し,それぞれの反応を区画化することで,生体内における転写活性が巧みに制御されている可能性が考えられる.次節では,転写活性化において重要な役割を担うMediatorなどのコアクティベーターと相分離の関連性について議論する.
2)コアクティベーター
配列特異的な転写因子は,MediatorやBrd4, p300/CBPといったコアクティベーターをエンハンサー上に呼び込むことで,標的遺伝子の転写活性を上昇させる.Mediatorは20種類以上のサブユニットから構成されるタンパク質複合体であり,遺伝子のコアプロモーター近傍への転写開始前複合体の形成を促進する49).これまでMediatorは,エンハンサー上に結合した転写因子と,遺伝子のコアプロモーター上における転写開始前複合体との間を橋渡しすることで安定的なループ構造を形成し,エンハンサーからの転写活性化シグナルを標的遺伝子へと伝達していると考えられてきた50–52).興味深いことに超解像顕微鏡を用いたライブイメージング解析から,MediatorサブユニットであるMed19がマウスES細胞においてPol IIとともに核内で液滴状のクラスターを形成することが報告された17).Med19が形成するクラスターは転写活性化領域と共局在しているが,JQ1などの転写阻害剤の存在下において消失することから,転写活性とMediatorによる液滴の形成は密接に共役していることが示唆された.また1,6-ヘキサンジオールなどの相分離阻害剤で細胞を処理した場合においても,核内におけるMediatorのクラスターは消失することから,転写因子と同様に相分離を介してその局在が制御されていることが示唆されている.同様に,Mediatorの主要サブユニットであるMed1は試験管内において自身の持つIDRを介して液滴の形成を誘導する53).細胞内では,Med1はスーパーエンハンサー領域に特に高いレベルで濃縮されており,標的遺伝子からの新生RNA合成の場として機能するようすが観察されている53).Mediator以外の主要なコアクティベーターであるBrd4も,核内においてMed1と同一のスーパーエンハンサー領域に高レベルで濃縮されることが報告されている53).Brd4は転写伸長促進因子であるP-TEFbを介してPol IIの活性を促進する機能を持つ54, 55).これらのことを踏まえると,MediatorとBrd4が共局在するハブは,標的遺伝子へのPol IIの呼び込みと伸長反応の両方を同時に促進する反応区画として働く可能性が考えられる.Brd4の精製タンパク質もまた試験管内においてIDRを介して液滴を形成し,Med1など他の異なる因子と共局在することができる53).前節で紹介したOct4の相分離活性を踏まえると,転写因子がエンハンサーへと配列特異的に結合することで相分離を誘導する足場が形成され,さらにそこへMediatorやBrd4などのコアクティベーターが引き込まれることで,転写活性化に働く液滴が生み出されるというモデルが考えられる(図1C).加えて,Brd4と相互作用するP-TEFbも細胞内において転写伸長の促進に働く液滴を形成することが報告されている56, 57).相分離活性を介して,エンハンサー上に転写因子の結合部位数以上の数のタンパク質群が動的に呼び込まれ,転写開始から伸長段階までを高効率に促進している新たな可能性が考えられる30).
また最近,エンハンサー領域から転写される非コードRNAであるenhancer RNA(eRNA)が,エンハンサー上におけるハブの形成を促進するという興味深い実験結果が報告された.eRNA中のアデニン塩基がメチル化(m6A修飾)されると,リーダータンパク質であるYTHDC1がエンハンサー上に呼び込まれる.YTHDC1はIDRを有しており,m6A修飾を持つeRNAとの相互作用を介して相分離を誘導し,Brd4を液滴へと呼び込む活性を示す58).このように液滴の形成やその局在様式は,転写因子だけではなく近傍のゲノム領域に存在するRNAなどの影響を強く受ける可能性がある.Brd4やMediator以外にも,ヒストンアセチル基転移酵素であるp300/CBPと呼ばれるコアクティベーターも自身のIDRを介して液滴を形成し,転写の活性化に働くことが示唆されている59, 60).p300/CBPはRNA結合活性を有しており,eRNAなどとの相互作用によってアセチルトランスフェラーゼ活性が上昇することも報告されている61).さらに,p300/CBPによって導入されたH3K27Acなどのヒストン修飾は,Brd4のブロモドメインによって直接認識される足場として働くことで,液滴へのBrd4の呼び込みをよりいっそう促進している可能性がある.以上のように,エンハンサー上における転写活性化の反応区画の形成は,転写因子やコアクティベーター間における協調的作用によって高度に制御されているものと考えられる.
3)RNAポリメラーゼII
Pol IIはDNAを鋳型としたmRNAの合成を直接担う必須のタンパク質である62–64).Pol II自身の活性制御においても,液–液相分離が重要な役割を果たすという新たな実験結果が報告されつつある.
重要なことにPol IIは核内において均一ではなく,クラスターを形成しながら不均一に存在していることが30年近く前から知られている65, 66).Pol II以外のPol IやPol IIIについても同様に,核内でクラスターを形成する性質が共通に観察されている67, 68).超解像顕微鏡を用いてPol IIクラスターの形成動態を解析すると,新生mRNAの転写量とPol IIの局所濃度との間には明瞭な正の相関関係がみられる69, 70).このことから,特定の遺伝子座からの転写量調節において,Pol IIクラスターの形成が重要な役割を担う可能性が示唆される.液–液相分離を示す多くのタンパク質と同様に,Pol IIの最大サブユニットであるRpb 1はそのC末端領域(C-terminal domain:CTD)に,Y1S2P3T4S5P6S7の七つのアミノ酸残基の繰り返しからなるIDRを有する(図1D).この7アミノ酸のコンセンサス配列の数は,ヒトでは52回,ショウジョウバエでは43回,出芽酵母では26回というように生物種によって異なる.精製されたCTDは試験管内において,FETファミリータンパク質であるFUS, EWF, TAF15のハイドロゲルや,cyclin T1の形成する液滴に取り込まれ,共濃縮されるという重要な性質を示す56, 71).さらに,CTDは試験管内や細胞内においてMed1が形成する液滴とも共局在することができる18).ヒトや酵母の精製CTDは単独でも試験管内で液滴を形成するが19),その活性はコンセンサス配列のリピート数の改変に伴って大きく変化する.細胞内でCTDのリピート数の異なるRpb1を発現させると,試験管内で強い液滴形成能を示すRpb1は,液滴形成能の弱い低リピート型の変異体に比べて,核内におけるPol IIクラスターの形成をより高いレベルで引き起こしていた.重要なことに,Pol IIクラスター近傍のゲノム領域では転写バーストが効率的に引き起こされるようすが観察されている69, 70).また,出芽酵母においてCTDのリピート数を短くすると,転写バーストの頻度や大きさが低下することが報告されている72).以上の知見を合わせると,Pol IIはCTDのリピート配列依存的にクラスターを形成し,その局所濃度を上昇させることで,転写バーストの誘導を促進している可能性が考えられる.すなわちエンハンサー上に形成された転写因子やコアクティベーターの液滴は,局所的に濃縮されたPol IIのリソースを消費することで,標的遺伝子へのPol IIの呼び込み,および転写伸長反応を促進しているという新たな作用機序が示唆される.
Pol IIが遺伝子のコアプロモーター領域に呼び込まれると,CTDを構成するY1S2P3T4S5P6S7の2番目(Ser2)および5番目のセリン残基(Ser5)が,CDK9やCDK7のキナーゼ活性によりリン酸化される(図1D).まず初めにCDK7を介したSer5のリン酸化によって転写開始が促進され,その後CDK9を介したSer2のリン酸化により転写伸長反応が促進される73).興味深いことに,リン酸化されたCTDはFUSのハイドロゲルやMed1の液滴内から離脱する18, 19, 71).この結果を踏まえると,リン酸化前の転写反応にまだ参加していないPol IIが核内において積極的にクラスターを形成し,その局所的な濃度上昇に寄与していることが示唆される.エンハンサーがこのリソースを利用してPol IIを標的遺伝子へと呼び込み,CTDのリン酸化が起こることで,Pol IIはクラスターを離脱して転写伸長反応へと突入すると考えられる18, 19).転写伸長中の新生RNAに作用するスプライシング因子もまた,核内において局所的に濃縮された液滴を形成する18).特筆すべきことに,こうしたスプライシング因子の液滴は,Mediatorが形成する液滴とは融合せずに隣接して存在している.このとき,CTDがリン酸化さていない転写開始前のPol IIはMediatorの液滴に取り込まれるのに対し,リン酸化され転写伸長段階にあるPol IIは隣接するスプラシング因子の液滴へと移行する.すなわち,転写が開始段階から伸長段階へと進むにつれ,Pol IIが新生RNAプロセシングのための液滴と共局在することで,効率的な遺伝子発現に寄与していると考えられる.
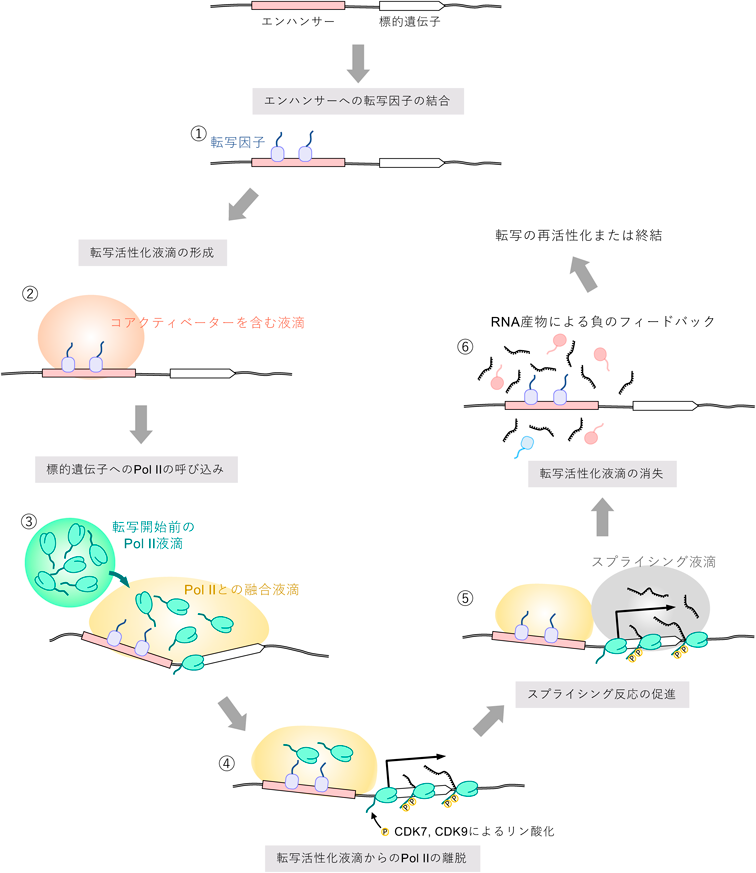
前述のとおり,エンハンサー領域から産生されるeRNAがm6A修飾を介して液滴の形成を促進することが報告されている58).その他にも,新たに産生されたRNA分子が近傍のゲノム領域におけるMed1の相分離活性に大きな影響を及ぼすことが,マウスES細胞を用いたレポーター解析から示唆されている74).エンハンサーや標的遺伝子本体から転写が起こり始めた直後で,新生RNAがそのゲノム領域に低濃度で存在する条件では,転写開始部位近傍におけるMed1の液滴形成が促進され,さらに転写活性が上昇するという正のフィードバックが起こる.一方で,ある一定のレベルまで転写活性が上昇し,局所的な新生RNAの濃度が閾値を超えると,逆にMed1による液滴の形成に対して阻害的に作用し,結果として転写が負に制御される.現時点で,新生RNAを介した転写抑制作用の分子基盤は明らかとなっていないが,負の電荷を持つRNA分子の局所的な集合がMed1の液滴を消失させる要因として働く可能性が示唆されている.負のフィードバック効果は,産生されるRNA分子の長さに大きく依存して変化する.
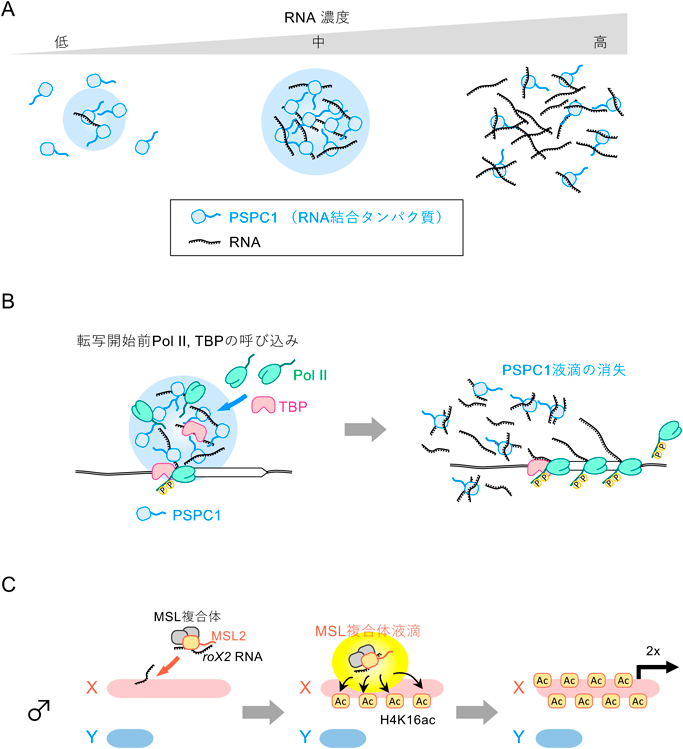
また最近,RNAを介した液滴の形成制御に,核内に局在するRNA結合タンパク質が重要な役割を担うことが報告された75).マウスES細胞のクロマチン画分に含まれるRNA結合タンパク質のうち,パラスペックルの構成タンパク質であるPSPC1(paraspeckle component 1)は,RNA結合ドメインに加えてIDRを有しており,試験管内において液滴を形成する活性を示す.そこへ精製したPol IIのCTDや,基本転写因子であるTBP(TATA binding protein)を加えると,PSPC1液滴内にそれらが同時に取り込まれる.興味深いことに,PSPC1とCTD/TBPによる液滴形成は,低濃度のRNA存在下において促進される一方で,高濃度のRNA存在下では顕著に阻害される(図2A).細胞内では,PSPC1は遺伝子の転写開始点近傍に主に結合している.これらの結果を踏まえると,新生RNAの局所的濃度が低い段階では,PSPC1が液滴の形成を介してPol IIやTBPなどの基本転写因子を積極的に呼び込み,転写を促進していると考えられる.一方で,転写反応が進行し新生RNAの局所濃度が上昇すると,PSPC1の液滴形成は阻害され,それと同時に転写開始点近傍へのPol IIやTBPの積極的な濃縮作用が失われる.以上のような仕組みを介して,RNAを介した正と負の遺伝子発現フィードバック機構が,転写反応の進行に伴って段階的に働く可能性が考えられる(図2B).
前述のとおり,新生RNAの局所的濃度が低い転写初期の段階では,PSPC1の液滴形成は促進され,Pol IIやTBPなどの因子を転写開始点近傍に呼び込む反応を補助している.PSPC1の他にも,RNA分子がタンパク質の液–液相分離を促進する例は数多く報告されている45, 76, 77).以前から液–液相分離活性を示すことが知られている代表的なタンパク質であるhnRNPA1やFUSは,IDRに加えてRNA結合ドメインを有しており,RNAに結合することで相分離が促進される45, 76, 78, 79).こうした背景を踏まえると,核内に局在するRNAの存在様式が個体発生・細胞分化過程に応じて変化することで,ある特定のタンパク質群を含んだ液滴の形成が動的に制御されている可能が考えられる.実際に,RNAを介してゲノム上における液滴形成が制御される機構の一つとして,ショウジョウバエ性染色体における遺伝子発現量補償に必須の非コードRNAであるroX2(long non-coding RNA on X 2)について紹介する80, 81).ショウジョウバエではXXが雌,XYが雄であるが,哺乳類とは異なり雄のX染色体からの転写量を倍にすることで,雌雄間の染色体数に起因する発現量の差を補っていると考えられている82–84).X染色体全体に及ぶ発現量補償を可能とするのは,雄特異的に発現するMSL(male-specific lethal)と呼ばれる複合体であり,roX2 RNAはその構成因子の一つである85–87).MSL複合体はヒストンアセチル化活性を有しており,雄のX染色体全域にわたってヒストンH4の16番目のリシン残基にアセチル化修飾(H4K16ac)を導入する88–90).MSL複合体の構成タンパク質であるMSL2はC末端側にIDRを有している.この領域は試験管内において,MSL2の結合基質であるroX2 RNA存在下でのみ,液滴を形成することができる91).興味深いことに,ショウジョウバエ由来のroX2とMSL2をマウスES細胞で異所的に発現させると,それらは核内に液滴を形成しながら,roX2 RNA産生領域近傍に位置する遺伝子の転写活性を上昇させた.このことから,X染色体から転写されたroX2 RNAが足場として機能することで,MSL2の相分離が誘導され,近傍の遺伝子領域に転写活性化のための因子群が呼び込まれるという新たなモデルが提唱された(図2C).実際に,MSL2のC末端側にあるIDRを特異的に欠失させた変異体ショウジョウバエでは,roX2 RNAへの結合活性は保持されているものの,X染色体からの発現量を十分に補償することができず,結果として雄は致死となる.このように,MSL2のIDRを介した相分離活性は,X染色体全域にわたるMSL複合体の呼び込みに重要な役割を果たしていることが示唆された.こうした研究から得られた知見をもとに,将来的にRNAを足場とした液滴形成を人為的に操作する技術が開発されれば,さまざまなゲノム機能を自在に制御するためのツールとして応用できる可能性がある.
1)高次ゲノム構造と液–液相分離
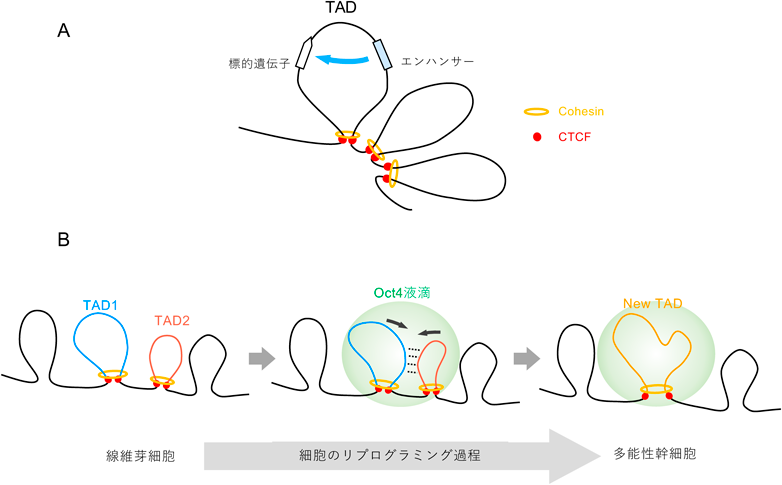
転写制御に働く液滴は,標的遺伝子の活性だけではなく,周囲の立体的なゲノム構造にまで大きな影響を及ぼすことが明らかとなってきた.Hi-C解析などの網羅的解析手法の発展に伴い,ゲノムの折りたたまれ方や,その立体的な構造変化が制御される仕組みについて飛躍的な理解がもたらされつつある.なかでもゲノム内で頻繁に相互作用し合う数kb~1 Mb程度の領域はTADと呼ばれる.TADは相互作用するエンハンサーと標的遺伝子との物理的な近接度を調節することで,特異的な転写制御を可能にする構造的基盤として働くと考えられている(図4A)92, 93).発生や細胞分化の過程においてTADが再編成されると,相互作用するエンハンサーとプロモーターの組合わせが変化し,組織特異的な遺伝子発現プログラムへと切り替えられる94).ヒトやマウスなどの哺乳細胞では,CTCFと呼ばれるZn-finger型のDNA結合タンパク質や,コヒーシン(cohesin)と呼ばれるリング型のタンパク質複合体が,TADの形成において中心的な役割を果たす92, 95).ごく最近の研究によって,これらの因子に加えて,転写因子が生み出す液滴がTADの形成に大きな影響を与えることが示された.前節で紹介したように,パイオニア転写因子として働くOct4は核内において液滴を形成する活性を持つ24, 31).またよく知られているように,Oct4は多能性幹細胞の誘導に必要な山中因子の一つでもある96).マウス胎仔線維芽細胞から多能性幹細胞へのリプログラミング過程においてHi-C解析を行うと,一度構築された複数のTADが大きな単一のTADへと再編成されるようすが観察された.このときOct4を可視化すると,自身が形成する液滴内部に複数の異なるTADを取り込むことで,それらが互いに近接する中間状態を作り出しているようすが観察された.つまりOct4の液滴が複数のTADどうしを融合し,新たな単一のTADへと再編成するための足場として機能する新たな可能性が示唆された(図4B)97).試験管内や細胞内でみられるOct4の相分離活性は,TADの再編成活性,およびリプログラミング効率と相関することも実験的に示されている.以上を踏まえると,Oct4などの転写因子が形成する液滴は,転写活性だけではなく立体的なゲノム構造やその核内配置を動的に変化させる足場として,細胞運命の決定に働いていると考えられる.細胞分化やリプログラミングの過程以外にも,出芽酵母では熱応答性の転写因子であるHsf 1が,熱ストレス存在下においてMediatorとともに液滴を形成し,立体的なゲノム構造の再編成を促進することが報告されている98).熱応答時には異なる染色体間どうしの相互作用が高頻度で起こることを踏まえると99),Hsf 1の形成する液滴が,通常の生育環境では物理的に離れたゲノム領域どうしを物理的に近接させるためのハブとして機能することが考えられる.
2)DNAの力学的張力を生み出す液–液相分離
よりミクロなスケールで,生物物理学的な視点に立ったエンハンサーの作用機構と相分離の関連性についても,近年興味深い報告がなされている.転写因子が形成する液滴はそれ自身がDNAを手繰り寄せる物理的な張力を生じさせる可能性が示された100).パイオニア転写因子の一つであるFoxA1(Forkhead box protein A1)の精製タンパク質とDNAを混合したところ,ガラス基盤に繋留したDNA表面にFoxA1が濃縮され,液滴が形成された.光ピンセットを用いた計測と理論的解析から,FoxA1液滴が外部のDNAを引き込んでさらに成長しようとする過程において,DNAを手繰り寄せる方向に張力が生じている可能性が示されている.こうした物理的な力は,生体内ではエンハンサーとプロモーター領域のようにゲノム中に離れて存在するDNAどうしを物理的に近接させる効果があると考えることができる.現時点では,生細胞内でどの程度こうした張力が転写制御やTADに寄与しているのかは不明である.しかし,DNA上における転写因子の物性変化がエンハンサーの長距離作用や立体的なゲノム構造の変化を促進するという可能性は大変興味深い.液相や固相界面の分子挙動の解析は物理・ソフトマター分野で大きく進んでいることから,今後こうした新たな視点を取り入れながらゲノムの働きを理解していくことがよりいっそう重要になっていくと考えられる101, 102).
本稿では,液–液相分離を介した分子集合と転写制御の関連について最新の知見を紹介してきた.転写活性化のハブは,一群の転写装置を液滴内部に呼び込むことで,転写開始から伸長段階までを高効率に促進することに大きく寄与していると考えられる.一方で,液滴内部で実際にどのように標的遺伝子の転写動態が経時的に制御されているのかといった機能的側面からの理解については,全容の理解からは依然として遠い.今回紹介した報告も含め,現時点で得られている知見は主にスーパーエンハンサーと呼ばれる特殊なゲノム領域をモデルとした観察事例に偏っており17, 24, 53),ハブの形成が典型的なエンハンサー領域でも共通して起こっているのかといった一般性については,今後さらに検証を重ねていく必要がある.また,転写因子の相分離活性はその転写活性化能と相関しているものの,相分離が起こること自体が転写活性化の直接的な原因なのか,あるいはその結果にすぎないのかという因果関係についても,依然として議論の余地が残されている103–105).
生体分子の相分離は,ともに濃縮されるパートナー分子を互いに選択しながら,特異的な機能を持った反応区画を形成するという重要な性質を示す.こうした液滴形成の特異性は,試験管内ではしばしばタンパク質のIDRだけで再現される18, 28, 53).最近のCRISPRライブラリーを用いた大規模スクリーニングと網羅的なインタラクトーム解析を組み合わせた研究から,転写因子ごとに相互作用するコアクティベーターの組合わせが明確に異なっているようすが示されている106).転写因子のIDRは特定の立体構造をとらず,安定的な分子複合体を作らなかったとしても,そこには細胞内に存在する膨大な種類の分子をふるいにかけ,適切なタンパク質とのみ相互作用するための情報が書き込まれている.さらにIDRの持つこうした情報は,リン酸化やアセチル化などの翻訳後修飾によって書き換えられ,結果として相分離活性とその特異性に大きな影響を与えていると考えられる18, 19, 107).しかし,こうした生体分子の相分離液滴の物性を決定している分子基盤の多くが未解明の状態であることから,今後の研究の進展が期待される.