生命科学に関連したデータベースが生化学や分子生物学に果たす役割はいまさら詳述する必要もないだろう.塩基配列やアミノ酸配列,タンパク質の立体構造などのデータが整備されるとともに,配列検索などのコンピュータプログラムが開発され,膨大なデータへのアクセスが日常的に可能となった.このように,データベースは生命科学のインフラストラクチャとしての役割を果たしている.我々のグループでは,10年にわたり天然変性タンパク質データベースIDEAL(https://www.ideal-db.org/)を開発・維持してきた1).天然変性タンパク質の発見は,生合成されたポリペプチド鎖がフォールドして固い立体構造をとり機能する,というタンパク質のこれまでの概念を刷新した.天然変性タンパク質の初期の報告に,Kriwackiらが発表したp21の実験がある2).p21は細胞周期の進行をつかさどるCDK2を結合阻害し細胞周期を制御する.p21のN末端側領域は単独では構造を形成しない天然変性領域だが,CDK2と共存させるとこの領域がCDK2に結合し立体構造を形成する.このような現象は“coupled folding and binding”(結合に伴う折りたたみ)として,天然変性タンパク質特有の性質として認知されている3).我々はこの「結合に伴う折りたたみ」に関わる領域をProtean Segment(ProS)と名づけ,天然変性領域とともに収集し,IDEALに登録してきた.IDEALでは,2022年3月の段階で1110のタンパク質,冗長性を除いた13,000ほどの天然変性領域と700ほどのProSを公開している.
上述したようなProSを介した相互作用は,天然変性タンパク質研究の中心的課題の一つではあるが,近年,天然変性タンパク質をめぐる研究動向には変化が生じている.最も大きな変化は液–液相分離(liquid–liquid phase separation:LLPS)との関連だろう.天然変性領域,特に低複雑性領域(low complexity region:LCR)と呼ばれる,少数のアミノ酸残基から構成される残基バラエティの低い領域が,LLPSを駆動することは広く認識されつつある.現在,LLPSに関連するデータベースが四つほど公開されており,タンパク質,核酸や実験の情報が収録されている.四つのLLPS関連データベースではデータの収集方針が異なっているので,内容には違いがあるだろう.また,収録されたタンパク質の多くは天然変性タンパク質だと類推されるが,LLPSに関連するタンパク質に特有の性質はあるのだろうか.本稿では,LLPS関連データベースの現状を把握するために,データベースの比較を行う.後半では,天然変性タンパク質の観点から,収録タンパク質の整理を試みる.
LLPS関連データベースは,2020年以降に公開された新しいものだ.基本的に文献検索により関連論文を取得し,キュレーターと呼ばれる専門家が論文を読み,注釈づけを行っている.また,生物種も特に限定することなく,真核生物から原核生物,ウイルスのデータまでを収録している.ただし,収録する情報やそのまとめ方は異なっている(表1).各データベースの特徴を以下に述べる.
表1 LLPSデータベースが収録する情報 | PhaSepDB | LLPSDB | PhaSePro | DrLLPS |
|---|
| エントリの単位 | 実験 | 実験/タンパク質 | タンパク質 | タンパク質 |
| エントリ数 | 961 | 2917 | 121 | 9285 |
| (150 scaffolds, 987 regulators, 8148 clients) |
| データベースへのリンク | UniProt, PubMed | UniProt, PubMed, OMIM, others | UniProt, PubMed | UniProt, Ensembl, PubMed |
| 局在するMLO | “MLO” | — | “Organelle” | “Condensate” |
| 液滴の種類 | “material_state” | “Morphology” | “Droplet type” | — |
| ロケーション | “location” | “localization” | — | * |
| 修飾 | “PTM” | “PTM” | “PTM affecting LLPS” | * |
| タンパク質構造 | “domain”, “repeat”, “oligomerization” | “protein structure type”, “IDR”, “LCR”, “Repeat” | — | * |
| タンパク質の領域 | “region” | “sequence_length” | “protein_region_mediating_LLPS” | — |
| LLPS diagram | あり | あり | — | — |
| 実験情報 | “cell_line_tissue”, “experiment(vivo/vitroや実験手法)”, “mutation”, “mutation note” | “Fusion”, “Cleaved”, “Mutation”, “Nucleic acid”, “Solute concentration”, “Salt concentration”, “Buffer”, “Crowding agent”, “Temperature”, “Pressure”, “Incubation time”, “other molecules”, “Detection method” | “Experimental techniqes”, “in vivo”, “in vitro”, “Experimental observations supporting the liquid material state” | — |
| 相互作用 | “partner”, “partner_uniprot”, “RNA”, “other” | — | “binding partners”, “Type of RNA(s) required”, “Molecular interaction types contributing to LLPS” | * |
| 疾患 | — | — | “Disease mutation” | * |
| 特徴 | PS-self, PS-otherの分類と予測器へのリンク | 詳細な実験情報 | 統制語による分類 | ホモログ情報,他のデータベース情報が豊富 |
| 二重引用符で囲まれた語は,データベース中での用語を表す.局在するMLO(membrane-less organelle)はnucleolusやP-bodyといったタンパク質が局在している膜のないオルガネラ,液滴の種類は“liquid”,“hydrogel”といった液滴の状態.PhaSepDB, LLPSDBのタンパク質領域は実験で使用された領域.*は別ファイルとしてダウンロード可能. |
1)PhaSepDB(http://db.phasep.pro/)
PhaSepDB4)は北京大学のグループにより運営されている.各エントリは実験単位でまとめられており,593タンパク質の情報からなる961の相分離(phase separation:PS)エントリが収録されている.これらのタンパク質は,単独で液滴を形成する“PS-self”と,他の分子の共存在下でPSを起こす,または細胞中でPSを起こす“PS-other”に分類されている.各エントリは文献を精読し人手をかけて注釈づけされた“reviewed”と,それ以外の“unreviewed”に二分されている.また,膜のないオルガネラ(membrane-less organelle:MLO)への局在情報も別カテゴリのエントリとして提供されている.UniProtの情報に基づくものを“UniProt reviewed”,ハイスループット実験(organelle purification, proximity labeling, immunofluorescence image-based screen, affinity purificationなど)により同定されたものを“High Throughput”としている.各エントリ情報はエクセルファイルとしてダウンロードできる.
2)LLPSDB(http://bio-comp.org.cn/llpsdb/home.html)
LLPSDB5)は中国科学院大学で運営されているデータベースで,2917エントリが収録されている.エントリはタンパク質を単位にしたものと,実験を単位にしたものがある.各エントリはまず“unambiguous”と“ambiguous”に分けられる.“unambiguous”はLLPSを起こす構成成分がすべて既知の場合,“ambiguous”は不確かな場合である.後者は,たとえばあるタンパク質がnucleosomeでLLPSを起こす場合,そこでの構成成分が確定できないという意味である.さらに“unambiguous”は,構成成分により“Protein(s)”,“Protein(s)+RNA”,“Protein(s)+DNA”に,タンパク質の種類により“natural”と“designed”に分類されている.このデータベースの最大の特徴は,一つの論文であっても実験ごとにエントリを分け,詳細な実験情報を収録している点である.また,“no Phase Separation”とうカテゴリがあり,ネガティブデータも明示的に検索できる.公開データはダウンロード可能で,分類基準ごとにエクセルファイルとして提供されている.
3)PhaSePro(https://phasepro.elte.hu/)
PhaSePro6)はLLPSを駆動するタンパク質を収録したデータベースである.ハンガリーのグループにより運営されている.公開中のデータは121エントリで317件の文献から抽出されている.各エントリはタンパク質単位でまとめられており,LLPSを駆動する領域144個の情報も含まれている.また,LLPSを駆動するというには実験情報が少し足りないタンパク質を“candidates”として公開している.PhaSeProの提供するデータで特徴的なのは,各エントリを統制語(controlled vocabulary)を用いて分類している点である.分類項目は,MLOの機能(“protective storage/reservoir”,“activation/nucleation/signal amplification/bioreactor”など8項目),LLPSを形成する相互作用(“electrostatic interaction”,“multivalent domain-motif interactions”など19項目),LLPSに関連する現象・要因(“partner dependent”,“PTM dependent”など6項目),凝集体(condensate)の流動性に関する実験的証拠(“dynamic movement/reorganization of molecules within the droplet”,“morphological traits”など7項目)である.
4)DrLLPS(http://llps.biocuckoo.cn/)
DrLLPS7)は,華中科技大学により運営されているデータベースで,LLPSに関連するタンパク質を収録している.他のデータベースに比べ収録数が多く9285件に上る.さらに,これらの相同タンパク質も合わせて437,887件の情報を提供している.膨大な情報は,人手を介した収集に加えコンピュータによる検索を駆使しているためと思われる.各タンパク質は,液滴中での機能によってScaffold(150タンパク質),Regulator(987タンパク質),Client(8148タンパク質)の三つに分類されている.Scaffoldとは液滴形成を駆動するタンパク質,Regulatorは液滴の形成,分散を制御する因子(修飾酵素など)であり,ClientはScaffoldと複合体を形成し機能するタンパク質,またはMLOに共局在するタンパク質とされている.公開データはエクセルファイルとしてダウンロード可能だが,含まれるデータはタンパク質の他のデータベースへのリンク情報およびMLOの種類,Scaffold, Regulator, Clientの区別のみである.その他,さまざまな情報は別ファイルとしてダウンロード可能であり,ウェブページではこれらに加えコンピュータによる解析結果などが閲覧できる.
5)LLPS関連データベースを比較する
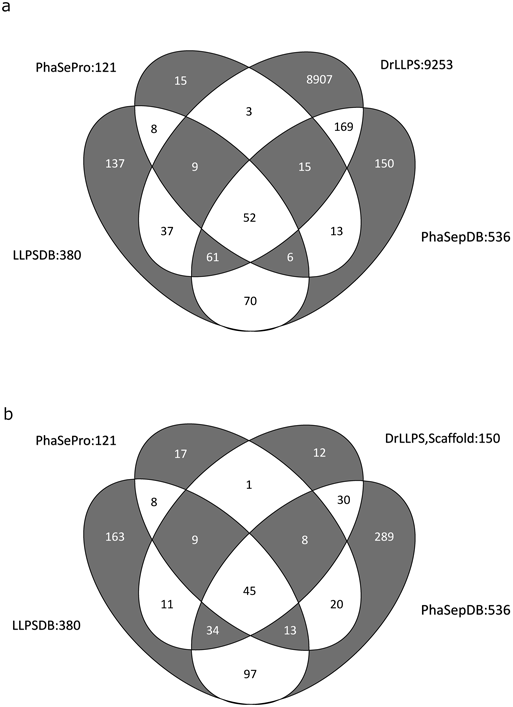
これら四つのデータベース間で,収録されたタンパク質はどの程度重複しているのだろうか? 図1aに,収録されているタンパク質をベン図としてまとめた.各データベースとも,独自のタンパク質がかなりの割合を占めている.PhaSepDBは150でデータベース登録数の28%,LLPSDBは137で36%,PhaSeProは15で10%弱,DrLLPSでは8907と96%に上る.先にも示したように,DrLLPSは収録タンパク質をScaffold, Regulator, Clientに分類している.この中でLLPS現象を駆動するとされるScaffoldタンパク質は150個である.図1bは,DrLLPSのタンパク質をScaffoldに限定したものである.この場合,DrLLPSにのみ収録されているのは12個なので(全体の8%),DrLLPS独自のエントリの多くはRegulator, Clientである.
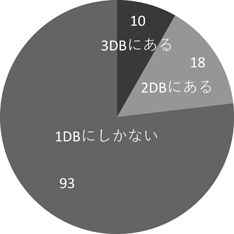
PhaSepDB, PhaSePro, DrLLPSには各タンパク質が局在するMLOが記載されている.これらの収録状況をMLOの表記に基づき調査した(図2).三つのデータベースにはのべ121種類のMLOが収録されており,そのうちの77%に当たる93個は一つのデータベースだけにみられた.また18種のMLOは二つのデータベースに見つかり,10種はすべてのデータベースに収録されていた.表記が同一のMLOを同じMLOとみなしたので,この10種のMLOはつづりが同じMLO(たとえば“Nucleolus”という単語)が三つのデータベースで用いられている,という意味である.表2に,二つおよび三つのデータベースに収録されているMLOと,そのMLOに局在するタンパク質の数をまとめた.三つすべてのデータベースでみられるMLOは核小体(Nucleolus)などよく知られているものが多いが,収録されているタンパク質数はデータベースごとに異なっている.ここで注意すべきは,同一タンパク質であっても異なるMLOが記載されることがあるという点だ.たとえば,RIMS-binding protein 2(Rimbp2;UniProt, Q9JIR1)はPhaSepDBでは“Presynaptic active zone condensate”に局在すると注釈づけされているが,PhaSeProでは“cytoplasmic protein granule”となっている.また,Spechle-type POZ protein(SPOP;UniProt, O43791)はPhaSeProでは“nuclear body”,“nuclear protein granule”,“SPOP/DAXX body”と三つのMLOが記されているが,PhaSepDBでは“Nuclear speckle”とされている.このMLOの食い違いからもわかるように,四つのデータベースは共通の用語やルールに基づいて注釈づけされているわけではない.表2のタンパク質の数のばらつきは用語や注釈づけルールの不一致による部分も多いだろう.
表2 LLPSデータベースに収録された膜のないオルガネラ(MLO)| MLO | Location | PhaSepDB | DrLLPS | PhaSePro |
|---|
| 三つのデータベースから見つかるMLO |
| Nucleolus | Nucleus | 34 | 36 | 5 |
| Nuclear speckle | Nucleus | 21 | 8 | 3 |
| PML nuclear body | Nucleus | 9 | 9 | 1 |
| Paraspeckle | Nucleus | 7 | 6 | 3 |
| Polycomb body | Nucleus | 5 | 1 | 1 |
| Postsynaptic density | Cytoplasm | 32 | 20 | 2 |
| P-body | Cytoplasm | 24 | 29 | 9 |
| Pericentriolar matrix | Cytoplasm | 8 | 1 | 1 |
| P granule | — | 23 | 8 | 5 |
| Balbiani body | — | 2 | 1 | 2 |
| 二つのデータベースから見つかるMLO |
| Nuclear pore complex | Nucleus | 31 | 4 | |
| Nuclear body | Nucleus | 9 | | 16 |
| Heterochromatin | Nucleus | 6 | | 4 |
| DNA damage foci | Nucleus | 4 | 1 | |
| Cajal body | Nucleus | 1 | 5 | |
| Histone locus body | Nucleus | 1 | | |
| Sam68 nuclear body | Nucleus | | 2 | 2 |
| Stress granule | Cytoplasm | 109 | 54 | |
| Centrosome | Cytoplasm | 11 | 14 | |
| Neuronal granule | Cytoplasm | 11 | 2 | |
| p62 cluster | Cytoplasm | 9 | | 1 |
| Spindle apparatus | Cytoplasm | 7 | 2 | |
| Inclusion body | Cytoplasm | 5 | | 3 |
| Yb body | Cytoplasm | 4 | | 1 |
| Negri body | Cytoplasm | 2 | | 2 |
| Nuage | — | 3 | 1 | |
| miRISC | — | 2 | | 2 |
| Germ plasm | — | 1 | | 2 |
| MLOが局在するとされる細胞内の局在をPhaSepDBの情報をもとにLocationとして記した.数字は各MLOに登録されたタンパク質の数.“—”はアノテーションがなかったもの. |
3. LLPS関連データベースを天然変性タンパク質の観点から整理する
これまでにみてきたように,四つのLLPS関連データベースは異なる方針で編集されたものでそれぞれ特徴がある.また,注釈づけで利用する用語の統一などもされておらず,異なるデータベース間で情報の対応づけも難しい.しかし,これらのデータベースが公開されたことでLLPS関連タンパク質のデータへのアクセスが容易になり,利便性が格段に向上したことは確かである.以下ではデータベースに収録されたタンパク質を,天然変性タンパク質の観点から整理してみたい.
1)LLPS関連タンパク質と天然変性タンパク質
天然変性領域はアミノ酸配列から予測可能であり,現状,予測は実用に足る精度があると考えられている.そこで天然変性領域予測とLCR予測をもとに天然変性率,LCR率を見積もることができる,本稿では天然変性領域予測として著者らが開発したNeProc8),LCRの抽出にSeg9)を用いた結果を示す.
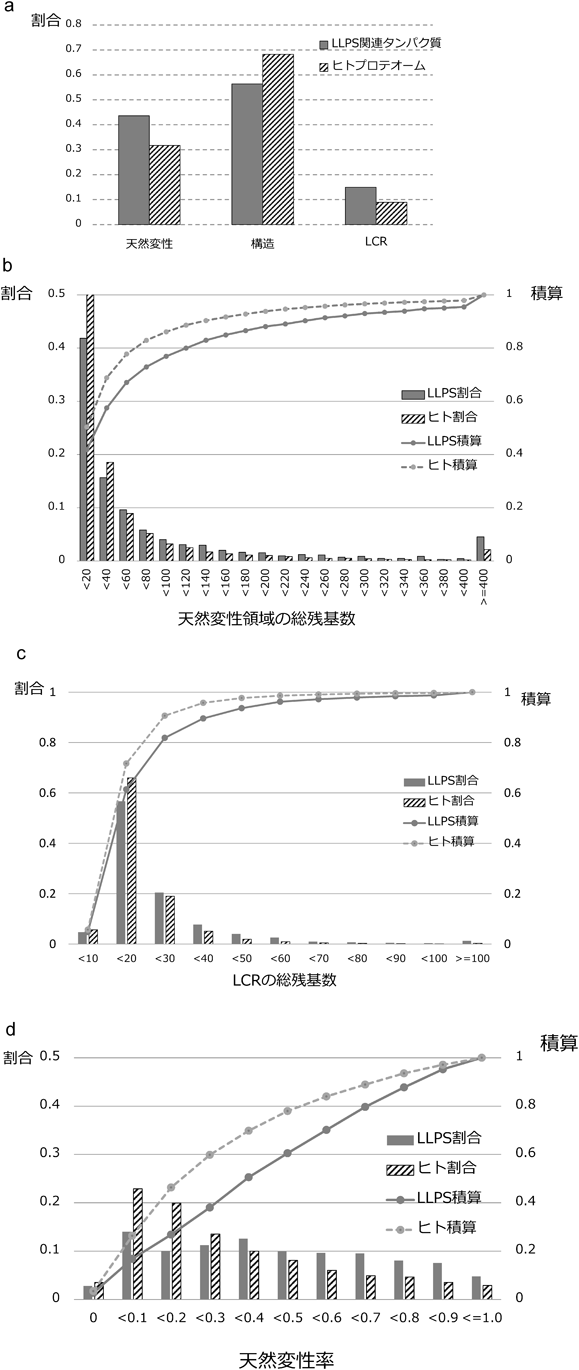
図3aにLLPS関連タンパク質とヒトプロテオームの天然変性率,およびLCR率を示す.LLPS関連タンパク質としてはデータベースのいずれかに収録されており,UniProtに登録されている天然のものを用いた.ただし,PhaSepDBに関しては“reviewed”のタンパク質,LLPSDBに関しては“unambiguous”かつ“natural”のタンパク質,DrLLPSに関してはScaffoldに限定した.これらの中には同じタンパク質だが生物種違いのものがあるので,アミノ酸配列の一致度90%でクラスタリングを行い,一つを代表として選んだ.このデータセットにはヒト以外の生物種由来のタンパク質も多く含まれているが,およそ6割は哺乳類由来である.よって比較対象データとしてヒトプロテオームを用いた.図3aからみてとれるように,天然変性率,LCR率ともLLPS関連タンパク質の方が高い.これは,LLPSに天然変性領域やLCRが関連しているという説を裏づけている.図3b, cは個々のタンパク質の天然変性領域およびLCRの長さの分布で,ここでもLLPS関連タンパク質が持つ天然変性領域およびLCRは長い傾向にある.特に天然変性領域の方が顕著で,ヒトプロテオームでは全体の8割が60残基以下の天然変性領域であるのに対して,LLPS関連タンパク質では積算が8割となる閾値は120残基である.さらに400残基以上の長大な天然変性領域も1割ほど含まれている.図3dは,各タンパク質の全長に対する天然変性領域率の分布である.この図からも,LLPS関連タンパク質の多くが天然変性タンパク質であることがわかる.率の低い部分ではヒトプロテームのタンパク質の方が割合が高いが,天然変性領域率が4割を超えてからは常にLLPS関連タンパク質の割合が高くなっている.
以上の結果からLLPS関連タンパク質は天然変性タンパク質が支配的であると確認できた.しかし,図3dをみると天然変性率が1割以下のタンパク質も数パーセント含まれている.これらのタンパク質は,天然変性タンパク質がなくてもLLPSを起こすのだろうか.PhaSepDBは収録タンパク質をPS-selfとPS-otherに分類している.天然変性率が5%以下でPhaSepDBに登録されているLLPS関連タンパク質は47個であった.そのうち,43個(91%)がPS-otherに分類されていた.すなわち,大多数が他の分子と共存することでLLPSを引き起こすものであった.共存する分子は,タンパク質が38例,タンパク質とRNAが4例,RNAが1例,その他の10例には記述がなかった.また,この43個のPS-otherタンパク質と共存するパートナータンパク質の天然変性率を調べてみると,平均が42.1%と図3aのLLPS関連タンパク質の平均にほぼ等しかった.すなわち,天然変性率が低い収録タンパク質の多くは,他の天然変性タンパク質との共存下で相分離を起こしていると考えられる.ただしデータベースには,構造ドメインのみでLLPSを起こす例も少数ながら収められていた10, 11).
2)LLPS関連タンパク質の天然変性領域の特徴
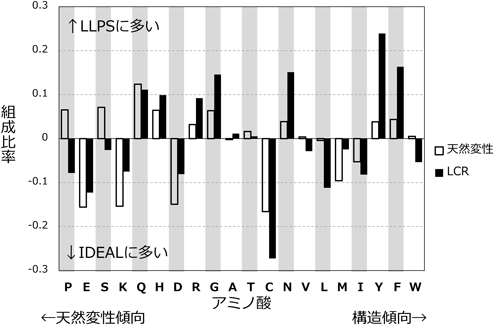
ではLLPSを駆動するタンパク質の天然変性領域には,他の天然変性領域にはない特徴があるのだろうか.まず,LLPS関連タンパク質の天然変性領域とLCRのアミノ酸組成を調べ,IDEALに収録された天然変性タンパク質と比較した(図4).LLPS関連タンパク質の方がIDEALに収録されたタンパク質より多く含むアミノ酸は,図4の縦軸がプラスの値をとる.また,横軸に置かれた各アミノ酸は,左に行くほど天然変性傾向が高く,右に行くほど構造傾向が高いように並べた12).興味深いのは,天然変性傾向が高い四つのアミノ酸(左から四つ)はLLPS関連タンパク質に少ない傾向があることだ.特にLCRでは4アミノ酸ともIDEALに収録されたタンパク質より少ない.さらに詳しくみると,プロリン,セリンはLLPSタンパク質の天然変性領域には多くみられるが,LCRには含まれていない.反対に,アルギニン,グリシン,アスパラギン,チロシン,フェニルアラニンといったアミノ酸は天然変性領域でも多いがLCRで顕著に多い.LLPSを駆動するLCRとしては,アルギニン・グリシンモチーフやチロシンリッチな領域13),芳香族アミノ酸の配列上のパターン14),非電荷極性アミノ酸に富むプリオン様ドメイン15)などが報告されている.またLLPSに寄与する相互作用様式として静電相互作用16),π–π相互作用17),π-カチオン相互作用18)などが知られている.図4の結果はこの事実を裏づけているようにみえる.以上のことから,LLPS関連タンパク質の天然変性領域は,これまで考えられてきた天然変性領域のアミノ酸組成とは少し異なっているように思われる.
LCRはLLPSに関連する天然変性領域中の機能部位と考えられるが,ProSとはどのような関係にあるのだろうか.IDEALに登録されているタンパク質について,ProSとLCRがどの程度重複するのかを調べてみた.IDEAL中のタンパク質のうちLCRと判定されたのは全残基の11.5%ほどであった.これらのタンパク質は9000残基弱のProS領域を含んでいるが,これらの領域のうちLCRと判断された領域は1000残基弱であり,全ProSの11.4%にあたる.仮にProSとLCRが似通ったものであるとすれば,ProSのLCR含有率は全残基のLCR含有率よりも高いはずである.しかし,ProSのLCR含有率はタンパク質全体の含有率とほぼ等しい.つまりProSとLCRは,天然変性領域中の相互作用部位という共通点はあるが,相関がない別ものだと考えられる.例外はあるが,ProSは一般に特定の相手と複合体構造をとり相互作用する.一方でLCRは,多価相互作用によりLLPSを駆動すると考えられている.多価相互作用とは,複数の結合部位を持つ一つの分子に複数の分子が結合する結合様式で,この相互作用により大きな構造体(液滴)が形成されると考えられている.ProSとLCRが異なる特徴を持つのは,相互作用様式の差に起因するのかもしれない.
3)LLPS関連タンパク質が持つ構造ドメイン
図3dの右端のデータが示すように天然変性タンパク質では全長にわたって天然変性領域であることはまれで,その多くは構造領域(ドメイン)と天然変性領域の両方からなっている.そこで,LLPS関連タンパク質はどのような構造ドメインを持つのかを,タンパク質構造分類データベースSCOP19, 20)をもとにまとめた(表3).一つのタンパク質が同じ構造ドメインを持つこともあるので,ドメイン数をタンパク質数で割ったものを平均リピート数とした.また,あるドメインを持つタンパク質のLLPSタンパク質データでの出現比率を,ヒトプロテオーム中での出現比率で割ったものを存在比とした.表3をみるとRNA-binding domain(RRM)はドメイン数,存在比で突出している.これは,タンパク質とRNAの混在によりLLPSが引き起こされる例が,現状では格段に多いことを表している.P-loop containing nucleoside triphosphate hydrolasesはタンパク質数,ドメイン数ともに多い.このドメインはヒトプロテーム中で3番目に多いドメインで,もともと多く存在するので上位にランクされているとも解釈できる.このドメインの存在比は約2なので,LLPSタンパク質データには2倍の率で含まれている.
表3 LLPSデータベースに収録されたタンパク質の持つ構造ドメイン| SCCS | ドメインネーム | タンパク質数 | ドメイン数 | 平均リピート数 | 平均天然変性率 | 存在比 |
|---|
| 15以上のタンパク質にみられるドメイン |
| c.37.1 | P-loop containing nucleoside triphosphate hydrolases | 65 | 83 | 1.28 | 0.21 | 1.91 |
| d.58.7 | RNA-binding domain, RBD, aka RNA recognition motif (RRM) | 64 | 127 | 1.98 | 0.55 | 9.18 |
| a.118.1 | ARM repeat | 25 | 33 | 1.32 | 0.32 | 2.34 |
| d.144.1 | Protein kinase-like (PK-like) | 21 | 21 | 1.00 | 0.26 | 1.00 |
| b.55.1 | PH domain-like | 17 | 17 | 1.00 | 0.44 | 1.23 |
| b.34.2 | SH3-domain | 16 | 27 | 1.69 | 0.43 | 3.64 |
| 20以上のドメインが見つかったもの |
| d.58.7 | RNA-binding domain, RBD, aka RNA recognition motif (RRM) | 64 | 127 | 1.98 | 0.55 | 9.18 |
| c.37.1 | P-loop containing nucleoside triphosphate hydrolases | 65 | 83 | 1.28 | 0.21 | 1.91 |
| g.3.11 | EGF/Laminin | 1 | 35 | 35.00 | 0.02 | 0.79 |
| a.118.1 | ARM repeat | 25 | 33 | 1.32 | 0.32 | 2.34 |
| b.34.2 | SH3-domain | 16 | 27 | 1.69 | 0.43 | 3.64 |
| b.36.1 | PDZ domain-like | 11 | 23 | 2.09 | 0.55 | 3.30 |
| d.15.1 | Ubiquitin-like | 14 | 22 | 1.57 | 0.33 | 3.10 |
| d.144.1 | Protein kinase-like (PK-like) | 21 | 21 | 1.00 | 0.26 | 1.00 |
| リピート率の高いドメイン |
| g.3.11 | EGF/Laminin | 1 | 35 | 35.00 | 0.02 | 0.79 |
| b.2.2 | Carbohydrate-binding domain | 1 | 10 | 10.00 | 0.01 | - |
| g.23.1 | TB module/8-cys domain | 1 | 9 | 9.00 | 0.02 | 4.87 |
| b.1.6 | Cadherin-like | 1 | 9 | 9.00 | 0.10 | 0.29 |
| f.14.1 | Voltage-gated ion channels | 1 | 4 | 4.00 | 0.41 | 0.48 |
| c.15.1 | BRCT domain | 3 | 11 | 3.67 | 0.70 | 6.38 |
| b.1.1 | Immunoglobulin | 5 | 17 | 3.40 | 0.42 | 0.19 |
| a.126.1 | Serum albumin-like | 1 | 3 | 3.00 | 0.04 | 5.22 |
| b.34.9 | Tudor/PWWP/MBT | 5 | 15 | 3.00 | 0.42 | 3.15 |
| g.39.1 | Glucocorticoid receptor-like (DNA-binding domain) | 3 | 9 | 3.00 | 0.78 | 1.24 |
| SCCSはSCOP concise classification stringで,データベースSCOP中でのタンパク質構造の分類コード.ドメインネームはSCOPのスーパーファミリー名,存在比“-”はヒトプロテオームには存在しないドメイン. |
LLPSのモデルとしてsticker–spacerモデルが提唱されている21).これはstickerと呼ばれる結合しやすい領域が繰り返した構造により,多価相互作用が起きるとするものである.SH3ドメインは表中,タンパク質数,ドメイン数とも多いが,これはSH3ドメインの繰り返し構造が,このsticker–spacerモデルの好例としてよく研究されていることによるのかもしれない.表中のドメインの平均リピート率は多くのタンパク質で1.5から2程度であり,各ドメインは一つのポリペプチド鎖中で繰り返し構造を持つようだ.LLPSに関しては天然変性領域に着目しがちだが,ここで述べたように構造ドメインにも特徴がありそうだ.
引用文献References
1) Fukuchi, S., Amemiya, T., Sakamoto, S., Nobe, Y., Hosoda, K., Kado, Y., Murakami, S.D., Koike, R., Hiroaki, H., & Ota, M. (2014) IDEAL in 2014 illustrates interaction networks composed of intrinsically disordered proteins and their binding partners. Nucleic Acids Res., 42(D1), D320–D325.
2) Kriwacki, R.W., Hengst, L., Tennant, L., Reed, S.I., & Wright, P.E. (1994) Structural studies of p21Waf1/Cip1/Sdi1 in the free and Cdk2-bound state: conformational disorder mediates binding diversity. Proc. Natl. Acad. Sci. USA, 93, 11504–11509.
3) Dyson, H.J. & Wright, P.E. (2005) Intrinsically unstructured proteins and their functions. Nat. Rev. Mol. Biol., 6, 197–208.
4) You, K., Huang, Q., Yu, C., Shen, B., Sevilla, C., Shi, M., Hermjakob, H., Chen, Y., & Li, T. (2020) PhaSepDB: a database of liquid–liquid phase separation related proteins. Nucleic Acids Res., 48(D1), D354–D359.
5) Wang, X., Zhou, X., Yan, Q., et al. (2022). Bioinformatics, 1–5.
6) Mészáros, B., Erodös, G., Szabó, B., et al. (2020) PhaSePro: the database of proteins driving liquid–liquid phase separation. Nucleic Acids Res., 48(D1), D360–D367.
7) Ning, W., Guo, Y., Lin, S., Mei, B., Wu, Y., Jiang, P., Tan, X., Zhang, W., Chen, G., Peng, D., et al. (2020) DrLLPS: a data resource of liquid–liquid phase separation in eukaryotes. Nucleic Acids Res., 48(D1), D288–D295.
8) Anbo, H., Amagai, H., & Fukuchi, S. (2020) NeProc predicts binding segments in intrinsically disordered regions without learning binding region sequences. Biophys. Physicobiol., 17, 147–154.
9) Wootton, J.C. & Federhen, S. (1993) Statistics of local complexity in amino acid sequences and sequence databases. Comput. Chem., 17, 149–163.
10) Wang, H., Yan, H., Aigner, H., Bracher, A., Nguyen, N.D., Hee, W.Y., Long, B.M., Price, G.D., Hartl, F.U., & Hayer-Hartl, M. (2019) Rubisco condensate formation by CcmM in β-carboxysome biogenesis. Nature, 566, 131–135.
11) Ladouceur, A.-M., Prmar, B.S., Biedzinski, S., et al. (2020) Clusters of bacterial RNA polymerase are biomolecular condensates that assemble through liquid-liquid phase separation. Proc. Natl. Acad. Sci. USA, 117, 18540–18549.
12) Campen, A., Williams, R.M., Brown, C.J., Meng, J., Uversky, V.N., & Dunker, A.K. (2008) TOP-IDP-scale: a new amino acid scale measuring propensity for intrinsic disorder. Protein Pept. Lett., 15, 956–963.
13) Kato, M., Han, T.W., Xie, S., Shi, K., Du, X., Wu, L.C., Mirzaei, H., Goldsmith, E.J., Longgood, J., Pei, J., et al. (2012) Cell-free formation of RNA granules: low complexity sequence domains form dynamic fibers within hydrogels. Cell, 11, 753–767.
14) Martin, E.W., Holehouse, A.S., Peran, I., Farag, M., Incicco, J.J., Bremer, A., Grace, C.R., Soranno, A., Pappu, R.V., & Mittag, T. (2020) Valence and patterning of aromatic residues determine the phase behavior of prion-like domains. Science, 367, 694–699.
15) Toombs, J.A., McCarty, B.R., & Ross, E.D. (2010) Compositional determinants of prion formation in yeast. Mol. Cell. Biol., 30, 319–332.
16) Nott, T.J., Petsalaki, E., Farber, P., Jervis, D., Fussner, E., Plochowietz, A., Craggs, T.D., Bazett-Jones, D.P., Pawson, T., Forman-Kay, J.D., et al. (2015) Phase transition of a disordered nuage protein generates environmentally responsive membraneless organelles. Mol. Cell, 57, 936–947.
17) Lin, Y., Currie, S.L., & Rosen, M.K. (2017) Intrinsically disordered sequences enable modulation of protein phase separation through distributed tyrosine motifs. J. Biol. Chem., 292, 19110–19120.
18) Wang, J., Choi, J.-M., Holehouse, A.S., Lee, H.O., Zhang, X., Jahnel, M., Maharana, S., Lemaitre, R., Pozniakovsky, A., Drechsel, D., et al. (2018) A Molecular Grammar Governing the Driving Forces for Phase Separation of Prion-like RNA Binding Proteins. Cell, 174, 688–699.
19) Andreeva, A., Kulesha, E., Gough, J., & Murzin, A.G. (2020) The SCOP database in 2020: expanded classification of representative family and superfamily domains of known protein structures. Nucleic Acids Res., 48(D1), D376–D382.
20) Chandonia, J.M., Guan, L., Yu, C., et al. (2022) SCOPe: improvements to the structural classification of proteins-extended database to facilitate variant interpretation and machine learning. Nucleic Acids Res., 50(D1), D553–D559.
21) Harmon, T.S., Holehouse, A.S., Rosen, M.K., & Pappu, R.V. (2017) Intrinsically disordered linkers determine the interplay between phase separation and gelation in multivalent proteins. eLife, 6, e30294.