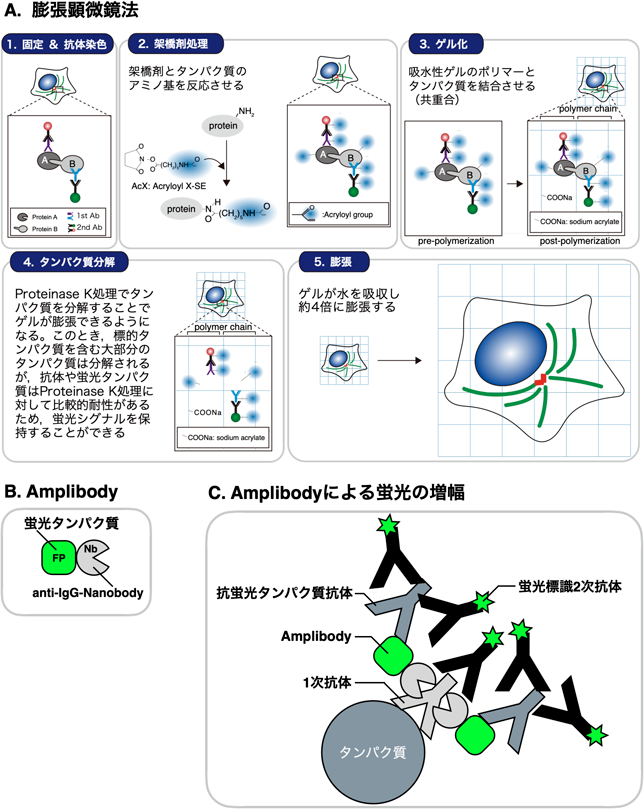
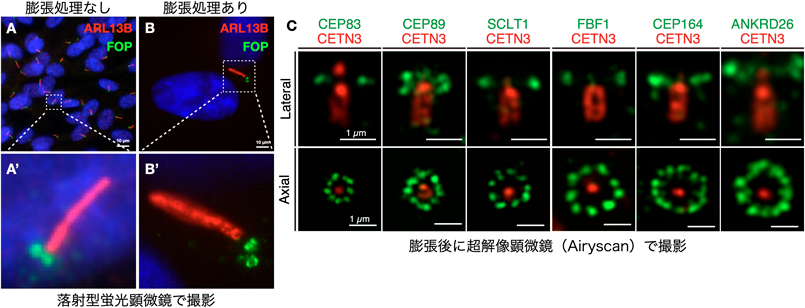
膨張顕微鏡法と蛍光増幅抗体(Amplibody)を用いた一次繊毛の超解像イメージングSuperresolution imaging of primary cilia by expansion microscopy using an amplibody for fluorescence signal amplification
1 京都大学大学院薬学研究科生体情報制御学分野Department of Physiological Chemistry, Graduate School of Pharmaceutical Sciences, Kyoto University ◇ 〒606–8501 京都市左京区吉田下阿達町46–29 ◇ 46–29 Yoshida-Shimoadachi-cho, Sakyo-ku, Kyoto 606–8501, Japan
2 東北大学大学院生命科学研究科分子細胞生物分野Laboratory of Molecular and Cellular Biology, Department of Biomolecular Sciences, Graduate School of Life Sciences, Tohoku University ◇ 〒980–8578 宮城県仙台市青葉区荒巻字青葉6–3 ◇ 6–3 Aoba, Aramaki, Aoba-ku, Sendai, Miyagi 980–8578, Japan