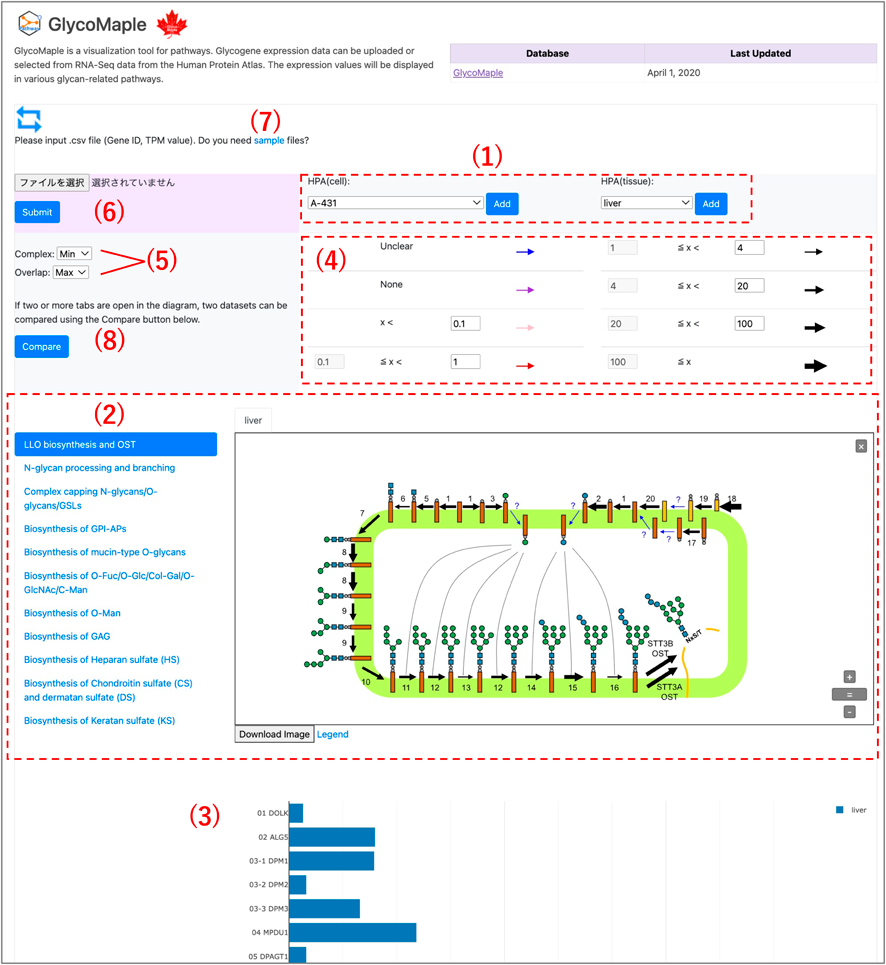
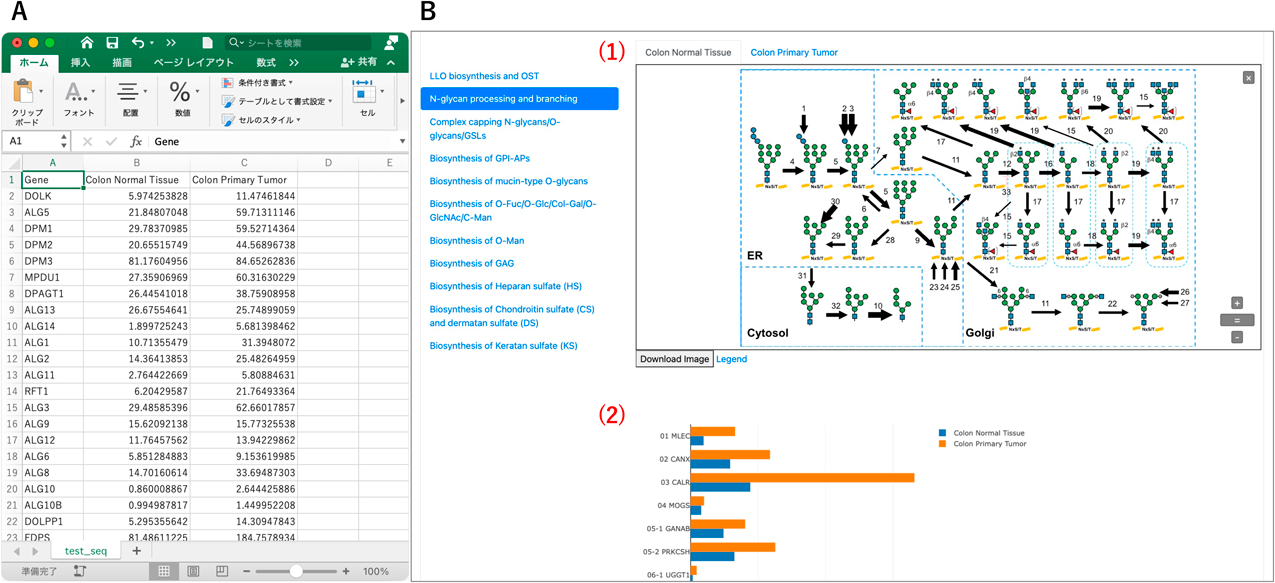
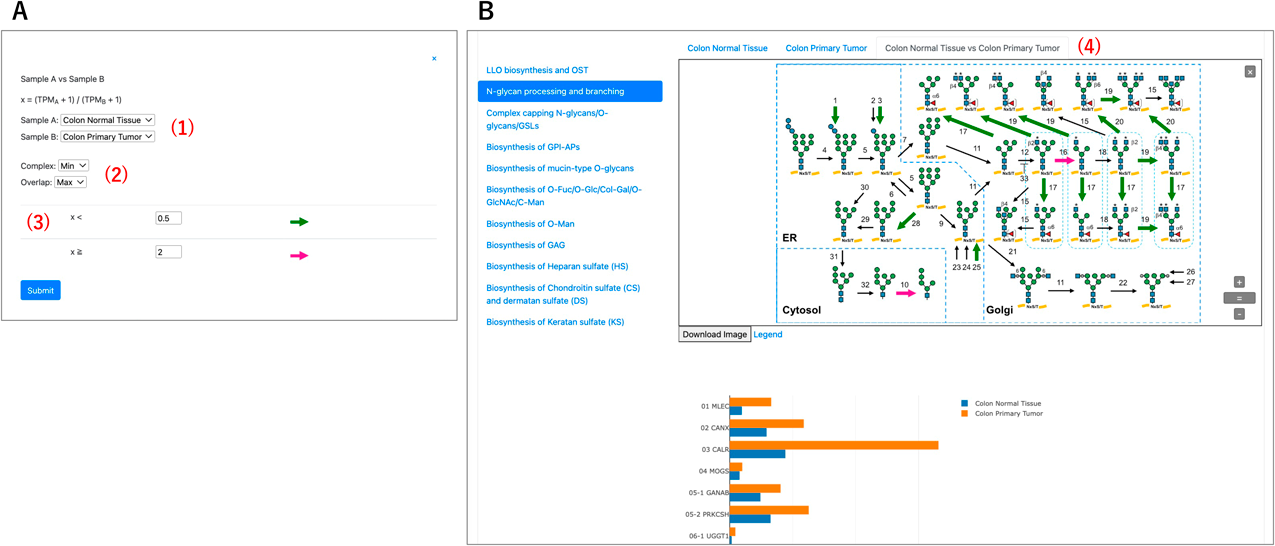
糖鎖遺伝子発現情報からの糖鎖構造推定ツールGlycoMapleの開発Development of GlycoMaple Tool estimating glycan structures from glycogene expression data
1 創価大学糖鎖生命システム融合研究所Glycan & Life Systems Integration Center (GaLSIC), Soka University ◇ 〒192–8577 東京都八王子市丹木町1–236 ◇ 1–236 Tangi-machi, Hachioji City, Tokyo, 192–8577, Japan
2 岐阜大学糖鎖生命コア研究所Institute for Glyco-core Research (iGCORE), Gifu University ◇ 〒501–1193 岐阜県岐阜市柳戸1–1 ◇ 1–1 Yanagido, Gifu 501–1193, Japan