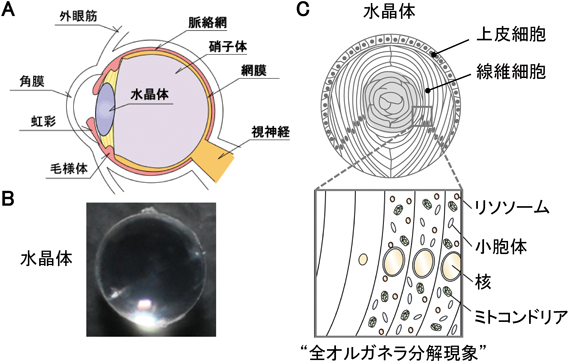
真核細胞の細胞内には,核,小胞体,ミトコンドリア,リソソームなどのさまざまなオルガネラ(細胞小器官)が存在している.これらのオルガネラはリン脂質を主成分とする脂質二重層で取り囲まれており,各オルガネラは細胞機能に必須な役割を果たしている.しかし,ある種の細胞ではその最終分化過程において,細胞内のすべてのオルガネラが分解あるいは除去されることが知られている.たとえば哺乳動物では,水晶体細胞,角化細胞(マウスでは皮膚,爪,毛髪,舌,食道,前胃などに存在する),赤血球の3種類の細胞の最終分化過程において,核,小胞体,ミトコンドリアなどのすべてのオルガネラが分解される(赤血球の核は脱核により細胞外に放出される)1–4).これらの細胞の細胞膜はオルガネラ除去後も残存しているため,個体内に一定期間(水晶体細胞は一生,角化細胞は数日~数週間,赤血球は約120日)留まることができる5).角化細胞の最終分化過程は角化(cornification),ケラチン化(keratinization),コルネオトーシス(corneoptosis)などと呼ばれることがあり,細胞死と関連した現象として考えられている3, 5–7).
全オルガネラ分解現象は赤血球や角化細胞ではすべての脊椎動物でみられるわけではないが,水晶体では脊椎動物で高度に保存されている8–10).筆者らはこの全オルガネラ分解現象の分子機構や生理的意義を解明するため水晶体をモデルとして,10年以上にわたり解析を進めてきた.本稿では,最近筆者らが報告したPLAATホスホリパーゼによるオートファジー非依存的全オルガネラ分解機構11)についての知見を中心に,水晶体における全オルガネラ分解現象の分子機構,生理的意義,未解決課題について議論する.
全オルガネラ分解現象については,これまで核DNAの分解機構を中心に研究が行われてきた.特にブレークスルーとなったのは,核DNAの分解を担うDNA分解酵素の同定である.長田重一博士(現・大阪大学)のグループは後述するように,マウスの水晶体の核DNAの分解にはリソソーム内に存在するDNASE2B/DNaseIIβ/DLAD(DNase II-like acid DNase)が必要であることや12, 13),赤血球の核DNAは脱核を経た後,マクロファージによって貪食されてリソソーム内のDNASE2/DNaseIIαによって分解されること,さらに各DNA分解酵素の生理機能を明らかにした14, 15).角化細胞の核DNA分解に必要なDNA分解酵素については,他のグループによってDNASE1L216–18),DNASE218, 19),TREX220),CAD(caspase-activated DNase)21)などの関与が報告されている.
一方,核DNA以外のミトコンドリア,小胞体,リソソームなどのオルガネラの分解の分子機構や生理的意義については,これまでほとんど不明であった.たとえばオートファジーの関与については,赤血球のミトコンドリア分解にはNIX/BNIP3L依存的マイトファジーやオールターナティブオートファジー(ULK1, FIP200, VPS34, ATG9Aなどには依存,ATG5やATG7には非依存)が関与することが報告されているが22–26),他のオルガネラ分解機構の関与は不明であった.さらに後述するように,水晶体や角化細胞の全オルガネラ分解現象は通常のオートファジーによらない未知のオルガネラ分解機構によることが示唆されていた27–31).全オルガネラ分解現象の生理的意義については,オルガネラ分解によって細胞内環境を均一化,最適化することで,水晶体では透明性や屈折率の獲得1, 11, 12, 32),皮膚や毛髪では機械的ストレスに対する抵抗性の獲得3, 4, 16),赤血球では貧血の抑制10, 23)などに貢献していると考えられてきたが,詳細な生理的意義は不明であった.このように全オルガネラ分解現象の分子機構や生理的意義については,生体内での解析が困難であるという問題もあり,細胞生物学や発生学の分野の重要課題として長らく未解明のまま残されていた.
1)水晶体の構造
水晶体は透明な無血管・無神経組織であり,主な機能は光を透過・屈折させ網膜に結像させることである.水晶体はその前面を覆う単層の上皮細胞と,上皮細胞が分化した線維細胞(水晶体の大部分を占める細長い細胞)の2種類の細胞から構成される(図1)33).上皮細胞から線維細胞への分化は一生にわたって認められるが,分化速度は加齢に伴い著しく低下する34, 35).上皮細胞は水晶体の赤道面で分化を開始し,新しく形成された線維細胞は既存の線維細胞の表面に追加される.その結果,線維細胞の年齢は,その位置から推測することができる.すなわち,発生初期に形成された最も古い線維細胞は水晶体の中心部に位置する一方で,最も新しく形成された線維細胞は表面近くに位置する.このように水晶体は細胞分化の全段階を一度に観察できるため,発生・分化の研究に適したモデルとして古くから用いられてきた.またイモリなどの一部の生物の水晶体は摘出後も虹彩色素上皮細胞から再生するため,分化転換や再生の研究対象としても用いられてきた36, 37).
2)水晶体の全オルガネラ分解現象の形態学的特徴
水晶体細胞の分化過程は非常にダイナミックであり,さまざまな形態変化や細胞内部の変容を伴う.分化の初期段階では,線維芽細胞増殖因子などの作用により光軸方向への細胞伸長が誘導される38).さらにクリスタリンと呼ばれる水晶体の透明化や屈折率の獲得に必須なタンパク質が多量に合成される.クリスタリンの細胞内濃度は水晶体の中心部ほど高濃度であり,最終的に水晶体の全タンパク質の90%を占める39).分化の最終段階では,核,ミトコンドリア,小胞体,ゴルジ体,リソソームなどのすべてのオルガネラが分解され,水晶体中心部にはオルガネラフリーゾーン(organelle-free zone)が形成される9).
水晶体の全オルガネラ分解現象が発見されたのは100年以上前であり,1898年にオーストリアの解剖学者Carl Rablがカナヘビの水晶体を観察する過程で,水晶体中心部の核は発生の過程で消失することを見いだした40–42).その後,1970年代以降の電子顕微鏡を用いた解析43, 44),1990年代以降の蛍光顕微鏡を用いた解析45–49)などにより,核DNAだけでなく核膜,ミトコンドリア,小胞体,ゴルジ体,リソソームを含むすべてのオルガネラが分解されることが明らかになった.さらに核分解時には核膜の断裂44, 49)や核ラミナの崩壊48, 50–52)が起こること,ミトコンドリアやゴルジ体は分解前に断片化すること46, 47, 53)なども明らかになった.しかし,これらの形態学的研究は,主に化学固定された水晶体や薄切された水晶体切片を用いていたことや,水晶体の全オルガネラ分解現象を再現できるin vitro分化誘導系やex vivo培養系が存在しなかったため,オルガネラ分解の詳細な過程はほとんど解明されていなかった.
3)ゼブラフィッシュを用いた全オルガネラ分解現象のライブイメージング
筆者らはライブイメージングや遺伝子改変が容易なゼブラフィッシュを用いることで,生体内における水晶体の全オルガネラ分解現象のリアルタイム可視化に初めて成功した11).方法としては,小胞体やミトコンドリアなどのオルガネラの内腔に局在化する可溶性の蛍光レポータータンパク質を発現するゼブラフィッシュを作製し,麻酔をかけた状態で受精後二日目(ゼブラフィッシュの水晶体では受精後二日目から核DNA分解が開始する54, 55))から水晶体をライブイメージング解析した.その結果,オルガネラ分解は水晶体中心部から同心円状に徐々に進行すること,リソソームと核の分解が起きた後にミトコンドリアと小胞体の分解が起きること,小胞体,ミトコンドリア,リソソームなどのオルガネラ分解時にはオルガネラの内腔タンパク質がサイトゾルに漏出すること(水晶体細胞の最終分化過程ではオルガネラ膜が崩壊する可能性を示唆)などが明らかになった11).このようにゼブラフィッシュを用いることでin vivoでのオルガネラ分解の過程の詳細な時間空間的情報を得ることが可能になった.
1)DNA分解酵素
水晶体線維細胞の最終分化過程では,核がTUNEL陽性となることや52),核DNAが断片化することが知られていたが56),DNA分解酵素は不明であった57).2003年に長田重一博士らのグループは,水晶体線維細胞に高発現するDNA分解酵素としてDNASE2Bを同定した12, 13).一方,アポトーシス時に核DNAの切断を担うCADや,マクロファージのリソソーム内において貪食した核DNAの分解を担うDNASE2は水晶体線維細胞に発現していなかった12).Dnase2b欠損マウスは水晶体細胞の核DNA分解抑制および白内障を認めたことから,DNASE2Bは核DNA分解および水晶体の透明性維持に必須であることが示された12).ゼブラフィッシュの水晶体ではDnase2bではなくDnase1l1lが高発現しており55, 58),dnase1l1l欠損ゼブラフィッシュは核DNAの分解抑制や白内障を認めた58).一方,DNASE2BやDnase1l1lは核膜,ミトコンドリア,小胞体などの分解には必要なかった12, 58).またDNASE2Bの遺伝子発現や核DNA分解には,熱ショック転写因子HSF4(heat shock transcription factor 4)59–61),Foxe3(Forkhead box protein E3)62),N-Myc(N-myc proto-oncogene protein)63),クロマチンリモデリング因子のSmarca4/Brg1/Snf2β64およびSmarca5/Snf2h65),RNA結合タンパク質Celf1(CUGBP Elav-like family member 1)66)などが必要であることも明らかになった.ゼブラフィッシュのDnase1l1lの遺伝子発現もHsf4依存的であった58).
2)DNA分解酵素のリソソームから核への移行機構
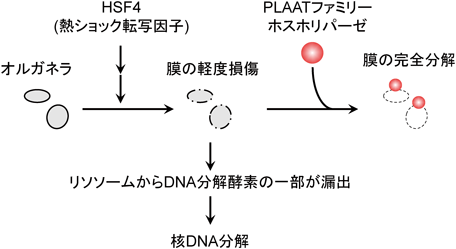
DNASE2Bは酸性条件で活性を持つDNA分解酵素であり12, 67),リソソームの内腔に存在する13, 48).ゼブラフィッシュのDnase1l1lもリソソームに存在する11).これまでリソソーム内のDNA分解酵素がどのような経路で核内に移行するかは不明であったが,最近筆者らは,核分解が始まる直前にリソソーム膜が部分的損傷を受けることで,DNA分解酵素がリソソーム内からサイトゾルへ漏出すること,リソソーム膜の部分的損傷にはHSF4が必要であることを見いだした11).したがって,HSF4はDNA分解酵素の遺伝子発現,リソソーム膜の部分的損傷という二つのステップで核DNA分解を制御していると考えられる.さらにCDK1(cyclin-dependent kinase 1)によるラミンA/Cのリン酸化および核膜の崩壊により,DNASE2Bの核内移行が促進されるという報告もある68–70).一方,ユビキチン・プロテアソーム系も核DNA分解を含めた水晶体細胞の正常な分化に必要という報告もあり,体細胞分裂の細胞周期を駆動するE3ユビキチンリガーゼAPC/C(anaphase promoting complex/cyclosome)が核DNA分解に関与することが示されているが54),実際に核DNA分解に関与するAPC/Cの基質は同定されていない.
5. 水晶体の全オルガネラ分解現象とオートファジー
1)オートファジーによるオルガネラ分解機構
オートファジーはタンパク質やオルガネラの分解を担う細胞内分解機構であり,Christian de Duveにより「細胞が自身の成分をリソソーム内で分解する現象」として1963年に定義された71, 72).その後の研究により,オートファジーはリソソームへの基質輸送経路の違いから複数の種類に分類されるようになった73, 74).一つ目はマクロオートファジー(通常「オートファジー」と呼ばれる)であり,オートファゴソームと呼ばれる二重膜のオルガネラに細胞質成分を取り込み,リソソームと融合することで内容物を分解する経路である75, 76).二つ目はミクロオートファジーであり,リソソーム膜の陥入により細胞成分を直接リソソーム内に取り込み分解する経路である77).多胞体あるいは後期エンドソームも膜タンパク質だけでなく細胞質成分も分解できることから,ミクロオートファジーの一種と考えられている78).三つ目はシャペロン介在性オートファジーであり,KFERQ様アミノ酸配列を有するタンパク質をリソソーム膜上のトランスポーターを介して直接的にリソソーム内に取り込み分解する経路である79).四つ目はクリノファジー(オルガネラ由来小胞分解機構とも呼ばれる)であり,分泌顆粒,ミトコンドリア由来小胞,小胞体などのオルガネラがリソソームと直接融合することで内容物を分解する経路である80–82).これらの経路のうち,マクロオートファジーはミトコンドリア,小胞体,ゴルジ体,リソソームなどのほぼすべてのオルガネラを選択的に分解できる.ミクロオートファジーやクリノファジーもある種の条件,細胞種ではオルガネラを分解できることが報告されている74).
2)オートファジーは水晶体の全オルガネラ分解に必要ない
筆者らはマウスやゼブラフィッシュの水晶体ではマクロオートファジーが恒常的に活性化していることを見いだしており27, 28, 83, 84),種々の遺伝子改変動物を用いて水晶体におけるマクロオートファジーの生理的意義を検討してきた.まずマクロオートファジーに必要なAtg5(autophagy-related 5)を全身で欠損させた新生仔マウスの水晶体を観察したところ,全オルガネラ分解現象は正常に認められた27).全身Atg5欠損マウスは脳神経系の異常や栄養不足により新生仔致死となるため85),次に水晶体特異的Atg5欠損マウスを作製したが,細胞内ホメオスタシスの破綻(ユビキチン化タンパク質やp62/SQSTM1などのマクロオートファジーの基質が蓄積)による加齢性白内障は認めたものの,全オルガネラ分解現象は正常に認められた28).さらに前述のAtg5非依存的オールターナティブオートファジー24)の関与を検証するため,水晶体特異的にVps3428)やFip200(筆者らの未発表データ)を欠損させたマウスや,全身でfip200を欠損させたゼブラフィッシュ11)を作製したが,全オルガネラ分解現象は正常に認められた.したがって,水晶体細胞の全オルガネラ分解現象はマクロオートファジー非依存的であることが明らかになった.他のグループから水晶体の線維細胞でマイトファジーが起きているとの報告もあるが86, 87),全オルガネラ分解時にマイトファジー活性は亢進しないとの報告があるため53),マイトファジーは全オルガネラ分解に貢献していないと考えられる.またニワトリの水晶体に対しマクロオートファジーを活性化する各種薬剤をex vivoで投与するとオルガネラ分解が早期に誘導されるという報告もあるが88, 89),生理的な条件で起きている全オルガネラ分解現象とは大きく異なると考えられる.
オートファジーのいずれの経路においてもオルガネラを分解するためにはリソソーム膜の連続性が保たれていることが必須と考えられる74).しかし前述のように,水晶体の全オルガネラ分解の過程ではリソソームの膜崩壊が他のオルガネラよりも早期に誘導されるため11),いずれのオートファジー経路が活性化したとしても,すべてのオルガネラをリソソームの内腔に取り込み効率的に分解することは困難と考えられる.
6. 全オルガネラ分解を担うPLAATホスホリパーゼの同定
1)ゼブラフィッシュを用いたin vivoノックアウトスクリーニング系の構築
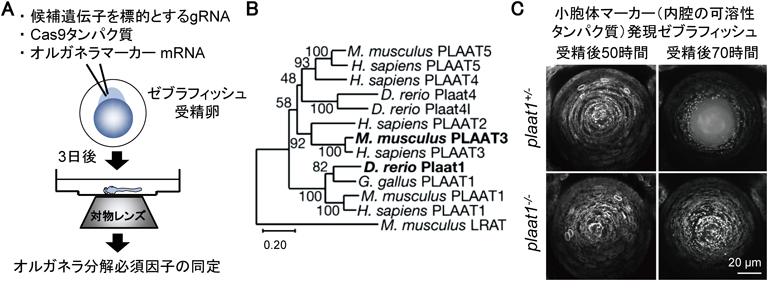
前述のようにマウスおよびゼブラフィッシュを用いた筆者らの研究から,水晶体の全オルガネラ分解現象はオートファジー非依存的であること27–29),そしてオルガネラ膜の崩壊を伴う現象であることが示唆された11).そこで筆者らは,オルガネラ内腔タンパク質の漏出の有無を指標とした,CRISPR/Cas9システムによるF0世代でのin vivoノックアウトスクリーニング系を構築した(図2A)11).ゼブラフィッシュはF0世代での遺伝子破壊が非常に容易であり,CRISPR/Cas9システムを用いることでわずか数日で簡易的にノックアウトすることができる90).具体的には,各候補遺伝子を標的とするgRNA,オルガネラマーカー(小胞体内腔およびミトコンドリアマトリックスに局在化する可溶性の蛍光タンパク質)をコードするmRNA, Cas9タンパク質をゼブラフィッシュの受精卵にインジェクションし,受精後3日目に水晶体のオルガネラをライブイメージング観察した(図2A).候補遺伝子群としては,トランスクリプトーム解析で同定されたゼブラフィッシュの水晶体に高発現する遺伝子群や,既知のオルガネラ分解に関連する遺伝子群などを選定した.100種類以上の遺伝子を解析した結果,ゼブラフィッシュの水晶体の全オルガネラ分解に必須な因子として,水晶体に高発現するPlaat1(phospholipase A/acyltransferase 1)を同定した11).
2)PLAATファミリータンパク質
Plaat1はPLAATファミリーに属する脂質代謝酵素であり,脊椎動物で高度に保存されている91–93).ヒトでは五つ(PLAAT1~5),マウスでは三つ(PLAAT1, 3, 5),ゼブラフィッシュでは三つ(Plaat1, 4, 4l)のパラログが存在する(図2B)11).PLAATの酵素学的特性は,2010年前後に宇山徹博士,上田夏生博士(香川大),Hei Sook Sul博士(カルフォルニア大)らのグループによって解明され,PLAATはホスファチジルコリン(PC),ホスファチジルエタノールアミン(PE)などのさまざまなグリセロリン脂質のsn-1位またはsn-2位からアシル基を切り出す加水分解反応を触媒するホスホリパーゼA1/A2(PLA1/A2)活性を有することが明らかになった94–99).さらにリゾリン脂質やPEがPLAATと共存すると,前者ではグリセロリン脂質から切り出したアシル基をリゾリン脂質の水酸基に転移してジアシル型グリセロリン脂質を生成するO-アシル転移酵素活性を,後者ではPEのアミノ基に転移してN-アシル-PEを生成するN-アシル転移酵素活性を有することも示されている94–99).PLAATはもともとがん抑制遺伝子として同定されていたが100–102),その後の研究により脂質代謝103, 104),ペルオキシソームの形成抑制92, 105–107),ウイルス感染108–110)などへの関与が明らかになった.しかし,水晶体やオルガネラ膜分解における役割は不明であった.
3)PLAATファミリータンパク質は水晶体の全オルガネラ分解に必要
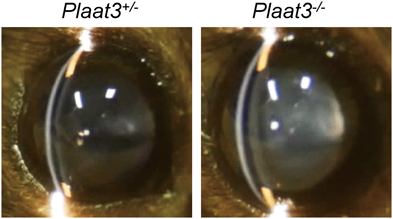
筆者らは全身でplaat1を欠損させたゼブラフィッシュを作製し表現型を詳細に解析したところ,plaat1欠損体では水晶体のサイズは変わらないが,ミトコンドリア,小胞体,リソソームなどのオルガネラの完全な分解が阻害されることを見いだした(図2C)11).plaat1欠損ゼブラフィッシュの表現型は活性中心に変異を持つPlaat1ではレスキューできなかったため,Plaat1の酵素活性がオルガネラ分解に必要であることも明らかになった11).一方マウスでは,PLAAT1, 3, 5のうちPLAAT3(別名:HRASLS3, PLA2G16, H-rev107, AdPLA)が水晶体に高発現していることが示唆されていた111).そこで全身性Plaat3欠損マウスを作製し水晶体を解析したところ,PLAAT3もPlaat1と同様にミトコンドリア,小胞体,リソソームなどの完全な分解に必須であることがわかった11).一方,ゼブラフィッシュ,マウスのいずれにおいてもPLAATファミリータンパク質は水晶体の核DNAの分解にはあまり重要でなかった11).その理由として,リソソーム膜はPLAAT非依存的に軽度損傷を受けるため,リソソーム内のDNA分解酵素の一部がサイトゾルに漏出するためであるという可能性が考えられた11).
7. PLAATを介する損傷オルガネラの選択的分解機構
1)PLAATの細胞内局在
Plaat1やPLAAT3はC末端近傍に膜貫通ドメインを有するテイルアンカー型タンパク質であるが91),通常の細胞では大部分はサイトゾルに存在する106, 107).水晶体においてもPlaat1やPLAAT3はサイトゾルに存在するが,オルガネラ分解直前にのみミトコンドリアやリソソームなどのさまざまなオルガネラに局在化することが明らかになった11).このオルガネラへの局在化には,膜貫通ドメインが必要かつ十分であった11).したがって,Plaat1やPLAAT3は膜貫通ドメインを介してオルガネラ膜の何らかの物理生化学的変化を感知している可能性が示唆された.
2)PLAATの膜局在化機構
PLAATホスホリパーゼは通常はサイトゾルに存在しているが,いくつかの条件ではそれらの一部がオルガネラに局在化することが報告されていた.たとえば,PLAAT3をHEK293細胞に過剰発現させると,PEX19(ペルオキシソーム膜タンパク質の輸送を担うシャペロン)と結合することでPLAAT3はペルオキシソームに移行する92, 106).一方,ピコルナウイルスを感染させたHeLa細胞では,PLAAT3はリソソームに局在化する108).ピコルナウイルスの感染時には,ウイルスがエンドサイトーシスされた後,ウイルス自身のタンパク質によりリソソーム膜にポアを形成することで,ウイルスゲノムをサイトゾルに移行させる112, 113).このとき,PLAAT3はそれらの損傷リソソームに移行し,ウイルスゲノムのサイトゾルへの移行を促進する108).同様にHeLa細胞にL-leucyl-L-leucine methyl ester(LLOMe)を投与した際にも,膜損傷を受けたリソソーム膜にPLAAT3が移行することから108),膜損傷がPLAAT3の膜移行のトリガーになっている可能性が示唆されていた.しかし,リソソーム膜が損傷を受けると,膜タンパク質のユビキチン化,糖鎖の露出,他のサイトゾルタンパク質(ガレクチン,ESCRTなど)の局在化などのさまざまな変化が誘導されるため114, 115),膜損傷自体がPLAAT3の膜移行に十分かどうかは不明であった.
そこでPLAAT3の膜移行に膜損傷が十分かどうかを検証するため,リコンビナントPLAAT3とリポソームを用いたin vitro再構成実験を実施した結果,PLAAT3とリポソームを混合しただけでは膜局在しなかったが,ハチ毒由来のポア形成ペプチドであるメリチン116)を用いてリポソーム膜を損傷させるとPLAAT3が膜局在することを見いだした11).さらにサイトゾル成分を除去したセミインタクト細胞を用いた解析系においても,培養液に添加したリコンビナントPLAAT3はメリチン存在下においてのみ核膜,小胞体,ミトコンドリアなどのオルガネラに移行した11).一方,ミトコンドリア外膜透過性を亢進させるcBid(Caspase-8-cleaved Bid)を添加した場合は,リコンビナントPLAAT3はミトコンドリアのみに移行した11).生細胞にアポトーシスを誘導しミトコンドリア外膜の透過性を亢進させた場合も,外来性PLAAT3はミトコンドリアに移行した11).これらの結果から,PLAAT3の膜移行には膜損傷が十分であると考えられた(図3).実際にゼブラフィッシュやマウスの水晶体においても,リソソーム膜だけでなくミトコンドリア外膜もPLAAT非依存的に部分的損傷を受けること,Plaat1やPLAAT3は軽度膜損傷とほぼ同じタイミングで膜移行することも見いだした11).したがって,水晶体においても膜損傷がPlaat1やPLAAT3の膜局在化を誘導していると考えられた.
3)PLAATによる膜分解機構
PLAATは損傷膜に局在化した後,そのホスホリパーゼ活性により膜リン脂質を分解していると考えられる.その根拠として,前述のように水晶体のオルガネラ分解にはPLAATの酵素活性が必要であること,in vitro再構成アッセイにおいてリコンビナントPLAAT3は単離ミトコンドリアのリン脂質を加水分解できること,培養細胞にPLAAT3を過剰発現させるとペルオキシソームの膜が崩壊すること,ミトコンドリアに恒常的に局在するPLAAT3変異体(C末端欠損体)はミトコンドリア膜の破裂を引き起こすことなどがあげられる11).さらに細胞外に分泌されるヘビ毒のホスホリパーゼA2,ミツバチ毒のホスホリパーゼA2,IIA型分泌性ホスホリパーゼA2(sPLA2-IIA)などの分泌性ホスホリパーゼA2もリン脂質を加水分解することで生体膜(細菌の細胞膜やウイルス膜)を分解できることが報告されている93, 117).膜リン脂質の直接的な分解だけでなく,リゾリン脂質などの分解産物が膜を効率的に可溶化する天然界面活性剤として機能している可能性もある118).前述のようにPLAATはアシル転移酵素活性も有しているが,水晶体のオルガネラ分解の過程におけるアシル転移の有無は不明である.水晶体内でオルガネラ分解が起きている細胞群はごく少数であるため,今後,高感度かつシングルセルでのリピドミクス解析手法の開発が期待される.
8. 熱ショック転写因子HSF4による全オルガネラ分解の制御機構
水晶体の全オルガネラ分解は時間空間的に厳密に制御されており,水晶体の中心部から同心円状に進行するという特徴がある(図1)11).これまでにカルシウムや酸素分圧などの濃度勾配と全オルガネラ分解現象のタイミングとの相関は示唆されていたが,それらの因果関係は実証されておらず2),水晶体の中心部において全オルガネラ分解をトリガーする因子は不明であった.ヒト遺伝性白内障の原因遺伝子産物であるHSF4は,水晶体線維細胞の分化の後期に働く重要な転写因子であり59, 119, 120),これまでにHsf4欠損マウスやhsf4欠損ゼブラフィッシュの水晶体では,クリスタリンやDNA分解酵素などの遺伝子発現が低下するだけでなく,オルガネラの分解も抑制されることが報告されていた58–61, 121).しかし,HSF4によるオルガネラ分解の制御機構は不明であった.筆者らはゼブラフィッシュのHsf4の全オルガネラ分解現象への関与を検証した結果,Hsf4はPlaat1のオルガネラへの移行,リソソーム膜やミトコンドリア外膜の軽度損傷,オルガネラ(核DNA,ミトコンドリア,小胞体,リソソーム)の分解のいずれにも必要な因子であることを見いだした(図3)11).Hsf4は水晶体線維細胞の分化の後期段階において高発現する転写因子であることから61, 122–125),全オルガネラ分解現象が水晶体の中心から辺緑に向かって起こる理由は,Hsf4の発現および活性化の時間空間パターンによって説明できる可能性がある.Hsf4と軽度膜損傷とを結びつける因子は現時点では不明であるが,クリスタリンなどのタンパク質の高発現59, 60, 64),あるいはオルガネラ膜を構成する脂質やタンパク質などの生化学的変化が膜の恒常性を破綻させ,オルガネラ膜の損傷を引き起こしている可能性がある.
筆者らは,PLAATファミリータンパク質がオルガネラ膜だけでなく,マトリックスタンパク質や膜貫通タンパク質の分解にも重要であることを見いだした11).PLAATファミリータンパク質によって膜リン脂質が分解されることで,ユビキチン・プロテアソーム系126)などの他の細胞質分解系がこれらの非脂質性オルガネラ成分にアクセスしやすくなると推測される.他の脂質代謝酵素の関与は不明であるが,in vitro再構成系や赤血球においてオルガネラ分解との関連が示唆されていたALOX15(15-lipoxygenase)127, 128)については,水晶体線維細胞での発現が低いこと13),ゼブラフィッシュにおける15-lipoxygenaseの機能的ホモログAlox12129)は水晶体のオルガネラ分解に必要ないこと11),Alox15欠損マウスの赤血球では明らかなオルガネラ分解の異常を認めないこと130–132),などの理由からそのオルガネラ分解への関与は低いものと考えられる.リソソーム内のプロテアーゼやリパーゼなどの加水分解酵素も,リソソーム内のDNA分解酵素と同様にサイトゾルに漏出した後にオルガネラ成分の分解に寄与している可能性はあるが,現時点では不明である.水晶体線維細胞にはCalpain 3や各種Caspaseなどのシステインプロテアーゼも高発現しているが133, 134),これらのオルガネラ分解への関連は十分に解析されていない.今後,水晶体の各オルガネラ成分の分解に関わる加水分解酵素やユビキチンリガーゼなどが同定されていくことで,全オルガネラ分解現象の分子機構の全体像の理解につながることが期待される.
PLAATファミリータンパク質による全オルガネラ分解の生理的意義を解析するため,生きた状態の野生型ゼブラフィッシュの水晶体中心部の光透過性を経時的に解析した結果,水晶体中心部のミトコンドリアや小胞体が分解される受精後60時間の前後において,光透過性の亢進を認めた11).一方,plaat1欠損ゼブラフィッシュの水晶体では,受精後60時間以降でも光透過性の亢進を認めず,成体でも水晶体の混濁および屈折率の異常を認めた11).同様に,Plaat3欠損マウスの水晶体も混濁および屈折率の低下を認めた(図4)11).したがって,PLAAT依存的全オルガネラ分解は,水晶体の透明化と適切な屈折率の獲得に必要であることが明らかになった.一般的にオルガネラとサイトゾルでは,オルガネラに脂質が多く含まれていることもあり,屈折率が異なる2).水晶体においてすべてのオルガネラが分解される理由は,オルガネラと周囲のサイトゾルとの屈折率の差を解消し,高濃度のクリスタリンによる透明性と高い屈折率を実現するためであると考えられる.
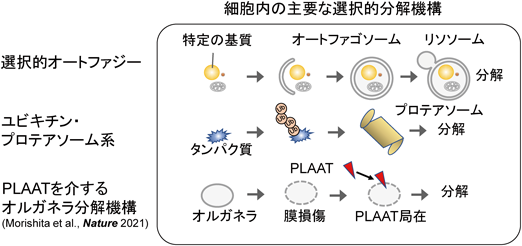
これまで水晶体における全オルガネラ分解機構については,核DNA分解機構以外はほとんど不明であった.今回,実行因子としてPLAATファミリータンパク質が同定され,さらにHSF4による制御機構が明らかになったことで,全オルガネラ分解現象についての統合的な理解が大きく進んだといえる.さらにPLAATファミリータンパク質による新規オルガネラ分解機構の発見により,これまでオートファジーのみによると考えられていたオルガネラ分解機構の多様性が明らかになった.またPLAAT依存的オルガネラ分解機構は損傷オルガネラを選択的に分解することから,今後,選択的オートファジー,ユビキチン・プロテアソーム系を含めた細胞内の選択的分解機構の包括的理解が進むことが期待される(図5).しかし,水晶体の全オルガネラ分解機構についてはいまだに多くの不明点が残っている.たとえば,HSF4依存的なオルガネラ膜の軽度損傷の分子メカニズム(クリスタリンやポア形成因子の関与),HSF4自体の転写制御機構(低酸素やカルシウム濃度などの環境因子の影響),PLAATファミリータンパク質による損傷膜の認識機構や膜分解の分子基盤,ユビキチン・プロテアソーム系との連携機構(ユビキチンリガーゼの同定),リソソーム由来加水分解酵素やCaspaseのオルガネラ分解への関与,核膜の崩壊あるいは分解の分子機構,細胞膜の保護機構などはほとんど不明である.さらに今回明らかになったPLAATファミリータンパク質による損傷オルガネラ分解現象が,水晶体以外の組織や細胞でも起きているかどうかは大変興味深い課題であり,今後PLAATファミリータンパク質の多重遺伝子改変動物やレポーター動物を用いた全身解析が待たれる.
謝辞Acknowledgments
本稿では,筆者が2009年に水島昇教授(当時・東京医科歯科大学,現・東京大学)の研究室で博士課程の大学院生として研究を始めてから2021年までの約12年間に得られた知見について概説しました.PLAATの同定までに8年を要しましたが,このような10年単位の研究を継続させていただいた水島昇教授,江口智也博士をはじめとした多くの共同研究者や同僚の方々,そして2019年に私が順天堂大学に異動した後も論文化にあたり寛大なご配慮をいただいた現所属先の小松雅明教授(順天堂大学)にこの場を借りて心から感謝申し上げます.
引用文献References
1) Nagata, S. (2005) DNA degradation in development and programmed cell death. Annu. Rev. Immunol., 23, 853–875.
2) Bassnett, S. (2009) On the mechanism of organelle degradation in the vertebrate lens. Exp. Eye Res., 88, 133–139.
3) Rogerson, C., Bergamaschi, D., & O’Shaughnessy, R.F.L. (2018) Uncovering mechanisms of nuclear degradation in keratinocytes: A paradigm for nuclear degradation in other tissues. Nucleus, 9, 56–64.
4) Eckhart, L., Lippens, S., Tschachler, E., & Declercq, W. (2013) Cell death by cornification. Biochim. Biophys. Acta Mol. Cell Res., 1833, 3471–3480.
5) Kroemer, G., Galluzzi, L., Vandenabeele, P., Abrams, J., Alnemri, E.S., Baehrecke, E.H., Blagosklonny, M.V., El-Deiry, W.S., Golstein, P., Green, D.R., et al. (2009) Classification of cell death: recommendations of the Nomenclature Committee on Cell Death 2009. Cell Death Differ., 16, 3–11.
6) Matsui, T., Kadono-Maekubo, N., Suzuki, Y., Furuichi, Y., Shiraga, K., Sasaki, H., Ishida, A., Takahashi, S., Okada, T., Toyooka, K., et al. (2021) A unique mode of keratinocyte death requires intracellular acidification. Proc. Natl. Acad. Sci. USA, 118, e2020722118.
7) Moore, J.L. & Greco, V. (2021) Functional cell death, corneoptosis, requires temporally controlled intracellular acidification. Proc. Natl. Acad. Sci. USA, 118, e2106633118.
8) Alibardi, L. (2016) The process of cornification evolved from the initial keratinization in the epidermis and epidermal derivatives of vertebrates: A new synthesis and the case of sauropsids. Int. Rev. Cell Mol. Biol., 327, 263–319.
9) Bassnett, S., Shi, Y., & Vrensen, G.F. (2011) Biological glass: structural determinants of eye lens transparency. Philos. Trans. R. Soc. Lond. B Biol. Sci., 366, 1250–1264.
10) Menon, V. & Ghaffari, S. (2021) Erythroid enucleation: a gateway into a “bloody” world. Exp. Hematol., 95, 13–22.
11) Morishita, H., Eguchi, T., Tsukamoto, S., Sakamaki, Y., Takahashi, S., Saito, C., Koyama-Honda, I., & Mizushima, N. (2021) Organelle degradation in the lens by PLAAT phospholipases. Nature, 592, 634–638.
12) Nishimoto, S., Kawane, K., Watanabe-Fukunaga, R., Fukuyama, H., Ohsawa, Y., Uchiyama, Y., Hashida, N., Ohguro, N., Tano, Y., Morimoto, T., et al. (2003) Nuclear cataract caused by a lack of DNA degradation in the mouse eye lens. Nature, 424, 1071–1074.
13) Nakahara, M., Nagasaka, A., Koike, M., Uchida, K., Kawane, K., Uchiyama, Y., & Nagata, S. (2007) Degradation of nuclear DNA by DNase II-like acid DNase in cortical fiber cells of mouse eye lens. FEBS J., 274, 3055–3064.
14) Yoshida, H., Kawane, K., Koike, M., Mori, Y., Uchiyama, Y., & Nagata, S. (2005) Phosphatidylserine-dependent engulfment by macrophages of nuclei from erythroid precursor cells. Nature, 437, 754–758.
15) Kawane, K., Fukuyama, H., Kondoh, G., Takeda, J., Ohsawa, Y., Uchiyama, Y., & Nagata, S. (2001) Requirement of DNase II for definitive erythropoiesis in the mouse fetal liver. Science, 292, 1546–1549.
16) Fischer, H., Szabo, S., Scherz, J., Jaeger, K., Rossiter, H., Buchberger, M., Ghannadan, M., Hermann, M., Theussl, H.C., Tobin, D.J., et al. (2011) Essential role of the keratinocyte-specific endonuclease DNase1L2 in the removal of nuclear DNA from hair and nails. J. Invest. Dermatol., 131, 1208–1215.
17) Jager, K., Fischer, H., Tschachler, E., & Eckhart, L. (2007) Terminal differentiation of nail matrix keratinocytes involves up-regulation of DNase1L2 but is independent of caspase-14 expression. Differentiation, 75, 939–946.
18) Fischer, H., Buchberger, M., Napirei, M., Tschachler, E., & Eckhart, L. (2017) Inactivation of DNase1L2 and DNase2 in keratinocytes suppresses DNA degradation during epidermal cornification and results in constitutive parakeratosis. Sci. Rep., 7, 6433.
19) Fischer, H., Fumicz, J., Rossiter, H., Napirei, M., Buchberger, M., Tschachler, E., & Eckhart, L. (2017) Holocrine secretion of sebum is a unique DNase2-dependent mode of programmed cell death. J. Invest. Dermatol., 137, 587–594.
20) Manils, J., Fischer, H., Climent, J., Casas, E., Garcia-Martinez, C., Bas, J., Sukseree, S., Vavouri, T., Ciruela, F., de Anta, J.M., et al. (2017) Double deficiency of Trex2 and DNase1L2 nucleases leads to accumulation of DNA in lingual cornifying keratinocytes without activating inflammatory responses. Sci. Rep., 7, 11902.
21) Yamamoto-Tanaka, M., Makino, T., Motoyama, A., Miyai, M., Tsuboi, R., & Hibino, T. (2014) Multiple pathways are involved in DNA degradation during keratinocyte terminal differentiation. Cell Death Dis., 5, e1181.
22) Novak, I., Kirkin, V., McEwan, D.G., Zhang, J., Wild, P., Rozenknop, A., Rogov, V., Löhr, F., Popovic, D., Occhipinti, A., et al. (2010) Nix is a selective autophagy receptor for mitochondrial clearance. EMBO Rep., 11, 45–51.
23) Sandoval, H., Thiagarajan, P., Dasgupta, S.K., Schumacher, A., Prchal, J.T., Chen, M., & Wang, J. (2008) Essential role for Nix in autophagic maturation of erythroid cells. Nature, 454, 232–235.
24) Nishida, Y., Arakawa, S., Fujitani, K., Yamaguchi, H., Mizuta, T., Kanaseki, T., Komatsu, M., Otsu, K., Tsujimoto, Y., & Shimizu, S. (2009) Discovery of Atg5/Atg7-independent alternative macroautophagy. Nature, 461, 654–658.
25) Honda, S., Arakawa, S., Nishida, Y., Yamaguchi, H., Ishii, E., & Shimizu, S. (2014) Ulk1-mediated Atg5-independent macroautophagy mediates elimination of mitochondria from embryonic reticulocytes. Nat. Commun., 5, 4004.
26) Kundu, M., Lindsten, T., Yang, C.Y., Wu, J., Zhao, F., Zhang, J., Selak, M.A., Ney, P.A., & Thompson, C.B. (2008) Ulk1 plays a critical role in the autophagic clearance of mitochondria and ribosomes during reticulocyte maturation. Blood, 112, 1493–1502.
27) Matsui, M., Yamamoto, A., Kuma, A., Ohsumi, Y., & Mizushima, N. (2006) Organelle degradation during the lens and erythroid differentiation is independent of autophagy. Biochem. Biophys. Res. Commun., 339, 485–489.
28) Morishita, H., Eguchi, S., Kimura, H., Sasaki, J., Sakamaki, Y., Robinson, M.L., Sasaki, T., & Mizushima, N. (2013) Deletion of autophagy-related 5 (Atg5) and Pik3c3 genes in the lens causes cataract independent of programmed organelle degradation. J. Biol. Chem., 288, 11436–11447.
29) Morishita, H. & Mizushima, N. (2016) Autophagy in the lens. Exp. Eye Res., 144, 22–28.
30) Rossiter, H., Konig, U., Barresi, C., Buchberger, M., Ghannadan, M., Zhang, C.F., Mlitz, V., Gmeiner, R., Sukseree, S., Födinger, D., et al. (2013) Epidermal keratinocytes form a functional skin barrier in the absence of Atg7 dependent autophagy. J. Dermatol. Sci., 71, 67–75.
31) Jaeger, K., Sukseree, S., Zhong, S., Phinney, B.S., Mlitz, V., Buchberger, M., Narzt, M.S., Gruber, F., Tschachler, E., Rice, R.H., et al. (2019) Cornification of nail keratinocytes requires autophagy for bulk degradation of intracellular proteins while sparing components of the cytoskeleton. Apoptosis, 24, 62–73.
32) Morishita, H., Eguchi, T., & Mizushima, N. (2021) A new insight into the lens: cytosolic PLAAT phospholipases degrade organelles to make the lens transparent. Autophagy, 17, 2645–2647.
33) Cvekl, A. & Ashery-Padan, R. (2014) The cellular and molecular mechanisms of vertebrate lens development. Development, 141, 4432–4447.
34) Bassnett, S. & Sikic, H. (2017) The lens growth process. Prog. Retin. Eye Res., 60, 181–200.
35) Hanna, C. & O’Brien, J.E. (1961) Cell production and migration in the epithelial layer of the lens. Arch. Ophthalmol., 66, 103–107.
36) Hayashi, T., Mizuno, N., & Kondoh, H. (2008) Determinative roles of FGF and Wnt signals in iris-derived lens regeneration in newt eye. Dev. Growth Differ., 50, 279–287.
37) Eguchi, G. (1988) Cellular and molecular background of wolffian lens regeneration. Cell Differ. Dev., 25(Suppl), 147–158.
38) Collins, T.N., Mao, Y., Li, H., Bouaziz, M., Hong, A., Feng, G.S., Wang, F., Quilliam, L.A., Chen, L., Park, T., et al. (2018) Crk proteins transduce FGF signaling to promote lens fiber cell elongation. eLife, 7, e32586.
39) Sharma, K.K. & Santhoshkumar, P. (2009) Lens aging: Effects of crystallins. Biochim. Biophys. Acta, Gen. Subj., 1790, 1095–1108.
40) Rabl, C. (1899) Uber den bau und die entwicklung der linse: III Teil:die lines der saugethiere: Ruckblick und Schluss. Z. Wiss. Zool., 67, 1–138.
41) Wride, M.A. (2011) Lens fibre cell differentiation and organelle loss: many paths lead to clarity. Philos. Trans. R. Soc. Lond. B Biol. Sci., 366, 1219–1233.
42) Bassnett, S. (2002) Lens organelle degradation. Exp. Eye Res., 74, 1–6.
43) Kuwabara, T. (1975) The maturation of the lens cell: A morphologic study. Exp. Eye Res., 20, 427–443.
44) Kuwabara, T. & Imaizumi, M. (1974) Denucleation process of the lens. Invest. Ophthalmol., 13, 973–981.
45) Bassnett, S. (1992) Mitochondrial dynamics in differentiating fiber cells of the mammalian lens. Curr. Eye Res., 11, 1227–1232.
46) Bassnett, S. (1995) The fate of the Golgi apparatus and the endoplasmic reticulum during lens fiber cell differentiation. Invest. Ophthalmol. Vis. Sci., 36, 1793–1803.
47) Bassnett, S. & Beebe, D.C. (1992) Coincident loss of mitochondria and nuclei during lens fiber cell differentiation. Dev. Dyn., 194, 85–93.
48) De Maria, A. & Bassnett, S. (2007) DNase IIbeta distribution and activity in the mouse lens. Invest. Ophthalmol. Vis. Sci., 48, 5638–5646.
49) Bassnett, S. (1997) Fiber cell denucleation in the primate lens. Invest. Ophthalmol. Vis. Sci., 38, 1678–1687.
50) De Maria, A. & Arruti, C. (2004) DNase I and fragmented chromatin during nuclear degradation in adult bovine lens fibers. Mol. Vis., 10, 74–82.
51) Dahm, R., Gribbon, C., Quinlan, R.A., & Prescott, A.R. (1998) Changes in the nucleolar and coiled body compartments precede lamina and chromatin reorganization during fibre cell denucleation in the bovine lens. Eur. J. Cell Biol., 75, 237–246.
52) Bassnett, S. & Mataic, D. (1997) Chromatin degradation in differentiating fiber cells of the eye lens. J. Cell Biol., 137, 37–49.
53) McWilliams, T.G., Prescott, A.R., Villarejo-Zori, B., Ball, G., Boya, P., & Ganley, I.G. (2019) A comparative map of macroautophagy and mitophagy in the vertebrate eye. Autophagy, 15, 1296–1308.
54) Imai, F., Yoshizawa, A., Fujimori-Tonou, N., Kawakami, K., & Masai, I. (2010) The ubiquitin proteasome system is required for cell proliferation of the lens epithelium and for differentiation of lens fiber cells in zebrafish. Development, 137, 3257–3268.
55) Iida, A., Tabata, Y., Baba, Y., Fujii, T., & Watanabe, S. (2014) Critical roles of DNase1l3l in lens nuclear degeneration in zebrafish. Biochimie, 106, 68–74.
56) Appleby, D.W. & Modak, S.P. (1977) DNA degradation in terminally differentiating lens fiber cells from chick embryos. Proc. Natl. Acad. Sci. USA, 74, 5579–5583.
57) Counis, M.F., Chaudun, E., Arruti, C., Oliver, L., Sanwal, M., Courtois, Y., & Torriglia, A. (1998) Analysis of nuclear degradation during lens cell differentiation. Cell Death Differ., 5, 251–261.
58) Zhang, J., Cui, W.W., Du, C., Huang, Y., Pi, X., Guo, W., Wang, J., Huang, W., Chen, D., Li, J., et al. (2020) Knockout of DNase1l1l abrogates lens denucleation process and causes cataract in zebrafish. Biochim. Biophys. Acta Mol. Basis Dis., 1866, 165724.
59) Fujimoto, M., Izu, H., Seki, K., Fukuda, K., Nishida, T., Yamada, S., Kato, K., Yonemura, S., Inouye, S., & Nakai, A. (2004) HSF4 is required for normal cell growth and differentiation during mouse lens development. EMBO J., 23, 4297–4306.
60) Min, J.N., Zhang, Y., Moskophidis, D., & Mivechi, N.F. (2004) Unique contribution of heat shock transcription factor 4 in ocular lens development and fiber cell differentiation. Genesis, 40, 205–217.
61) Cui, X., Wang, L., Zhang, J., Du, R., Liao, S., Li, D., Li, C., Ke, T., Li, D.W., Huang, H., et al. (2013) HSF4 regulates DLAD expression and promotes lens de-nucleation. Biochim. Biophys. Acta Mol. Basis Dis., 1832, 1167–1172.
62) Medina-Martinez, O., Brownell, I., Amaya-Manzanares, F., Hu, Q., Behringer, R.R., & Jamrich, M. (2005) Severe defects in proliferation and differentiation of lens cells in Foxe3 null mice. Mol. Cell. Biol., 25, 8854–8863.
63) Cavalheiro, G.R., Matos-Rodrigues, G.E., Zhao, Y., Gomes, A.L., Anand, D., Predes, D., de Lima, S., Abreu, J.G., Zheng, D., Lachke, S.A., et al. (2017) N-myc regulates growth and fiber cell differentiation in lens development. Dev. Biol., 429, 105–117.
64) He, S., Pirity, M.K., Wang, W.L., Wolf, L., Chauhan, B.K., Cveklova, K., Tamm, E.R., Ashery-Padan, R., Metzger, D., Nakai, A., et al. (2010) Chromatin remodeling enzyme Brg1 is required for mouse lens fiber cell terminal differentiation and its denucleation. Epigenetics Chromatin, 3, 21.
65) He, S., Limi, S., McGreal, R.S., Xie, Q., Brennan, L.A., Kantorow, W.L., Kokavec, J., Majumdar, R., Hou, H. Jr., Edelmann, W., et al. (2016) Chromatin remodeling enzyme Snf2h regulates embryonic lens differentiation and denucleation. Development, 143, 1937–1947.
66) Siddam, A.D., Gautier-Courteille, C., Perez-Campos, L., Anand, D., Kakrana, A., Dang, C.A., Legagneux, V., Méreau, A., Viet, J., Gross, J.M., et al. (2018) The RNA-binding protein Celf1 post-transcriptionally regulates p27Kip1 and Dnase2b to control fiber cell nuclear degradation in lens development. PLoS Genet., 14, e1007278.
67) Shiokawa, D. & Tanuma, S. (1999) DLAD, a novel mammalian divalent cation-independent endonuclease with homology to DNase II. Nucleic Acids Res., 27, 4083–4089.
68) Chaffee, B.R., Shang, F., Chang, M.L., Clement, T.M., Eddy, E.M., Wagner, B.D., Nakahara, M., Nagata, S., Robinson, M.L., & Taylor, A. (2014) Nuclear removal during terminal lens fiber cell differentiation requires CDK1 activity: appropriating mitosis-related nuclear disassembly. Development, 141, 3388–3398.
69) Rowan, S., Chang, M.L., Reznikov, N., & Taylor, A. (2017) Disassembly of the lens fiber cell nucleus to create a clear lens: The p27 descent. Exp. Eye Res., 156, 72–78.
70) Lyu, L., Whitcomb, E.A., Jiang, S., Chang, M.L., Gu, Y., Duncan, M.K., Cvekl, A., Wang, W.L., Limi, S., Reneker, L.W., et al. (2016) Unfolded-protein response-associated stabilization of p27(Cdkn1b) interferes with lens fiber cell denucleation, leading to cataract. FASEB J., 30, 1087–1095.
71) De Duve, C. (1963) The lysosome. Sci. Am., 208, 64–72.
72) Klionsky, D.J. (2008) Autophagy revisited: A conversation with Christian de Duve. Autophagy, 4, 740–743.
73) Mizushima, N. & Komatsu, M. (2011) Autophagy: Renovation of cells and tissues. Cell, 147, 728–741.
74) Mizushima, N. (2022) SnapShot: Organelle degradation. Mol. Cell, 82, 1604–e1.
75) Tooze, S.A. & Yoshimori, T. (2010) The origin of the autophagosomal membrane. Nat. Cell Biol., 12, 831–835.
76) Nakatogawa, H. (2020) Mechanisms governing autophagosome biogenesis. Nat. Rev. Mol. Cell Biol., 21, 439–458.
77) Oku, M. & Sakai, Y. (2018) Three distinct types of microautophagy based on membrane dynamics and molecular machineries. BioEssays, 40, e1800008.
78) Sahu, R., Kaushik, S., Clement, C.C., Cannizzo, E.S., Scharf, B., Follenzi, A., Potolicchio, I., Nieves, E., Cuervo, A.M., & Santambrogio, L. (2011) Microautophagy of cytosolic proteins by late endosomes. Dev. Cell, 20, 131–139.
79) Kaushik, S. & Cuervo, A.M. (2018) The coming of age of chaperone-mediated autophagy. Nat. Rev. Mol. Cell Biol., 19, 365–381.
80) Chino, H. & Mizushima, N. (2020) ER-Phagy: Quality control and turnover of endoplasmic reticulum. Trends Cell Biol., 30, 384–398.
81) Csizmadia, T. & Juhasz, G. (2020) Crinophagy mechanisms and its potential role in human health and disease. Prog. Mol. Biol. Transl. Sci., 172, 239–255.
82) Sugiura, A., McLelland, G.L., Fon, E.A., & McBride, H.M. (2014) A new pathway for mitochondrial quality control: mitochondrial-derived vesicles. EMBO J., 33, 2142–2156.
83) Kaizuka, T., Morishita, H., Hama, Y., Tsukamoto, S., Matsui, T., Toyota, Y., Kodama, A., Ishihara, T., Mizushima, T., & Mizushima, N. (2016) An Autophagic Flux Probe that Releases an Internal Control. Mol. Cell, 64, 835–849.
84) Mizushima, N., Yamamoto, A., Matsui, M., Yoshimori, T., & Ohsumi, Y. (2004) In vivo analysis of autophagy in response to nutrient starvation using transgenic mice expressing a fluorescent autophagosome marker. Mol. Biol. Cell, 15, 1101–1111.
85) Kuma, A., Komatsu, M., & Mizushima, N. (2017) Autophagy-monitoring and autophagy-deficient mice. Autophagy, 13, 1619–1628.
86) Costello, M.J., Brennan, L.A., Basu, S., Chauss, D., Mohamed, A., Gilliland, K.O., Johnsen, S., Menko, S., & Kantorow, M. (2013) Autophagy and mitophagy participate in ocular lens organelle degradation. Exp. Eye Res., 116, 141–150.
87) Khan, S.Y., Ali, M., Kabir, F., Na, C.H., Delannoy, M., Ma, Y., Qiu, C., Costello, M.J., Hejtmancik, J.F., & Riazuddin, S.A. (2022) The role of FYCO1-dependent autophagy in lens fiber cell differentiation. Autophagy, 28, 1–18.
88) Basu, S., Rajakaruna, S., Reyes, B., Van Bockstaele, E., & Menko, A.S. (2014) Suppression of MAPK/JNK-MTORC1 signaling leads to premature loss of organelles and nuclei by autophagy during terminal differentiation of lens fiber cells. Autophagy, 10, 1193–1211.
89) Gheyas, R., Ortega-Alvarez, R., Chauss, D., Kantorow, M., & Menko, A.S. (2022) Suppression of PI3K signaling is linked to autophagy activation and the spatiotemporal induction of the lens organelle free zone. Exp. Cell Res., 412, 113043.
90) Jao, L.E., Wente, S.R., & Chen, W. (2013) Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proc. Natl. Acad. Sci. USA, 110, 13904–13909.
91) Mardian, E.B., Bradley, R.M., & Duncan, R.E. (2015) The HRASLS (PLA/AT) subfamily of enzymes. J. Biomed. Sci., 22, 99.
92) Uyama, T., Tsuboi, K., & Ueda, N. (2017) An involvement of phospholipase A/acyltransferase family proteins in peroxisome regulation and plasmalogen metabolism. FEBS Lett., 591, 2745–2760.
93) Murakami, M., Sato, H., & Taketomi, Y. (2020) Updating phospholipase A2 biology. Biomolecules, 10, 1457.
94) Shinohara, N., Uyama, T., Jin, X.H., Tsuboi, K., Tonai, T., Houchi, H., & Ueda, N. (2011) Enzymological analysis of the tumor suppressor A-C1 reveals a novel group of phospholipid-metabolizing enzymes. J. Lipid Res., 52, 1927–1935.
95) Jin, X.H., Okamoto, Y., Morishita, J., Tsuboi, K., Tonai, T., & Ueda, N. (2007) Discovery and characterization of a Ca2+-independent phosphatidylethanolamine N-acyltransferase generating the anandamide precursor and its congeners. J. Biol. Chem., 282, 3614–3623.
96) Jin, X.H., Uyama, T., Wang, J., Okamoto, Y., Tonai, T., & Ueda, N. (2009) cDNA cloning and characterization of human and mouse Ca(2+)-independent phosphatidylethanolamine N-acyltransferases. Biochim. Biophys. Acta Mol. Cell Biol. Lipids, 1791, 32–38.
97) Uyama, T., Morishita, J., Jin, X.H., Okamoto, Y., Tsuboi, K., & Ueda, N. (2009) The tumor suppressor gene H-Rev107 functions as a novel Ca2+-independent cytosolic phospholipase A1/2 of the thiol hydrolase type. J. Lipid Res., 50, 685–693.
98) Uyama, T., Jin, X.H., Tsuboi, K., Tonai, T., & Ueda, N. (2009) Characterization of the human tumor suppressors TIG3 and HRASLS2 as phospholipid-metabolizing enzymes. Biochim. Biophys. Acta Mol. Cell Biol. Lipids, 1791, 1114–1124.
99) Duncan, R.E., Sarkadi-Nagy, E., Jaworski, K., Ahmadian, M., & Sul, H.S. (2008) Identification and functional characterization of adipose-specific phospholipase A2 (AdPLA). J. Biol. Chem., 283, 25428–25436.
100) Hajnal, A., Klemenz, R., & Schafer, R. (1994) Subtraction cloning of H-rev107, a gene specifically expressed in H-ras resistant fibroblasts. Oncogene, 9, 479–490.
101) Sers, C., Emmenegger, U., Husmann, K., Bucher, K., Andres, A.C., & Schafer, R. (1997) Growth-inhibitory activity and downregulation of the class II tumor-suppressor gene H-rev107 in tumor cell lines and experimental tumors. J. Cell Biol., 136, 935–944.
102) DiSepio, D., Ghosn, C., Eckert, R.L., Deucher, A., Robinson, N., Duvic, M., Chandraratna, R.A., & Nagpal, S. (1998) Identification and characterization of a retinoid-induced class II tumor suppressor/growth regulatory gene. Proc. Natl. Acad. Sci. USA, 95, 14811–14815.
103) Jaworski, K., Ahmadian, M., Duncan, R.E., Sarkadi-Nagy, E., Varady, K.A., Hellerstein, M.K., Lee, H.Y., Samuel, V.T., Shulman, G.I., Kim, K.H., et al. (2009) AdPLA ablation increases lipolysis and prevents obesity induced by high-fat feeding or leptin deficiency. Nat. Med., 15, 159–168.
104) Hummasti, S., Hong, C., Bensinger, S.J., & Tontonoz, P. (2008) HRASLS3 is a PPARgamma-selective target gene that promotes adipocyte differentiation. J. Lipid Res., 49, 2535–2544.
105) Uyama, T., Ichi, I., Kono, N., Inoue, A., Tsuboi, K., Jin, X.H., Araki, N., Aoki, J., Arai, H., & Ueda, N. (2012) Regulation of peroxisomal lipid metabolism by catalytic activity of tumor suppressor H-rev107. J. Biol. Chem., 287, 2706–2718.
106) Uyama, T., Kawai, K., Kono, N., Watanabe, M., Tsuboi, K., Inoue, T., Araki, N., Arai, H., & Ueda, N. (2015) Interaction of phospholipase A/Acyltransferase-3 with Pex19p: A possible involvement in the down-regulation of peroxisomes. J. Biol. Chem., 290, 17520–17534.
107) 宇山徹(2015)ホスホリパーゼA/アシルトランスフェラーゼ-3を介したペルオキシソームの制御機構.生化学,87, 633-635.
108) Staring, J., von Castelmur, E., Blomen, V.A., van den Hengel, L.G., Brockmann, M., Baggen, J., Thibaut, H.J., Nieuwenhuis, J., Janssen, H., van Kuppeveld, F.J., et al. (2017) PLA2G16 represents a switch between entry and clearance of Picornaviridae. Nature, 541, 412–416.
109) McKnight, K.L. & Lemon, S.M. (2017) Virology: Ins and outs of picornaviruses. Nature, 541, 299–300.
110) Elling, U., Wimmer, R.A., Leibbrandt, A., Burkard, T., Michlits, G., Leopoldi, A., Micheler, T., Abdeen, D., Zhuk, S., Aspalter, I.M., et al. (2017) A reversible haploid mouse embryonic stem cell biobank resource for functional genomics. Nature, 550, 114–118.
111) Wu, C., Orozco, C., Boyer, J., Leglise, M., Goodale, J., Batalov, S., Hodge, C.L., Haase, J., Janes, J., Huss, J.W. 3rd, et al. (2009) BioGPS: an extensible and customizable portal for querying and organizing gene annotation resources. Genome Biol., 10, R130.
112) Tosteson, M.T. & Chow, M. (1997) Characterization of the ion channels formed by poliovirus in planar lipid membranes. J. Virol., 71, 507–511.
113) Schober, D., Kronenberger, P., Prchla, E., Blaas, D., & Fuchs, R. (1998) Major and minor receptor group human rhinoviruses penetrate from endosomes by different mechanisms. J. Virol., 72, 1354–1364.
114) Eapen, V.V., Swarup, S., Hoyer, M.J., Paulo, J.A., & Harper, J.W. (2021) Quantitative proteomics reveals the selectivity of ubiquitin-binding autophagy receptors in the turnover of damaged lysosomes by lysophagy. eLife, 10, e72328.
115) Aits, S., Kricker, J., Liu, B., Ellegaard, A.M., Hamalisto, S., Tvingsholm, S., Corcelle-Termeau, E., Høgh, S., Farkas, T., Holm Jonassen, A., et al. (2015) Sensitive detection of lysosomal membrane permeabilization by lysosomal galectin puncta assay. Autophagy, 11, 1408–1424.
116) Lee, M.T., Sun, T.L., Hung, W.C., & Huang, H.W. (2013) Process of inducing pores in membranes by melittin. Proc. Natl. Acad. Sci. USA, 110, 14243–14248.
117) Dennis, E.A., Cao, J., Hsu, Y.H., Magrioti, V., & Kokotos, G. (2011) Phospholipase A2 enzymes: physical structure, biological function, disease implication, chemical inhibition, and therapeutic intervention. Chem. Rev., 111, 6130–6185.
118) Weltzien, H.U. (1979) Cytolytic and membrane-perturbing properties of lysophosphatidylcholine. Biochim. Biophys. Acta Rev. Biomembr., 559, 259–287.
119) Tanabe, M., Sasai, N., Nagata, K., Liu, X.D., Liu, P.C., Thiele, D.J., & Nakai, A. (1999) The mammalian HSF4 gene generates both an activator and a repressor of heat shock genes by alternative splicing. J. Biol. Chem., 274, 27845–27856.
120) Bu, L., Jin, Y., Shi, Y., Chu, R., Ban, A., Eiberg, H., Andres, L., Jiang, H., Zheng, G., Qian, M., et al. (2002) Mutant DNA-binding domain of HSF4 is associated with autosomal dominant lamellar and Marner cataract. Nat. Genet., 31, 276–278.
121) Gao, M., Huang, Y., Wang, L., Huang, M., Liu, F., Liao, S., Yu, S., Lu, Z., Han, S., Hu, X., et al. (2017) HSF4 regulates lens fiber cell differentiation by activating p53 and its downstream regulators. Cell Death Dis., 8, e3082.
122) Hoang, T.V., Kumar, P.K., Sutharzan, S., Tsonis, P.A., Liang, C., & Robinson, M.L. (2014) Comparative transcriptome analysis of epithelial and fiber cells in newborn mouse lenses with RNA sequencing. Mol. Vis., 20, 1491–1517.
123) Hu, Y.Z., Zhang, J., Li, S., Wang, C., Chu, L., Zhang, Z., Ma, Z., Wang, M., Jiang, Q., Liu, G., et al. (2013) The transcription activity of heat shock factor 4b is regulated by FGF2. Int. J. Biochem. Cell Biol., 45, 317–325.
124) Chauss, D., Basu, S., Rajakaruna, S., Ma, Z., Gau, V., Anastas, S., Brennan, L.A., Hejtmancik, J.F., Menko, A.S., & Kantorow, M. (2014) Differentiation state-specific mitochondrial dynamic regulatory networks are revealed by global transcriptional analysis of the developing chicken lens. G3 (Bethesda), 4, 1515–1527.
125) Cui, X., Han, J., Li, J., Cui, W.W., Wu, D.D., Liu, S., Xue, W., Wang, X.X., Ma, Y., Zhang, J., et al. (2018) Downregulation of heat shock factor 4 transcription activity via MAPKinase phosphorylation at Serine 299. Int. J. Biochem. Cell Biol., 105, 61–69.
126) Pereira, P., Shang, F., Hobbs, M., Girao, H., & Taylor, A. (2003) Lens fibers have a fully functional ubiquitin-proteasome pathway. Exp. Eye Res., 76, 623–631.
127) Grullich, C., Duvoisin, R.M., Wiedmann, M., & van Leyen, K. (2001) Inhibition of 15-lipoxygenase leads to delayed organelle degradation in the reticulocyte. FEBS Lett., 489, 51–54.
128) van Leyen, K., Duvoisin, R.M., Engelhardt, H., & Wiedmann, M. (1998) A function for lipoxygenase in programmed organelle degradation. Nature, 395, 392–395.
129) Loynes, C.A., Lee, J.A., Robertson, A.L., Steel, M.J., Ellett, F., Feng, Y., Levy, B.D., Whyte, M.K.B., & Renshaw, S.A. (2018) PGE2 production at sites of tissue injury promotes an anti-inflammatory neutrophil phenotype and determines the outcome of inflammation resolution in vivo. Sci. Adv., 4, eaar8320.
130) Rademacher, M., Kuhn, H., & Borchert, A. (2020) Systemic deficiency of mouse arachidonate 15-lipoxygenase induces defective erythropoiesis and transgenic expression of the human enzyme rescues this phenotype. FASEB J., 34, 14318–14335.
131) Sun, D. & Funk, C.D. (1996) Disruption of 12/15-lipoxygenase expression in peritoneal macrophages. Enhanced utilization of the 5-lipoxygenase pathway and diminished oxidation of low density lipoprotein. J. Biol. Chem., 271, 24055–24062.
132) Funk, C.D., Chen, X.S., Johnson, E.N., & Zhao, L. (2002) Lipoxygenase genes and their targeted disruption. Prostaglandins Other Lipid Mediat., 68-69, 303–312.
133) De Maria, A., Shi, Y., Kumar, N.M., & Bassnett, S. (2009) Calpain expression and activity during lens fiber cell differentiation. J. Biol. Chem., 284, 13542–13550.
134) Zandy, A.J., Lakhani, S., Zheng, T., Flavell, R.A., & Bassnett, S. (2005) Role of the executioner caspases during lens development. J. Biol. Chem., 280, 30263–30272.
著者紹介Author Profile
 森下 英晃(もりした ひであき)
森下 英晃(もりした ひであき)順天堂大学医学部生理学第二講座准教授.医学博士(東京医科歯科大学).
略歴1982年広島市生まれ.07年九州大学医学部医学科卒業.09年国立国際医療研究センター初期臨床研修修了.10年日本学術振興会特別研究員.13年東京医科歯科大学大学院医歯学総合研究科博士課程修了.同年東京大学大学院医学系研究科特任研究員.16年同助教.17年ERATO水島細胞内ダイナミクスプロジェクト・グループリーダー.19年より順天堂大学医学部講師,22年より現職.21年よりAMED PRIME(プロテオスタシス領域)研究開発代表.
研究テーマと抱負マウスやゼブラフィッシュを用いて,生体内における多様な細胞内分解現象の分子機構や生理的意義の解明を目指しています.
ウェブサイトhttps://researchmap.jp/h-morishita
趣味登山,野球観戦,水泳,キノコ狩り,クラシック音楽鑑賞(特にブルックナー,ベートーヴェン),読書.