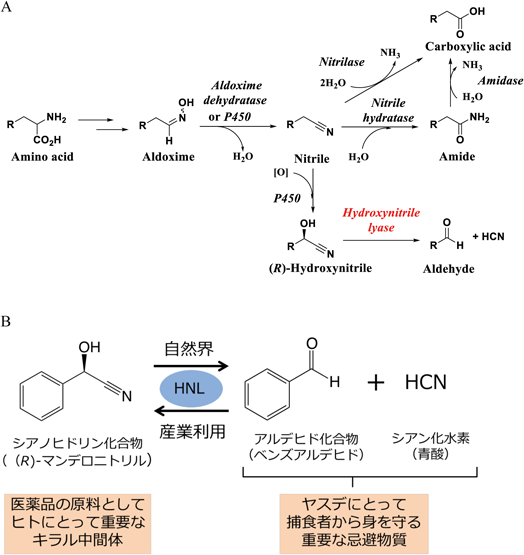
ヒドロキシニトリルリアーゼ(hydroxynitrile lyase:HNL)(EC 4.1.2.10, 4.1.2.11, 4.1.2.46, 4.1.2.47)は主に植物に存在することが知られてきた1).特に,アーモンドなどの植物由来のHNLに関して,有機合成への利用研究が酵素化学的な研究に先行していた.近年になって,HNLの分布,特性,構造決定などについての報告が急増している2).HNLは,自然界におけるシアン生合成(アルドキシム–ニトリル経路)の最後のステップを触媒し,シアノヒドリン(α-ヒドロキシニトリル)をシアン化水素と対応するアルデヒドまたはケトンに分解する.この過程は,草食動物や微生物に対する防御機構の一つとして機能すると考えられている1).一方,光学活性なシアノヒドリンは,医薬,農薬など,多くの化学製品を合成するための重要なキラル中間体であり,HNLを生体触媒として用い,プロキラルなアルデヒドやケトンに対して立体選択的にシアン化水素を付加することで不斉合成される3–5).
我々は,微生物酵素の探索から酵素分子の構造および産業利用に至る研究を行ってきたが,さらに植物を研究対象として加え,応用微生物学分野で行われるスクリーニングと同様な手法で,市販の果物や植物から活性を指標として新しいHNLを探索してきた2, 6).植物由来のHNLとしては,ウメ(Prunus mume)などバラ科に属する5種類の植物,パッションフルーツ(Passiflora edulis)7)および南天(Nandina domestica)8)にR立体選択的なHNL活性を見いだし,そのうちのパッションフルーツ由来HNL(PeHNL)の構造を明らかにした9).さらに,同様に中国雲南省原産のBaliospermum montanumの葉に,S立体選択的なHNLを見いだし10),酵素化学的諸性質を明らかにするとともにX線結晶構造解析を行った11).一方で,他の研究者によってシダ植物のDavallia tyermannii由来のHNL12)や細菌から金属依存性のHNLが発見されている13).本稿では,節足動物であるヤスデ,特にヤンバルトサカヤスデなどにおける新しいHNLの発見,立体構造解析および本酵素の産業応用に関する研究を中心に,これまでに筆者が取り組んだ植物由来HNLをはじめ,生体内シアン生合成(アルドキシム–ニトリル経路)関連酵素(図1A)に関する研究成果を織り交ぜて紹介する.なお,細菌のアルドキシム–ニトリル経路は,アルドキシム脱水酵素やニトリルヒドラターゼなどのニトリル分解酵素からなり,HNLを有さない.代謝における存在意義は,細菌,動物および植物でそれぞれに異なる(図1B)2).
1)ヤンバルトサカヤスデにおけるHNL活性の発見
自然界から広くHNL活性を探索してきた経緯から,筆者は節足動物であるヤスデ類もHNLと類似する酵素を有しているのではないかと考えるようになった.以前に購入した書籍の一部に以下の記述があり,大いに興味を抱いた.すなわち,中部地方の山間部では,ほぼ6~11年に1度,秋にヤスデ(学名:ヤスデ綱ハバヤスデ科キシャヤスデ;Parafontaria laminata: Diplopoda:Xystodesmidae)の大発生が起き,小海線などにおいて列車の運行が阻害されることが起こっていた14).これらのヤスデはオビヤスデ目(Polydesmida)に属し,防御化合物の混合物を持つシアン発生ヤスデとして知られていた15).しかし,一般にヤスデは野外での個体密度が低く,大量に採集することが困難であるので,微生物のように培養して酵素化学実験の対象にすることは難しい状態であった.そこでヤンバルトサカヤスデ(Chamberlinius hualienensis Wang)に着目した.ヤンバルトサカヤスデは台湾からの侵入種であり,沖縄本島で大発生し,一部の地域では,産卵期直前の秋に大群遊することが報告されていた16).2010年,ヤンバルトサカヤスデが南九州で大発生し列車を停止させた事件が起き,ニュースを手がかりに現地に向かい,予備実験に必要なヤスデ(約3 kg)を採捕した.ヤンバルトサカヤスデの摩砕抽出液と(R,S)-マンデロニトリル(MAN)を反応させ,ベンズアルデヒド(BA)の生成量が反応時間とともに増加することを確認した.また,BAとシアン化カリウムから(R)-MANが生成されることも確認し,ヤンバルトサカヤスデがHNL(ChuaHNL)活性を有することを発見した.それ以来10年以上にわたって南九州,静岡,八丈島などに赴き,ヤンバルトサカヤスデおよび国内のヤスデなどを採捕し研究を継続している.
2)ヤスデの防御物質組成と生合成経路
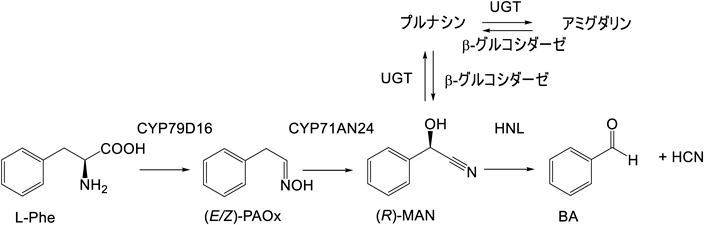
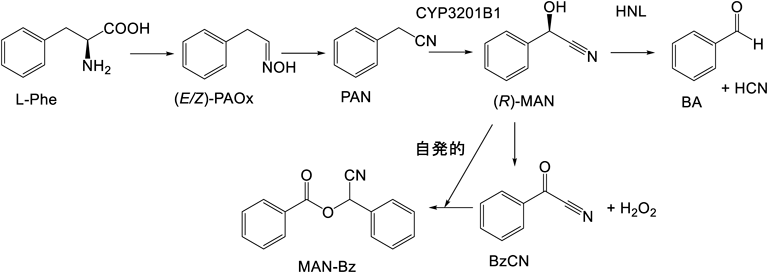
ヤスデが強力な防御物質としてシアン化水素とBAなどを分泌することは知られていたが15),近年の機器分析技術の進歩により,分泌物の主要成分はBAにシアン化水素が付加した(R)-MANとその分解物[BA,安息香酸,ベンジルアルコール,ベンゾイルシアニド(BzCN),マンデロニトリルベンゾアート(MAN-Bz)など]であることが明らかにされた17–20).当時我々はウメのシアン化水素発生経路(図2)21)を明らかにしていたので,ヤスデは,(R)-MANからBAとシアン化水素を生成する点でウメと類似の代謝経路を有していると期待された.そこでERATO浅野酵素活性分子プロジェクトにおいて,桑原らは,重水素標識したL-Phe(D5-L-Phe),(E/Z)-フェニルアセトアルドキシム(PAOx, D5-PAOx)またはフェニルアセトニトリル(PAN, D5-PAN)水溶液をさまざまな週齢のヤンバルトサカヤスデとともにバイヤル中に置き,(R)-MANへの取り込みを調べた.その結果,6齢(老齢幼虫)と7齢(亜成体)のヤスデで,BA,安息香酸,(R)-MANおよびMAN-BzのD5化物が検出された.これらの結果から,(R)-MANは,(E/Z)-PAOxの脱水によるPANの生成を経て,その後水酸化されて生成されると推定した(図3)22, 23).この推定代謝経路を構成する酵素タンパク質をコードする遺伝子を同定し,2種類の新規酵素を発見した.その概要を以下に紹介する.
3)新規酵素マンデロニトリル酸化酵素の発見とヤスデの防御機構における本酵素の役割
ヤンバルトサカヤスデからは,HNL(ChuaHNL)24)の他に,シアノヒドリンの分解活性も検出されたことから,まず酵素本体であるマンデロニトル酸化酵素(ChuaMOX)を同定した25).ChuaMOXは,533アミノ酸からなる糖鎖修飾された単量体酵素であり,FADを補酵素として含む.生理学的に関連する基質としては(R)-MANのみに対して活性を示し,BzCNと過酸化水素を生成した(図3).本酵素は,幅広いpHおよび温度条件下で安定である.その一次構造は,アメリカンロブスター(Homarus americanus),アルゼンチンアリ(Linepithema humile)などのグルコース酸化酵素と推定されるタンパク質に50%程度の相同性を有している.本酵素には,新酵素,マンデロニトリル酸化酵素(R)-mandelonitrile:oxygen oxidoreductaseとしてEC 1.1.3.49が付与された.
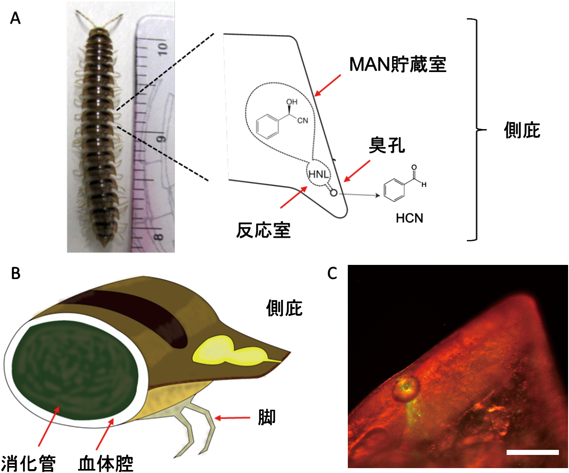
組織化学的な研究から,本酵素は血液中に局在しているが,一方基質の(R)-MANは,体節にある側庇と呼ばれる部位にある貯蔵室で生合成および貯蔵されている(図4)26).酵素が基質と出会うのはどのようなときであろうか.ヤスデが大型の捕食者に捕まったとき,体内の筋肉を激しく収縮させ,その結果,血圧が非常に高くなる.(R)-MAN貯蔵室と反応室の膜を内因的に破裂させると,血液中のChuaMOXは,貯蔵室と反応室に流れ込む.酵素活性はpH 3.5からpH 5の範囲で高く,血液のpH 7で最も低い.pHが7から4.6に低下することにより(R)-MANからBzCNが合成され,犠牲となった体から放出され,毒性滲出液として作用する.このように,ChuaMOXは大型の捕食者から無傷の群れを守ることができるように進化した酵素であると推定した.本酵素はヤスデの防御機構に働くと考えられる.
4)ヤンバルトサカヤスデにおける(R)-マンデロニトリル生合成酵素の探索
植物におけるシアン発生は,アミノ酸代謝の一部である.すでに,Møllerらによってシアン配糖体のデュリン(Dhurrin)の生合成がL-Tyrからなされることが知られていたが27),我々は,ウメにおいて,L-PheからシトクロムP450(CYP79D16)によって,(E/Z)-PAOxに変換され,さらにCYP71AN24が触媒する逐次反応によって,(R)-MANが生合成されること21),ならびにHNLの一次構造28),立体構造29)および(R)-シアノヒドリンの合成への利用30, 31)などを明らかにした(図1).
ヤンバルトサカヤスデにおいても,上述のとおり,植物と同様な(R)-MAN生合成経路上の化合物が検出されたことから(図2),同様にシトクロムP450が関与していると予想された.トランスクリプトームデータからシトクロムP450をコードするcDNAをクローニングし,異種発現することで(R)-MANの生合成に関わるシトクロムP450を同定した.ヤンバルトサカヤスデから32種のミクロソーム型シトクロムP450をコードするcDNAをクローニングし,そのうち,CYP3201B1がPANを水酸化して(R)-MANに変換することを見いだした.このCYP3201B1は,ウメ由来CYP71AN24とは異なり,(E/Z)-PAOxを基質とせず,PANを基質とした.よって,ヤスデの(R)-MANなどの生合成経路は,植物の経路と似てはいるものの相違点もあり,関連して生合成経路を構成する酵素群も異なることが示された(図3)23).
1)ヤンバルトサカヤスデ由来HNLの性質とシアン代謝
上記の経緯で,我々は台湾原産のヤンバルトサカヤスデ抽出液中にHNL活性とともにユニークなシアノヒドリン合成経路を新たに見いだし,ヤンバルトサカヤスデに新規のHNL(ChuaHNL)の存在が強く示唆された.ChuaHNLのシアノヒドリン合成の比活性は,既知HNLより著しく高値であった.よって,ChuaHNLの反応メカニズムを解明することは,ChuaHNLの産業利用だけでなく,既知のHNLの理解にもつながると考えられた.しかし,ChuaHNLは新規酵素であったため,既知のHNLやBLASTデータベースの情報からは構造や触媒作用のメカニズムは予測できなかった.そこでヤンバルトサカヤスデ生体試料からChuaHNLを精製し,本酵素の構造と機能を詳細に解析した.
合計約30 kgのヤンバルトサカヤスデからChuaHNLを精製し,酵素化学的諸性質を明らかにした24).精製ChuaHNLは,PaHNLと同様に広い温度範囲と広いpH領域で安定であった.また,ChuaHNLによるMAN合成の比活性は7420 U/mgであり,ビワ由来HNL(31 U/mg)32),パッションフルーツ由来PeHNL(136 U/mg)33),ウメ由来HNL(220 U/mg)21)および産業利用されているアーモンド由来PaHNL(1450 U/mg)34)を大きく上回る.ChuaHNLは,表1に示したアルデヒドを含めて計29種類の化合物を基質とし,対応するシアノヒドリンを7.5~90%の範囲の鏡像体過剰率(enantiomeric excess:e.e.)で不斉合成することが可能であった.
表1 ChuaHNLの基質特異性24)| 基 質 | 構 造 | Km (mM) | kcat (s−1) | kcat/Km (mM−1 s−1) | Vmax (µmol min−1 mg−1) |
|---|
| benzaldehyde | 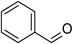 | 3.0±0.2 | 3,390±71 | 1,100 | 8,186±171 |
| 4-bromo-benzaldehyde | 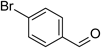 | 10.0±0.7 | 8,610±190 | 860 | 20,778±470 |
| 3-methyl-benzaldehyde | 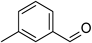 | 10.5±2.5 | 6,890±620 | 660 | 16,639±1491 |
| 4-methyl-benzaldehyde | 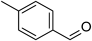 | 5.4±0.9 | 5,520±250 | 1,000 | 13,338±611 |
| 3-methoxy-benzaldehyde | 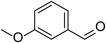 | 7.7±1.3 | 1,680±87 | 220 | 4,060±211 |
| 4-methoxy-benzaldehyde | 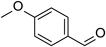 | 7.5±1.1 | 3,480±15 | 460 | 8,394±373 |
| 2,4-dimethyl-benzaldehyde | 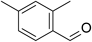 | 1.3±0.3 | 300±16 | 230 | 730±38 |
| 2-thiophene-carboxaldehyde | 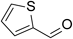 | 18.0±2.1 | 1,490±85 | 83 | 3,605±204 |
| 4-biphenyl-carboxaldehyde | 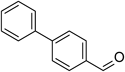 | 10.3±2.1 | 49.0±3.5 | 4.7 | 118.7±8.4 |
我々は,組織化学的な局在化実験により,ChuaHNLは,ヤスデ体節の側庇の内部にある臭孔の開口部近傍に局在していることを明らかにした24).ヤスデは,臭孔と呼ばれる穴を通って生成物であるシアン化水素ガスとBAが排出される道の上流に,内部細胞によって合成された純粋な(R)-MANの貯蔵室を有すると考えられている(図4)26).ヤスデでは植物のような配糖体化されたMANが検出されず,そのまま貯蔵されていることは注目に値する24).貯蔵室に接続された次の反応室では,ChuaHNLが特異的に生合成されており,貯蔵室から(R)-MANが高濃度で供給され,生成物であるBAとシアン化水素は,刺激応答時に瞬時に臭孔を通って体節の外に押し出される26).ChuaHNLが示した(R)-MANに対する高いKm値(8.7 mM)35)および(R,S)-MAN分解に対する高い比活性[Vmax:1900 U/mg(Dadashipour,未発表)]は,他の植物HNLがシアン配糖体をβ-グルコシダーゼ反応で加水分解した後,より低い基質濃度で作用するのとは異なり,行動的な動物の酵素としてきわめて合目的的である.(R)-MANに対するKm値は1.0 mM前後,BAに対するKm値は10 mM以下である1, 8).
2)HNLの立体構造にみる多様性
HNL研究によって,生物学的なシアン発生の興味深い側面が明らかになり,またHNLの産業用触媒としての可能性が研究者によって認識されて以来,HNLが次々と植物や細菌から発見され,その酵素学的・生化学的特性解析と並行してさまざまな生物種由来の酵素の結晶構造解析が行われてきた.現在Protein Data Bank(PDB)に登録されているHNLの結晶構造は,FAD結合性酸化還元酵素36),α/β-ヒドロラーゼ11, 37, 38),キュピン39),Bet v112),二量体α+βバレルフォールド9),Zn2+依存性アルコール脱水素酵素40),および本報告のリポカリン35, 41)の七つのスーパーファミリーに属する.従来からよく研究されてきたアーモンド(Prunus dulcis Miller(D.A. Webb)syn. P. amygdalus Batsch)由来のHNL(PaHNL)にはFADが含まれている12).X線結晶構造解析からFADは酵素の触媒作用には直接関与していないが,酵素の構造を安定化させる役割を持っていると考えられている42).キャッサバManihot esculenta(MeHNL),パラゴムノキHevea brasiliensis(HbHNL)および我々が発見した中国雲南省原産のBaliospermum montanum(BmHNL)由来HNLの構造は,α/β-ヒドロラーゼファミリーに属する.我々は,X線結晶構造解析を行い,(S)-MANを活性部位に結合させたBmHNL(apo2)とS-HbHNLの構造比較から,BmHNLは,ベンゼン環を含む基質に対する高い親和性を有しており,BmHNLの入り口領域の疎水性残基が基質のベンゼン環と疎水性相互作用を形成していることを明らかにした11).グラム陰性好冷性細菌Granulicella tundricola由来のHNL(GtHNL)は初めて結晶化された唯一の細菌由来HNLであり,金属依存性酵素として小さなバレル構造を持つキュピンフォールドに属しているが,現在ではシアン代謝とは無関係であると考えられている39).また,シダ植物由来のDtHNLの構造は,反平行のβシートがαヘリックスで包まれたBet v1様の構造であることが報告された12).Zn2+依存性アルコール脱水素酵素を祖先とする亜麻(Linum usitatissimum)由来HNLはNAD+を含んでいるが,このNAD+もPaHNLのFADと同様に酸化還元反応には関与していないと判断される40).我々が単離したパッションフルーツ(Passiflora edulis)の種および葉に含まれるHNL(PeHNL)は,二量体α+βバレルファミリーに属する初めてのHNLであり,既知のHNLの中で最も短いHNLである9).HNLにこれほど多くの立体構造の多様性がみられる理由は,本酵素の反応機構がたった1残基側鎖の塩基性触媒作用に依存する比較的単純なものであったために,種々の祖先タンパク質がHNLに進化したからと考えられ,収束進化の拡大例として興味深い.新しい三次構造を有するHNLが今後も報告される可能性はまだ残っている8).
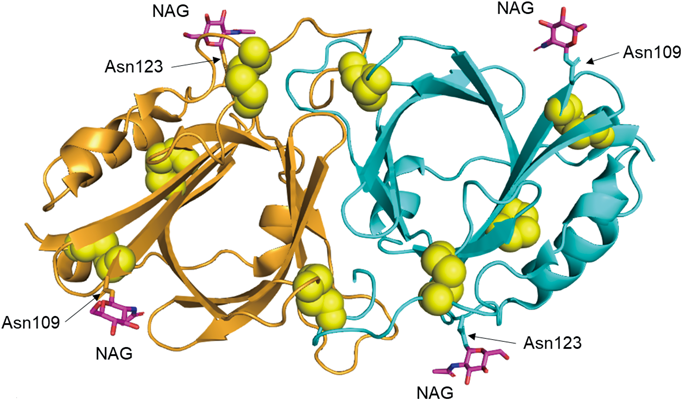
3)ヤンバルトサカヤスデ由来HNLの構造機能解析—輸送タンパク質リポカリンファミリーに属する酵素—35, 41)
天然のChuaHNLの配位子フリーの状態,ならびに酢酸,シアン化物イオン,阻害剤(チオシアン酸塩,ヨード酢酸塩)との複合体の結晶構造を明らかにした.本酵素の全体構造は,八つの逆平行βシートからなり,バレル構造を形成していた.本酵素は,二量体を形成しており,1分子内に三つ,二量体間に二つのジスルフィド結合を有していた.
BLAST検索ではChuaHNLとの相同配列は見つからなかったが,DALIサーバーを用いた構造比較検索では,リポカリンファミリーのタンパク質の構造に類似していることが明らかになった.代表的なリポカリンの多くは,Zスコアが10以上となり,構造的に大きく類似していることがわかった.ChuaHNLと典型的なリポカリンとのアミノ酸配列の同一性は8%未満であった.一次構造の相同性が非常に低いにもかかわらず,ChuaHNLは,ヒトのレチノール結合タンパク質4(RBP4)の二次構造とよく類似していた(図5).ヤスデから精製したChuaHNLは,Asn109とAsn123においてN-アセチルグルコサミン(NAG)部位が観察された.また,Pichia pastorisの発現系で生産したChuaHNLにおいても,同じ糖鎖付加部位で糖鎖修飾されていることを確認した.
(R)-MANとChuaHNLのドッキングモデルでは,水酸基がArg38とLys117と水素結合を形成し,ニトリル基がTyr103と水素結合を形成することが示唆された.また,変異解析により,これらの残基が酵素反応において重要であることが示された.これらの研究から,ChuaHNLではLys残基が塩基として作用する新しい反応機構が推定された.シアノヒドリンの開裂反応において,Lys117はシアノヒドリンの水酸基からプロトンを奪う一般塩基として働き,Arg38はシアン化物イオンにプロトンを供与する酸として働くことが提案される.シアノヒドリンの合成時には,その逆のメカニズムが起こることになる.
従来報告されたHNLの反応機構のモデルにおいて,いずれも触媒反応の第一段階はシアノヒドリンの水酸基からの脱プロトン化であるが,酵素によって異なる触媒残基を用いている.PaHNL36)やPeHNL9)では,His残基が一般的塩基として働くが,HbHNL38)などでは,Hisによって脱プロトン化されたSerが一般的塩基として働く.DtHNL12)とChuaHNLでは,それぞれTyrとLysを一般的塩基として用いている.
リポカリンタンパク質ファミリーは,生物界に広く分布することが知られており,分子量は主に18,000から20,000で,その一次配列の多様性は大きい43).典型的なリポカリンの80%以上は,配列の同一性が1~20%という低い範囲にある一方で,リポカリンの三次元構造は非常によく保存されている44).機能的には多様性があり,レチノールの輸送,嗅覚,隠蔽着色,フェロモンなどの役割を担っている.また,免疫調節や細胞のホメオスタシスの調節にも機能すると考えられている45).プロスタグランジンD合成酵素(prostaglandin-D synthase, EC 5.3.99.2)は,リポカリンファミリーに属するタンパク質の中で,よく特徴づけられた唯一の酵素である46, 47).最近,このタンパク質は,酵素としての機能と,脳脊髄液中の多様な親油性化合物の細胞外輸送体としての機能の二重の役割を担っていることが報告されている48).
4)ChuaHNL遺伝子の発現
ChuaHNLは,HNLとしてきわめて高い比活性(7420 U/mg)と高いエナンチオ選択性を示すため,(R)-MANなどさまざまなシアノヒドリンの合成に利用できると考えられる.しかし,ChuaHNLを容易に調製し,基礎および応用研究に用いるには,酵素生産が可能なシステムを構築することが必要である.そこで,酵母P. pastorisを宿主として使用し,本酵素の生産に必要な条件を精査した.まず,ChuaHNL遺伝子のコドン最適化,およびタンパク質のジスルフィド結合の酸化還元を触媒するジスルフィドイソメラーゼ(protein disulfide isomerase:PDI)の影響を検討した.P. pastoris由来PpPDI, C. hualienensis由来ChuaPDI1,あるいはChuaPDI2遺伝子を共発現させた結果,コドン最適化されたChuaHNLとPpPDI遺伝子の共発現により,ChuaHNLの生産性は,単位培養あたり約140倍に増加した49).
5)種々のヤスデ由来HNLの探索,構造と利用
次に,国内産の多くのヤスデからHNLをコードするcDNAのクローニングを行った.その結果,キシャヤスデ[Parafontaria laminata(Attems)]由来HNL(PlamHNL)やヤケヤスデ[Oxidus gracilis(C.L.Koch)]由来HNL(OgraHNL)など10種類のヤスデHNL遺伝子をコードするcDNAのクローニングができた.これらのヤスデ由来HNLの一次構造の類似性は45~93%であり,バキュロウイルス-昆虫細胞発現系で遺伝子発現させることができ,すべてがHNL活性を示した.よって,HNLはヤスデ間で保存されていることが強く示唆された.これらのヤスデは,高い安定性と比活性を有するHNL遺伝子の大きな資源になることであろう.ヤスデ由来HNLの比活性は,一般的に植物由来HNLと比較すると5~230倍高かった50).
富山県内でキシャヤスデを採捕し,そのHNL(PlamHNL)を均一に精製し,部分構造からPlamHNLをコードする完全長cDNAをクローニングした.PlamHNL遺伝子は,20アミノ酸長のシグナルペプチドを含む183アミノ酸をコードする552塩基対で構成されていた.PlamHNLは,ChuaHNLとは異なり,大腸菌SHuffle T7株で一部可溶性に生産された.また,P. pastorisで遺伝子発現させ,グリコシル化された組換えPlamHNLの酵素化学的諸性質についても検討した.野生型PlamHNLは,種々のシアノヒドリンの合成において,他のヤスデ由来HNLより広い基質特異性を示し,抗血栓薬クロピドグレルの重要中間体(R)-2-クロロマンデロニトリルの合成(76%e.e.)を触媒する.そこで,PlamHNLをタンパク質工学により(R)-2-クロロマンデロニトリル生産に適するように改変するために,X線結晶構造解析を行った.PlamHNLの立体構造を決定し,MOEソフトウエアを使ってPlamHNLの活性部位での(R)-2-クロロマンデロニトリルとのドッキングシミュレーションを行い,変異型酵素N85Yを得た.N85Yではエナンチオ選択性が改善され,高い変換率(91%)により(R)-2-クロロマンデロニトリル合成(98.2%e.e.)が可能になった41).
引用文献References
1) Dadashipour, M. & Asano, Y. (2011) Hydroxynitrile lyases: Insights into biochemistry, discovery and engineering. ACS Catal., 1, 1121–1149.
2) 浅野泰久(2014)微生物・植物由来の新規ニトリル分解・合成酵素の産業利用.化学と生物,52, 651–658.
3) Bracco, P., Busch, H., von Langermann, J., & Hanefeld, U. (2016) Enantioselective synthesis of cyanohydrins catalysed by hydroxynitrile lyases—A review. Org. Biomol. Chem., 14, 6375–6389.
4) Effenberger, F. & Heid, S. (1995) (R)-Oxynitrilase catalyzed synthesis of (R)-ketone cyanohydrins. Tetrahedron Asymmetry, 6, 2945–2952.
5) Griengl, H., Klempier, N., Pöchlauer, P., Schmidt, M., Shi, N., & Zabelinskaja-Mackova, A.A. (1998) Enzyme catalysed formation of (S)-cyanohydrins derived from aldehydes and ketones in a biphasic solvent system. Tetrahedron, 54, 14477–14486.
6) Asano, Y., Tamura, K.i., Doi, N., Ueatrongchit, T., H-Kittikun, A., & Ohmiya, T. (2005) Screening for new hydroxynitrilases from plants. Biosci. Biotechnol. Biochem., 69, 2349–2357.
7) Nuylert, A., Ishida, Y., & Asano, Y. (2017) Effect of glycosylation on the biocatalytic properties of hydroxynitrile lyase from the passion fruit, Passiflora edulis—A comparison of natural and recombinant enzymes. ChemBioChem, 18, 257–265.
8) Isobe, K., Kitagawa, A., Kanamori, K., Kashiwagi, K., Matsui, D., Yamaguchi, T., Fuhshuku, K., Semba, H., & Asano, Y. (2018) Characterization of a novel hydroxynitrile lyase from Nandina domestica Thunb. Biosci. Biotechnol. Biochem., 82, 1760–1769.
9) Motojima, F., Nuylert, A., & Asano, Y. (2018) The crystal structure and catalytic mechanism of hydroxynitrile lyase from passion fruit, Passiflora edulis. FEBS J., 285, 313–324.
10) Dadashipour, M., Yamazaki, M., Momonoi, K., Tamura, K., Fuhshuku, K., Kanase, Y., Uchimura, E., Kaiyun, G., & Asano, Y. (2011) S-selective hydroxynitrile lyase from a plant Baliospermum montanum: Molecular characterization of recombinant enzyme. J. Biotechnol., 153, 100–110.
11) Nakano, S., Dadashipour, M., & Asano, Y. (2014) Structural and functional analysis of hydroxynitrile lyase from Baliospermum montanum with crystal structure, molecular dynamics and enzyme kinetics. Biochim. Biophys. Acta. Proteins Proteomics, 1844, 2059–2067.
12) Lanfranchi, E., Pavkov-Keller, T., Koehler, E.M., Diepold, M., Steiner, K., Darnhofer, B., Hartler, J., Van Den Bergh, T., Joosten, H.J., Gruber-Khadjawi, M., et al. (2017) Enzyme discovery beyond homology: A unique hydroxynitrile lyase in the Bet v1 superfamily. Sci. Rep., 7, 46738.
13) Hajnal, I., Lyskowski, A., Hanefeld, U., Gruber, K., Schwab, H., & Steiner, K. (2013) Biochemical and structural characterization of a novel bacterial manganese-dependent hydroxynitrile lyase. FEBS J., 280, 5815–5828.
14) 渡辺弘之(1978)土壌動物の世界.pp. 95–102, 東海大学出版会.
15) Eisner, T., Eisner, H.E., Hurst, J.J., Kafatos, F.C., & Meinwald, J. (1963) Cyanogenic glandular apparatus of a millipede. Science, 139, 1218–1220.
16) 新島渓子,有村利浩(2002)ヤンバルトサカヤスデによる列車妨害記録.Edaphologia, 69, 47–49.
17) Noguchi, S., Mori, N., Higa, Y., & Kuwahara, Y. (1997) Identification of mandelonitrile as a major secretory compound from Chamberlinius hualienensis Wang (Polydesmida: Paradoxosomatidae). Kandokon, 8, 208–214.
18) Kuwahara, Y., Shimizu, N., & Tanabe, T. (2011) Release of hydrogen cyanide via a post-secretion Schotten–Baumann reaction in defensive fluids of polydesmoid millipedes. J. Chem. Ecol., 37, 232–238.
19) Shear, W.A. (2015) The chemical defenses of millipedes (Diplopoda): Biochemistry, physiology and ecology. Biochem. Syst. Ecol., 61, 78–117.
20) Kuwahara, Y., Yamaguchi, T., Ichiki, Y., Tanabe, T., & Asano, Y. (2017) Hydrogen peroxide as a new defensive compound in “benzoyl cyanide” producing polydesmid millipedes. Naturwissenschaften, 104, 19.
21) Yamaguchi, T., Yamamoto, K., & Asano, Y. (2014) Identification and characterization of CYP79D16 and CYP71AN24 catalyzing the first and second steps in L-phenylalanine-derived cyanogenic glycoside biosynthesis in the Japanese apricot, Prunus mume Sieb. et Zucc. Plant Mol. Biol., 86, 215–223.
22) ERATO浅野酵素活性分子プロジェクト,最終報告書(2018), 82.
23) Yamaguchi, T., Kuwahara, Y., & Asano, Y. (2017) A novel cytochrome P450, CYP3201B1, is involved in (R)-mandelonitrile biosynthesis in a cyanogenic millipede. FEBS Open Bio, 7, 335–347.
24) Dadashipour, M., Ishida, Y., Yamamoto, K., & Asano, Y. (2015) Discovery and molecular and biocatalytic properties of hydroxynitrile lyase from an invasive millipede Chamberlinius hualienensis. Proc. Natl. Acad. Sci. USA, 112, 10605–10610.
25) Ishida, Y., Kuwahara, Y., Dadashipour, M., Ina, A., Yamaguchi, T., Morita, M., Ichiki, Y., & Asano, Y. (2016) A sacrificial millipede altruistically protects its swarm using a drone blood enzyme, mandelonitrile oxidase. Sci. Rep., 6, 26998.
26) Eisner, T., Alsop, D., Hicks, K., & Meinwald, J.(1978). Defensive secretions of millipedes. In Arthropod venoms (Handbook of Pharmacology No. 48), (ed. S. Bettini), pp. 41–72. Springer-Verlag, Berlin.
27) Halkier, B.A. & Møller, B.L. (1989) Biosynthesis of the cyanogenic glucoside dhurrin in seedlings of Sorghum bicolor (L.) Moench and partial purification of the enzyme system involved. Plant Physiol., 90, 1552–1559.
28) Fukuta, Y., Nanda, S., Kato, Y., Yurimoto, H., Sakai, Y., Komeda, H., & Asano, Y. (2011) Characterization of a new (R)-hydroxynitrile lyase from the Japanese apricot Prunus mume and cDNA cloning and secretory expression of one of the isozymes in Pichia pastoris. Biosci. Biotechnol. Biochem., 75, 214–220.
29) Cielo, C.B.C., Yamane, T., Asano, Y., Watanabe, N., Suzuki, A., & Fukuta, Y. Crystal structure of a native FAD-dependent hydroxynitrile lyase derived from the Japanese apricot, Prunus mume, unpublished. PDB:3RED
30) Nanda, S., Kato, Y., & Asano, Y. (2005) A new (R)-hydroxynitrile lyase from Prunus mume: Asymmetric synthesis of cyanohydrins. Tetrahedron, 61, 10908–10916.
31) Nanda, S., Kato, Y., & Asano, Y. (2006) PmHNL catalyzed synthesis of (R)-cyanohydrins derived from aliphatic aldehydes. Tetrahedron Asymmetry, 17, 735–741.
32) Ueatrongchit, T., Kayo, A., Komeda, H., Asano, Y., & H-Kittikun, A. (2008) Purification and characterization of a novel (R)-hydroxynitrile lyase from Eriobotrya japonica (Loquat). Biosci. Biotechnol. Biochem., 72, 1513–1522.
33) Ueatrongchit, T., Tamura, K., Ohmiya, T., H-Kittikun, A., & Asano, Y. (2010) Hydroxynitrile lyase from Passiflora edulis: Purification, characteristics and application in asymmetric synthesis of (R)-mandelonitrile. Enzyme Microb. Technol., 46, 456–465.
34) Glieder, A., Weis, R., Skranc, W., Pöchlauer, P., Dreveny, I., Majer, S., Wubbolts, M., Schwab, H., & Gruber, K. (2003) Comprehensive step-by-step engineering of an (R)-hydroxynitrile lyase for large-scale asymmetric synthesis. Angew. Chem. Int. Ed., 42, 4815–4818.
35) Motojima, F., Izumi, A., Nuylert, A., Zhai, Z., Dadashipour, M., Shichida, S., Yamaguchi, T., Nakano, S., & Asano, Y. (2021) R-hydroxynitrile lyase from the cyanogenic millipede, Chamberlinius hualienensis—A new entry to the carrier protein family lipocalines. FEBS J., 288, 1679–1695.
36) Dreveny, I., Gruber, K., Glieder, A., Thompson, A., & Kratky, C. (2001) The hydroxynitrile lyase from almond: A lyase that looks like an oxidoreductase. Structure, 9, 803–815.
37) Lauble, H., Miehlich, B., Förster, S., Wajant, H., & Effenberger, F. (2002) Crystal structure of hydroxynitrile Lyase from Sorghum bicolor in complex with the inhibitor benzoic acid: A novel cyanogenic enzyme. Biochemistry, 41, 12043–12050.
38) Guterl, J.K., Andexer, J.N., Sehl, T., von Langermann, J., Frindi-Wosch, I., Rosenkranz, T., Fitter, J., Gruber, K., Kragl, U., Eggert, T., et al. (2009) Uneven twins: Comparison of two enantiocomplementary hydroxynitrile lyases with alpha/beta-hydrolase fold. J. Biotechnol., 141, 166–173.
39) Wiedner, R., Gruber-Khadjawi, M., Schwab, H., & Steiner, K. (2014) Discovery of a novel (R)-selective bacterial hydroxynitrile lyase from Acidobacterium capsulatum. Comput. Struct. Biotechnol. J., 10, 58–62.
40) Zheng, D.-J., Nakabayashi, M., & Asano, Y. (2022) Structural characterization of Linum usitatissimum hydroxynitrile lyase: A new cyanohydrin decomposition mechanism involving a cyano-zinc complex. J. Biol. Chem., 298, 101650.
41) Nuylert, A., Nakabayashi, M., Yamaguchi, T., & Asano, Y. (2020) Discovery and structural analysis to improve the enantioselectivity of hydroxynitrile lyase from Parafontaria laminata millipedes for (R)-2-chloromandelonitrile synthesis. ACS Omega, 5, 27896–27908.
42) Pavkov-Keller, T., Bakhuis, J., Steinkellner, G., Jolink, F., Keijmel, E., Birner-Gruenberger, R., & Gruber, K. (2016) Structures of almond hydroxynitrile lyase isoenzyme 5 provide a rationale for the lack of oxidoreductase activity in flavin dependent HNLs. J. Biotechnol., 235, 24–31.
43) Flower, D.R. (1996) The lipocalin protein family: Structure and function. Biochem. J., 318, 1–14.
44) Lakshmi, B., Mishra, M., Srinivasan, N., & Archunan, G. (2015) Structure-based phylogenetic analysis of the lipocalin superfamily. PLoS One, 10, e0135507.
45) Grzyb, J., Latowski, D., & Strzałka, K. (2006) Lipocalins—A family portrait. J. Plant Physiol., 163, 895–915.
46) Shimamoto, S., Yoshida, T., Inui, T., Gohda, K., Kobayashi, Y., Fujimori, K., Tsurumura, T., Aritake, K., Urade, Y., & Ohkubo, T. (2007) NMR solution structure of lipocalin-type prostaglandin D synthase: Evidence for partial overlapping of catalytic pocket and retinoic acid-binding pocket within the central cavity. J. Biol. Chem., 282, 31373–31379.
47) Kumasaka, T., Aritake, K., Ago, H., Irikura, D., Tsurumura, T., Yamamoto, M., Miyano, M., Urade, Y., & Hayaishi, O. (2009) Structural basis of the catalytic mechanism operating in open-closed conformers of lipocalin type prostaglandin D synthase. J. Biol. Chem., 284, 22344–22352.
48) Elmes, M.W., Volpe, A.D., d’Oelsnitz, S., Sweeney, J.M., & Kaczocha, M. (2018) Lipocalin-type prostaglandin D synthase is a novel phytocannabinoid-binding protein. Lipids, 53, 353–360.
49) Zhai, Z., Nuylert, A., Isobe, K., & Asano, Y. (2019) Effects of codon optimization and glycosylation on the high-level production of hydroxynitrile lyase from Chamberlinius hualienensis in Pichia pastoris. J. Ind. Microbiol. Biotechnol., 46, 887–898.
50) Yamaguchi, T., Nuylert, A., Ina, A., Tanabe, T., & Asano, Y. (2018) Hydroxynitrile lyases from cyanogenic millipedes: Molecular cloning, heterologous expression, and whole-cell biocatalysis for the production of (R)-mandelonitrile. Sci. Rep., 8, 3051.