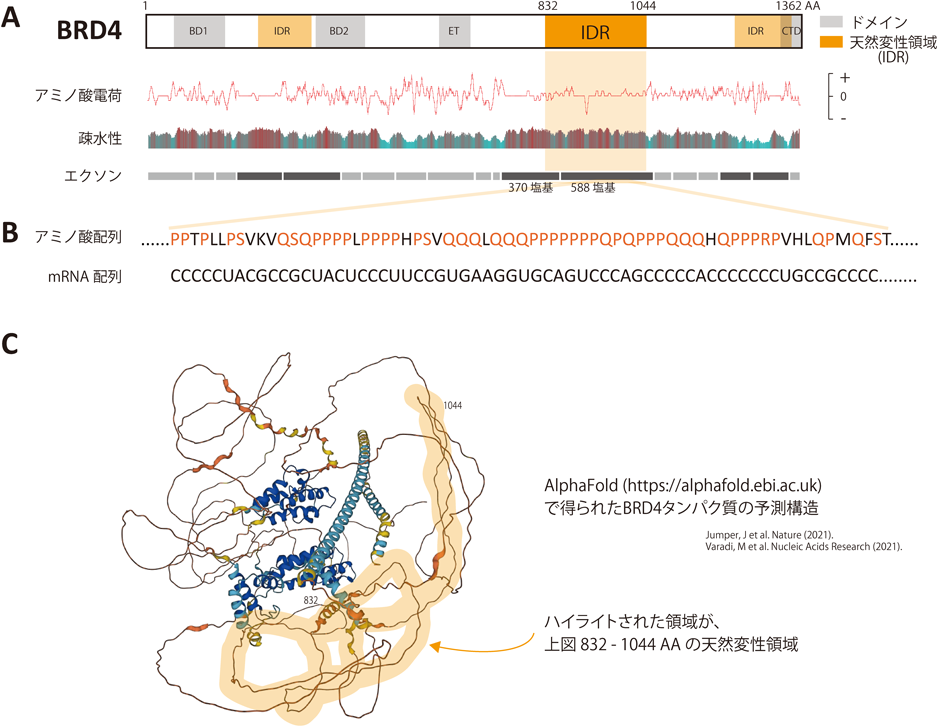
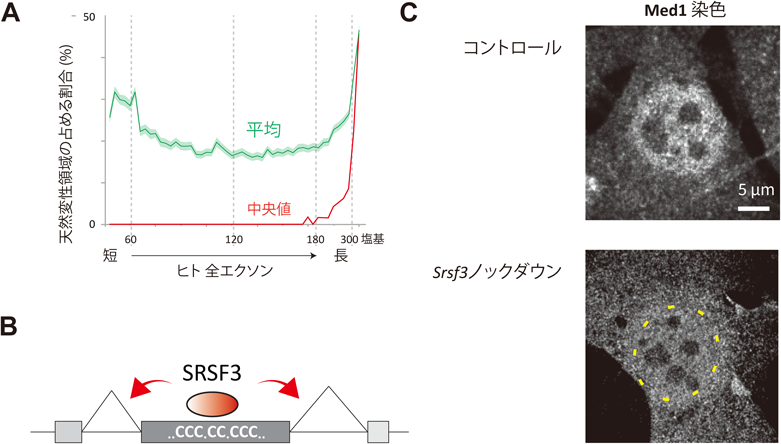
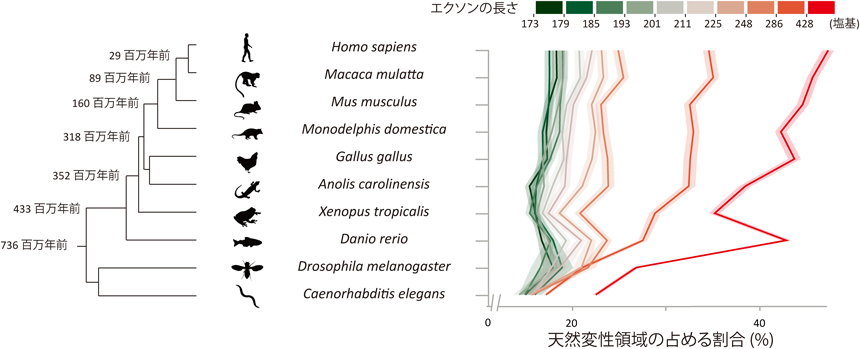
長いエクソン群が支える液–液相分離転写ネットワークLiquid–liquid phase separation of transcription factors are sustained by large exons in vertebrates
名古屋大学大学院医学系研究科先端応用医学部門神経遺伝情報分野Division of Neurogenetics, Center for Neurological Diseases and Cancer, Nagoya University Graduate School of Medicine ◇ 〒466–8550 愛知県名古屋市昭和区鶴舞町65 ◇ 65 Tsurumai, Showa-ku, Nagoya, Aichi 466–8550, Japan