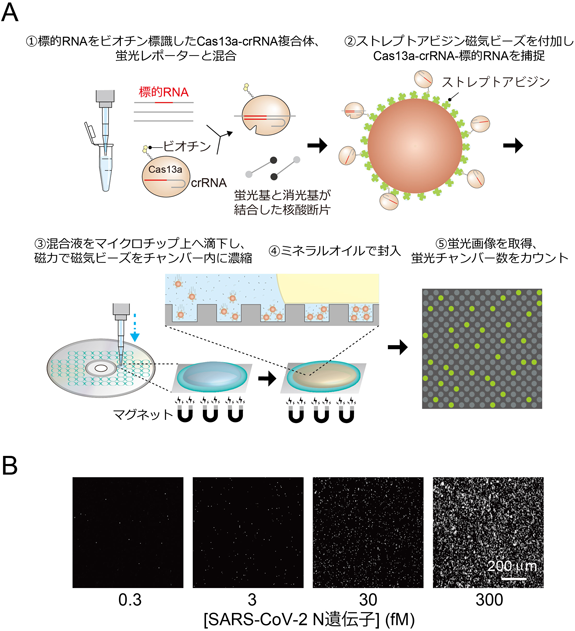
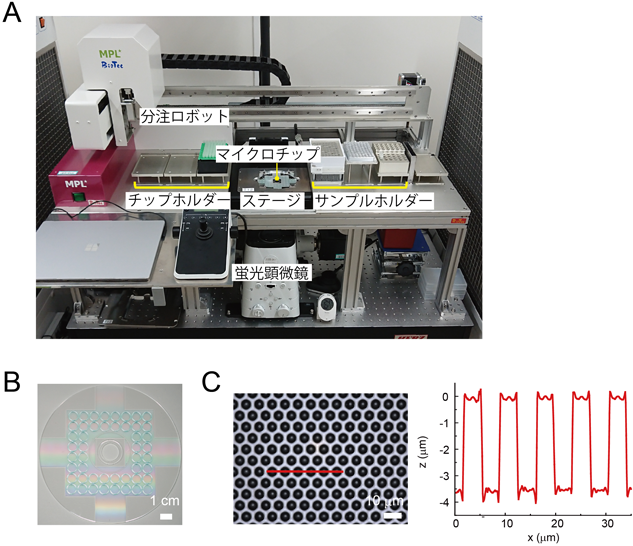
一分子定量法に基づいたRNAの全自動迅速検出装置の開発と感染症診断への展開Development of an automated platform for rapid RNA detection at the single molecule level and its application to the diagnosis of infectious diseases
理化学研究所開拓研究本部渡邉分子生理学研究室Molecular Physiology Laboratory, Cluster for Pioneering Research, RIKEN ◇ 〒351–0198 埼玉県和光市広沢2–1 生物科学研究棟S252 ◇ Molecular Physiology Lab, 2–1 Hirosawa, Wako, Saitama 351–0198, Japan