まずスプライシング反応の概要について説明する.
1)mRNA上に存在するスプライシングに必須なシグナルとスプライシング反応
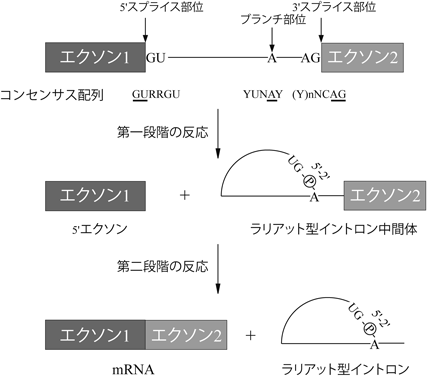
高等真核生物では,スプライシングはタンパク質をコードする遺伝子の発現に必須な過程である1, 2).スプライシングには大きく分けて二つの様式があるが,mRNA上のすべてのエクソンを包含する様式を恒常的スプライシング,いくつかのエクソンの使用を使い分け,1種類のmRNA前駆体から複数種のmRNAを産生する様式を選択的スプライシングと呼ぶ1, 2).どちらの様式でも,スプライシング反応に必要な配列は主にイントロン上に存在する(図1).まず,イントロンの5′末端に,5′スプライス部位と呼ばれる配列が存在し,そのコンセンサス配列はGURAGU[Rはプリン残基(AまたはG)]である.3′末端にもCAGというコンセンサス配列を持つ3′スプライス部位が存在する.3′スプライス部位の上流には,ピリミジン残基(CまたはU)が複数個連続する配列があり,ピリミジントラクトと呼ばれている(図1).また,このピリミジントラクトの上流に,ブランチ部位と呼ばれる配列が存在し,ほとんどの場合,A残基である(図1).ピリミジントラクトはブランチ部位の認識と3′スプライス部位の認識の両方を補助する.
スプライシング反応は2段階の反応からなる.まず,第一段階の反応では,5′スプライス部位での切断が起こり,イントロンの5′末端のGとブランチ部位のAとの間で2′–5′ホスホジエステル結合が形成される(図1).その後,3′スプライス部位での切断が起きてエクソンどうしが連結され,mRNAとなる.イントロンが複数存在する場合,この過程を繰り返し,恒常的スプライシングではすべてのエクソンがつなぎ合わされたmRNAが産生される(図1).切り出されたイントロンは,ラリアット(投げ縄)構造をとっており,mRNAから解離した後は核内にとどまり分解される(図1).一方mRNAは核外へと輸送され,タンパク質翻訳の鋳型となる.
2)スプライソソーム形成とスプライシング反応
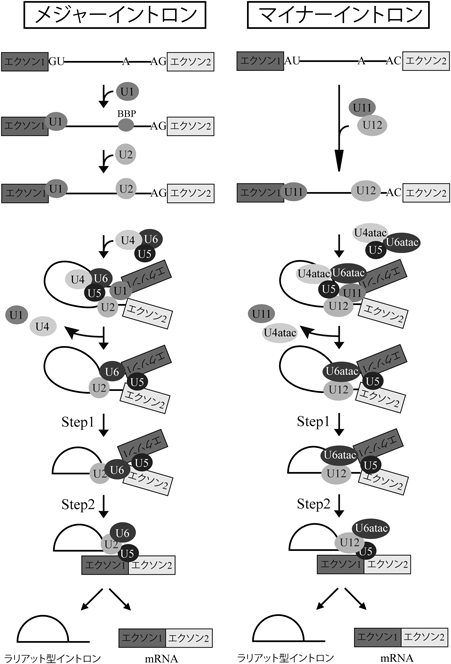
上記のスプライシング反応は,スプライソソームと呼ばれる,リボソームに匹敵する巨大な複合体中で起きる1, 2).スプライソソーム形成は段階的に起きることが知られている(図2).まず5′スプライス部位がuridine-rich small nuclear ribonucleoprotein(U snRNP)の一つであるU1 snRNPによって認識される.ブランチ部位はbranch point binding protein(BBP)によって認識され,3′スプライス部位およびピリミジントラクトはU2 snRNP auxiliary factor(U2AF)によって認識される.その後,ブランチ部位はU2 snRNPが認識し,その複合体にU4/U6, U5 snRNP複合体が会合して成熟したスプライソソームとなる.そしてU1, U4 snRNPの解離を経て,スプライシング反応が進行し,最終的にmRNAとラリアット型イントロンが産生される(図2).
上述のスプライソソーム形成は,イントロンの両端の配列がGUとAGであるイントロンで起きる反応である.ヒトではほとんどのイントロンがGUで始まってAGで終わる.それらのイントロンは,ゲノム上ではGTとAGのためGTAGイントロン,またはメジャーイントロンと呼ばれる.それに対し,ヒトではわずか0.4%程度しか存在しないが,ATAC(アタック)イントロンまたはマイナーイントロンと呼ばれるイントロンが存在する.マイナーイントロンの両端の配列はAUとACになっている(ゲノム上ではATとAC)場合が多く,それゆえにATACイントロンとも呼ばれている(図2).このイントロンのスプライシングには,まずU11/U12 snRNP複合体が関わる.U11は5′スプライス部位を,U12はブランチ部位をそれぞれ認識する(図2).そして,U4/U6 snRNPの代わりに,U4atac/U6atac snRNPと呼ばれる複合体が関与する.興味深いことに,U5 snRNPだけは,メジャーイントロンと共通である(図2).
3)エクソン認識とそこに関わるシス制御配列およびトランス制御因子
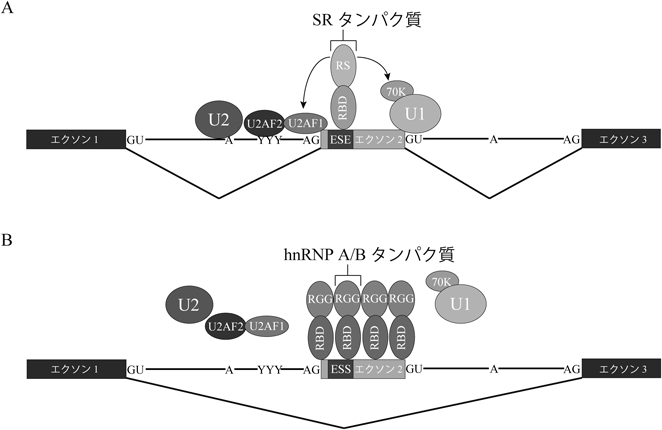
脊椎動物においては,イントロンの平均長は下等な真核生物に比べてずっと長い.しかしエクソンの平均長にはそれほど大きな違いはない.そこで,イントロンの認識よりも,エクソンの認識が重要であると考えられている.このモデルはエクソン認識モデルと呼ばれているが,このモデルの根拠としては,1)5′末端と3′末端のエクソンを除いたエクソンは300塩基長を超えないこと,2)ヒトの疾患では5′スプライス部位に変異が入った場合,そのイントロンのスプライシングのみが阻害されるのではなく,その上流のエクソンが飛ばされることが多い,ということがある.エクソン認識モデルでは,対応するエクソンの上流側イントロン領域のブランチ部位,ピリミジントラクト,3′スプライス部位,下流のイントロン領域の5′スプライス部位が重要な役割を果たす.ブランチ部位にはU2 snRNPが結合し,ピリミジントラクトと3′スプライス部位はそれぞれU2AF2, U2AF1が認識している.また,エクソン下流の5′スプライス部位にはU1 snRNPが結合している.これらのイントロン内の要素に加えエクソンの中には,エクソニックスプライシングエンハンサー(exonic splicing enhancer:ESE)と呼ばれる配列が存在する場合がある(図3).ESEには主にセリン-アルギニンリッチ(serine-arginine-rich:SR)タンパク質が結合し,エクソン認識を促進する.SRタンパク質は,アミノ末端に一つまたは二つのRNA結合ドメイン(RBD),カルボキシ末端にアルギニン-セリンリッチ(RS)ドメインを持つという共通の特徴を持つ,ヒトでは12種類知られているファミリータンパク質である2–4).RSドメインは,他のRSドメインを含むタンパク質とのタンパク質間相互作用をつかさどる.前述のU2AF1, U2AF2,そしてU1 snRNPの構成要素であるU1-70KもRSドメインを持っている.SRファミリータンパク質はそのRBDによってESEと結合して,RSドメインを通じてU2AFおよびU1-70Kと相互作用し,エクソンを挟んで3′スプライス部位と5′スプライス部位の橋渡しをしている(図3).このクロストークにより,ESEを持つエクソンの認識が促進される.これに対して,エクソンの認識を低下させる制御配列も存在する.この制御配列はエクソニックスプライシングサイレンサー(exonic splicing silencer:ESS)と呼ばれる(図3).ESSは別のスプライシング制御因子heterogeneous nuclear ribonucleoprotein(hnRNP)ファミリータンパク質と結合している.hnRNPタンパク質ファミリーはA1からUまでの20種類のタンパク質からなり,転写,スプライシング,RNAの輸送・局在化,翻訳,RNAの安定性など,多くの細胞機能を持つ核内タンパク質である5).hnRNPファミリータンパク質の一つであるhnRNP A1は,エクソン内のESSに結合し,さらにhnRNP A1がエクソンの上流および下流領域と結合することにより,ESEやスプライス部位領域を覆い隠すことでU1 snRNPやU2AFの結合が阻害され,サイレンシングが行われる(図3).この結果,エクソンの認識が阻害される.
3. 骨髄異形成症候群におけるスプライシング因子の変異とそれに伴う異常スプライシング
前節で述べたシス制御配列やトランス制御因子の変異が異常なスプライシングパターンを引き起こし,しばしばヒトに疾患をもたらす.このようなRNAの代謝過程に異常がある病気は「RNA病」とも呼ばれる.その一つである骨髄異形成症候群(MDS)において,スプライシング因子が頻繁に変異していることが明らかになった.MDSは慢性骨髄性腫瘍の一種で,造血,末梢血細胞減少,急性骨髄性白血病への進行の高いリスクなどの多くの症状を特徴とする一群の疾患である6).患者のゲノムDNAを次世代シーケンサーで解析した結果,SF3B1, U2AF1, SRSF2,およびZRSR2が最も頻繁に変異しているスプライシング因子であることが判明した7–9).SF3B1, U2AF1, SRSF2は,多くの患者で変異した特定のアミノ酸残基を持つことから,これらの変異は「機能獲得型」変異であると考えられている.一方,ZRSR2遺伝子には多くの異なる変異が見つかっており,これらの変異は「機能喪失」を引き起こすことが示唆されている.次項では,上記四つのスプライシング因子を中心に,それらの変異が引き起こす異常なスプライシング機構を紹介し,MDSの発症の原因についてスプライシングの観点から考察する.
1)Splicing Factor 3B1(SF3B1)
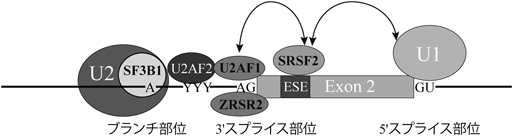
SF3B1は,スプライシングの際にU2 snRNPのブランチ部位への結合を安定化させるSF3B複合体の構成要素の一つである(図4)10).SF3B1遺伝子は染色体2q33.1上に位置しており,MDS患者の約20~28%はSF3B1の変異を有している8, 11–13).驚くべきことに,SF3B1変異は,98%の患者において環状鉄芽球(ring sideroblast:RS)表現型の原因となっている12).またNOD scid gamma(NSG)マウスを用いた研究において,SF3B1変異MDS-RS患者の造血幹細胞(HSC)を移植したマウスは,特徴的な環状鉄芽球を形成することが報告されている14).MDS患者におけるSF3B1遺伝子変異は,HEATドメインに存在する700番目のリシンがグルタミンに変化した変異が多くみられ,「ホットスポット」としてクラスターを形成している.他のホットスポット(R625, H662, K666)もHEATドメイン内で空間的に近接しているため,同様の機能的影響を持つと推測される13, 15).SF3B複合体はブランチ部位配列の認識に関与していることから,この変異は本来とは異なるブランチ部位配列と3′スプライス部位の利用を引き起こす可能性が高い.そのような異常なスプライシングは,しばしばmRNA上に終止コドンを生じ,nonsense-mediated decay(NMD)によるmRNAの分解が起こる.RNAシークエンス解析により,SF3B1変異体では多くの遺伝子の制御異常が検出された.その中から特定の標的遺伝子を同定し,その機能を明らかにすることは,創薬ターゲットの探索のために非常に重要である.創薬候補としては,SF3B1阻害剤が前臨床試験で白血病の前段階やそれに関連する骨髄系疾患の治療薬となる可能性が示唆されている.SF3B1はスプライシングに必須であることから,SF3B1阻害剤は,すべてのイントロンのスプライシング反応を阻害すると予想された.しかし,スプライソスタチンA(Spliceostatin A:SSA)によるスプライシングの阻害は培養細胞では部分的であり,腫瘍細胞では短いタンパク質に翻訳される転写物が産生される16, 17).もう一つの化合物であるプラジエノライドBは,抗腫瘍マクロライドであり,SF3Bと相互作用してスプライシングを阻害することが見いだされた18).また,プラジエノライドDの誘導体であるE7107は,強い抗腫瘍活性を示し,U2 snRNPとスプライソソームの強固な結合を阻害することで,スプライソソームの形成を阻害することが明らかになっている17).変異を持ったSF3B1と強く結合し阻害する低分子化合物を設計することは,MDSだけではなく,同様に変異がみられる乳がん,肺腺がんなどの他のがんの治療薬候補を見つけることにつながると期待される.
2)serine-arginine rich splicing factor 2(SRSF2)
SRSF2はもともとSC35と呼ばれ,SRタンパク質ファミリーのメンバーであり,恒常的スプライシングと選択的スプライシングの両方に関与している19).SRSF2遺伝子は染色体17q25.2に位置し,その変異はMDS患者の約14%で発見されている20).SRSF2にはRNA結合のためのRBDと他のタンパク質と相互作用するためのRSドメインがあり,そのRBDを介してmRNA前駆体のESEに結合し,エクソン認識を促進する(図4).MDS患者においての変異は95番目のプロリン残基に「ホットスポット」として集積している9, 21).MDSにおけるSF3B1の変異と同様に,これらのホットスポット変異は変異体特有の機能獲得を引き起こすと仮定された.実際,SRSF2変異によるスプライシングパターンの変化は,培養細胞,マウスモデル,ヒト患者サンプルで報告されている21–24).このプロリン残基は,RNA結合ドメインのわずかに外側に存在するため,MDSの変異はRNA結合には影響を与えないと考えられていた.しかし,野生型と変異型SRSF2タンパク質の結合配列を比較した結果,変異型SRSF2タンパク質は,野生型が効率よく結合するプリンに富んだ配列に加えて,GGNGおよびCCNGモチーフ(Cはシトシン,Gはグアニン,Nは任意の塩基)に対して高い結合親和性を示した21–24).この親和性の変化により,多くの遺伝子のスプライシングが変化するが,その中にはMDSの原因遺伝子の一つであるenhancer of Zeste homolog 2(EZH2)も含まれていた.EZH2はSETドメインを持つヒストンメチルトランスフェラーゼでPolycomb repressive complex 2(PRC2)の構成要素である.PRC2はヒストンH3のLys 27のトリメチル化(H3K27me3)を触媒し,遺伝子発現を制御している.変異型SRSF2におけるEZH2の異常スプライシングは,終止コドンを含むエクソン9.5を含むため,EZH2のタンパク質レベルが低下し遺伝子発現制御が正常に行われない.このことがMDS発症につながっていると考えられる.また,SRSF2とU2AF1におけるMDSでみられる変異が,Rループ構造の拡大を引き起こすことも明らかにされている25, 26).Rループ構造とは,RNAポリメラーゼによる転写が停止するのに伴い,二本鎖DNAのうちの鋳型鎖が,転写産物であるRNAとDNA:RNAハイブリッドを形成し,非鋳型鎖が一本鎖の状態で存在する構造のことである.Rループ構造形成の拡大は,DNA損傷応答経路の活性化につながる.MDSにおいてRループ構造が効率的に形成されるのは,スプライシング反応中または反応後のmRNA-タンパク質複合体の再編成を遅くすることによって行われるのかもしれない.Rループ構造の形成や解消にスプライシング因子がどのように関与しているのかは,非常に興味深い.
3)U2 snRNP auxiliary factor 1(U2AF1)
U2AF1は,もともとU2 snRNP auxiliary factor(U2AF)の構成要素として同定され,U2 snRNPのブランチ部位への結合を促進する(図4).U2AF1は,U2AFヘテロ二量体の小さい方のサブユニットで,3′スプライス部位であるAGジヌクレオチドを認識する役割を担っている(図4).もう一つの大きい方のサブユニットであるU2AF2は,ブランチ部位と3′スプライス部位の間に存在するピリミジントラクトを認識する(図4).U2AF1とU2AF2はともにRSドメインを持つため,エクソン認識において,同じくRSドメインを持つU1-70Kタンパク質と相互作用し,3′スプライス部位と下流の5′スプライス部位との間の相互作用をつかさどる.U2AF1遺伝子は21q22.3に位置し,その変異はMDS患者の約7~11%にみられ,保存されたジンクフィンガードメインに位置する34番目のセリンと157番目のグルタミンに「ホットスポット」として存在している9, 11, 13, 27).U2AF1のS34とQ157の両変異は,RNA結合を通じてスプライシングに影響を与えることが示されているが,3′スプライス部位認識に対する影響は異なる.U2AF1のS34変異体は,3′スプライス部位の配列としてCAGまたはAAGを持つ異常エクソンの使用を促進する傾向があることが示された28–34).一方,Q157変異体では,3′スプライス部位の1塩基下流がGである場合,エクソンの包含を促進する30).また,U2AF1のS34変異体がInterleukin-1 receptor-associated kinase 4(IRAK4)遺伝子のエクソン4の包含を誘導することが報告されている35).IRAK4は,Toll-like receptor(TLR)スーパーファミリーの下流のシグナル伝達を介し,NF-κBとMAPK経路を活性化する.IRAK4は,エクソン4の有無により,IRAK4-LとIRAK4-Sという二つのスプライシングアイソフォームに分けられる.IRAK4-Sは正常な造血器官における自然免疫応答を制御する一方,IRAK4-LはNF-κBの活性化を通して,悪性造血細胞における自然免疫反応の制御不能をもたらす.また,IRAK4-Lは乳がんや大腸がん細胞でも高発現しており,がん原性との関連が示唆されている.さらに,変異型U2AF1(S34F)AML細胞はIRAK4-Lへの依存性を獲得しており,IRAK4阻害剤に感受性があるため,治療法の標的となっている.最近,酵母U2AF1の結晶構造解析から,3′スプライス部位であるAGジヌクレオチドが,二つのZnフィンガードメインによって強く認識されることが明らかになった35).そしてU2AFヘテロ二量体(U2AF1+U2AF2)は,ピリミジンの連続が少ない弱いピリミジントラクトを認識する際に,閉じたU2AF2と開いたU2AF2の混合物として結合することがわかり,U2AF1のS34F変異は,U2AF2の開閉型コンホメーション間のシフトを調節することも明らかになった36).このことから,ZnフィンガードメインのRNA結合活性を阻害または修正するために,変異したZnフィンガードメインをターゲットとする化合物を設計することが,MDSの治療法として役立つ可能性があることを示している.
4)zinc finger CCCH-type, RNA binding motif and serine/arginine rich 2(ZRSR2)
ZRSR2遺伝子は染色体Xp22.2に位置し,MDS患者の約5%で変異している9).このタンパク質のスプライシングにおける機能については,ほとんど知られていないが,U2AFと相互作用する因子として単離された.そしてin vitroスプライシング反応系において,メジャー,マイナー両方のイントロンの3′スプライス部位の認識に関わることが示された(図4)37).MDS患者にみられるZRSR2の変異は,フレームシフトの挿入,欠失,ナンセンス,ミスセンスであることから,機能喪失変異である可能性が高いと考えられている.また,ZRSR2変異の患者では,マイナーイントロンのイントロン保持が起きている.ヒトでは800個ほどのマイナーイントロンが存在するが,E2F転写因子を含むいくつかの遺伝子やmitogen-activated protein kinase(MAPK)経路のいくつかの遺伝子のマイナーイントロンが,ZRSR2変異の患者サンプルにおいて異常なスプライシングを示した.最近では,ZRSR2タンパク質のノックアウトによるマイナーイントロン除去の障害が,造血幹細胞の自己複製を促進することが明らかになり,イントロンの変異ががんのドライバーとなる可能性が示唆されている38).しかし,ZRSR2の変異がマイナーイントロンのスプライシングに影響を与える正確な分子メカニズムはまだ解明されていない.その分子機構を解明することは,マイナーイントロンスプライシングの基礎知識だけではなく,ZRSR2変異を有するMDSの標的分子の同定につながることが期待される.
5)MDSにおけるその他のスプライシング因子の変異
MDS患者では,上記四つのスプライシング因子に加え,他のいくつかのスプライシング因子にも,まれに変異が確認された.その一つはpre-mRNA processing factor 8(PRPF8)である.酵母のPrp8タンパク質の研究から,PRPF8タンパク質はスプライシングに必須な因子であり,U5 snRNAと相互作用してスプライソソームにおいて5′と3′のスプライス部位の位置合わせを行っていることが明らかになっている39).MDSにおけるPRPF8タンパク質の変異は異常スプライシングを引き起こすが,これはスプライス部位の選択が変化したためである可能性が非常に高い.しかしPRPF8変異がどのようにエクソン認識に影響を与えるかについては,今後解析が必要である.別の因子はLUC7 like 2(LUC7L2)である.LUC7L2はスプライシング因子lethal unless cap-binding complex is produced(LUC7)のオルソログであり,U1 snRNPと連携して,コンセンサス配列から外れた5′スプライス部位の認識に関与していると考えられている40).他にもRNAヘリカーゼタンパク質の一つであるDEAD-box helicase 41(DDX41)は,MDSとAMLで変異していることが示された41).いくつかのRNAヘリカーゼはATP依存性のヘリカーゼ活性を持ち,スプライソソームの構造変化を引き起こすことで,スプライシングに関与することが知られている.実際,最も一般的な変異はR525H変異であり,これはATP結合に影響を及ぼし,結果的にヘリカーゼ活性を発揮できないと考えられている.さらに,他のスプライシング因子にも非常にまれな頻度で変異が見つかっているが42),ここまでに報告されている変異を持つスプライシング因子は,主にブランチ部位および3′スプライス部位の認識に関与している.これらの変異がスプライシングにどのような影響を与えるかを調べることは,MDSの病因だけでなく,基本的なスプライシング機構の解明にもつながると期待される.
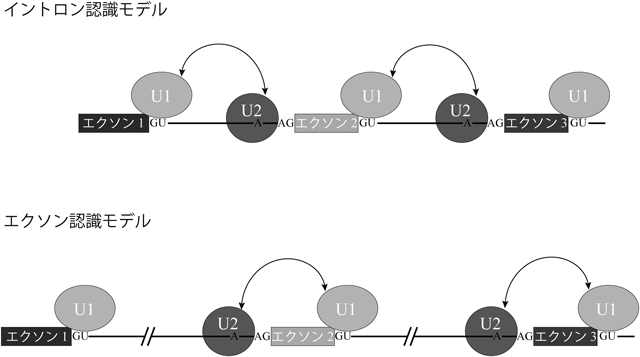
本稿では,MDSで変異した四つの主要なスプライシング因子について,その変異と異常スプライシングを紹介した.注目すべきことに,本稿で紹介したタンパク質のほとんどは3′スプライス部位の認識に関与している(図4).さらに,SRSF2はESEとの結合を通じてエクソン認識にも関与している.これらを総合すると,MDSの異常スプライシングには,エクソン認識と呼ばれるスプライシングモードが関与していると考えられる(図5)43).高等真核生物では,イントロンの平均長は下等真核生物のそれよりもはるかに長い.実際,ヒトのイントロンの平均長は5849ヌクレオチドであるのに対し,線虫のイントロンの平均長は335ヌクレオチドである.一方,5′末端と3′末端のエクソンを除いた内部のエクソンの平均長は300ヌクレオチド以下であり,脊椎動物と下等真核生物の間で差はない.したがって,イントロンのサイズが大きい脊椎動物では,エクソン認識がスプライシングの主要な様式である可能性が高い.一方,イントロンが比較的短い下等真核生物では,イントロン認識が支配的であると考えられる(図5).5′スプライス部位の変異が隣接するエクソンをスキップさせ,ヒトの病気を引き起こすという事実も,エクソン認識モデルを支持している.5′末端エクソンでは,キャップ構造が3′スプライス部位の代用として機能していると考えられる.一方3′末端エクソンに関しては,ポリ(A)付加シグナルとポリ(A)付加複合体が5′スプライス部位の代わりを果たしていると思われる.エクソン認識モデルでは,3′スプライス部位の定義が最初に行われる可能性が高く,この段階はエクソン認識に重要である.脊椎動物のエクソン認識機構については,これまでに多くの優れた研究成果があるが,エクソンによって異なる因子や機構が関与しているかは不明である.MDSにおける異常スプライシング機構の解析から,この機構に関するさらなる知見が得られると期待される.
これまでに述べたように,スプライシング因子変異による異常スプライシングがMDS発症に関与していることが確認されているが,異なるスプライシング因子の変異が,どのように異なるMDS表現型を引き起こすかについては明らかになっておらず,スプライシング異常がMDS発症の原因となる共通遺伝子も見つかっていない.しかしこれまでの解析から,スプライシング因子の変異によって影響を受ける共通の経路は,エピジェネティクスとシグナル伝達経路であることが示唆された.スプライシング異常の機構解析からのアプローチで得られた知見は,MDSの治療研究において,創薬ターゲットの同定につながることが期待される.今後のさらなる研究の進展が待たれる.