ヒトを含む真核生物の遺伝子は,イントロンによって細かく分断されている.当然のことながら,mRNA前駆体から正確にイントロンを取り除くmRNA前駆体スプライシング(以下スプライシング)は,正常な遺伝子発現に必須な過程である.スプライシング反応は非常に複雑で,スプライソソームと呼ばれる巨大なタンパク質–RNA複合体が動的に変化しながら遂行される1, 2).スプライソソームを構成する170種類以上のタンパク質因子とsnRNA(核内低分子RNA)は,真核生物種間でよく保存されている.ところが,その基質となるmRNA前駆体のイントロンの長さ分布は驚くほど広い.面白いことに,真核生物が高等になればなるほど,イントロンは長くなっていく(表1).イントロン長の分布をみると,分裂酵母では35~817塩基,ほとんどのイントロンは100塩基以下である3).一方,最も高等なヒトでは,30~116万塩基,その長さの比は4万倍近くにも達する4).ところが,すべての生物の翻訳エクソンの長さの平均は206塩基であり,ほとんどのエクソンは856塩基以内に収まってしまう4).
表1 真核生物のイントロン長の平均値| 真核生物種 | 平均イントロン長(塩基) |
|---|
| 分裂酵母(S. pombe) | 107 |
| シロイヌナズナ(A. thaliana) | 169 |
| 線虫(C. elegans) | 324 |
| ショウジョウバエ(D. melanogaster) | 1657 |
| ゼブラフィッシュ(D. rerio) | 2281 |
| マウス(M. musculus) | 5622 |
| ヒト(H. sapiens) | 7386 |
なぜ,高等真核生物のイントロンは長くなっていったのか? 最も端的な回答は,(i)イントロンが長くなることによって,イントロン内に核小体内低分子RNA(snoRNA)やマイクロRNA(miRNA)といった機能的な非翻訳RNA遺伝子を宿すことができたこと,(ii)長くなったイントロンでは,より複雑な選択的スプライシング制御が可能となり,多種多様な遺伝子発現を実現することができたこと,(iii)タンパク質をコードしない長いイントロンの存在は,細胞分裂における頻繁な交差に有利となったこと,以上である.まさに,長いイントロンは生物進化を加速させていく大きな原動力になったと考えられる5).すこぶる興味深いことに,長いイントロンを有する巨大な遺伝子は,他のいかなる臓器よりも脳で多く発現している6).精神活動という最も高次な表現型に,ひょっとすると長いイントロンが一役買っているのだろうか?
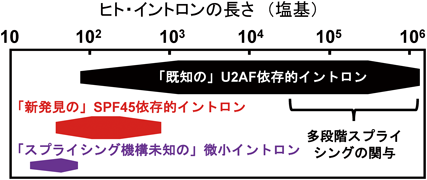
進化とともにイントロン長が長くなり,結果的に高等真核生物の遺伝子には,さまざまな長さのイントロンが混在するようになった.当然のことながら,mRNAはタンパク質の設計図であるから,どんな長さのイントロンであっても一寸の狂いもなくスプライシングにより取り除かれなければならない(実際,正確無比に取り除かれている).よって,長さに極端な差があるイントロンを持つ高等真核生物では,イントロンの長さを識別し,さまざまな長さのイントロン除去を可能にする巧みなスプライシング機構が存在するに違いない.本稿ではイントロンの「長さ」に対応したスプライシング機構について考察しよう.
2. スプライシングされるべきイントロンとエクソンの認識
スプライシングの分子機構は,主に酵母を用いた遺伝学的解析と,培養細胞の核抽出液を用いたin vitroスプライシング系による生化学的解析の互いの長所を組み合わせて解明されてきた.その研究に使われたmRNA前駆体基質は,in vitroスプライシング系で効率よくスプライシングされる適度な長さのイントロンが用いられてきた,という事実に留意しておく必要がある.実際に,多くの研究室で頻繁に使われてきたmRNA前駆体は,ヒトβグロビン遺伝子の第1イントロン(158塩基長)と,アデノウイルス主要後期転写単位の第1イントロン(231塩基長)である.すなわち,以下に説明する,現在の分子生物学の教科書に記載されているスプライシングの分子機構は,正確には,その「適度な長さのイントロン」に対応した一つの代表的な分子機構である,というべきなのである.
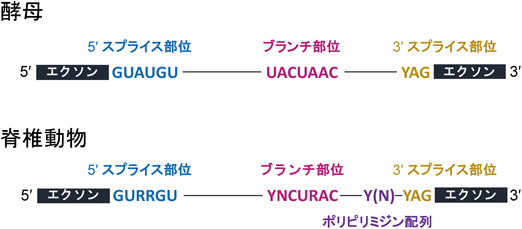
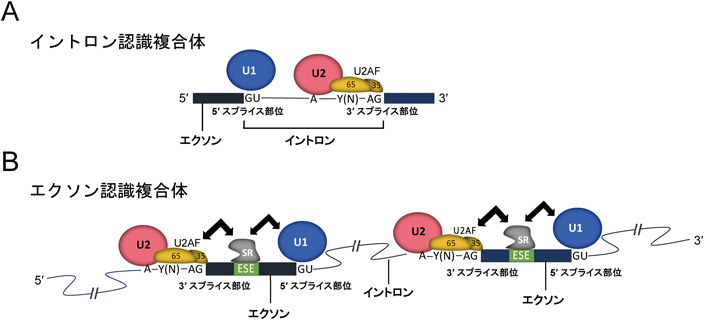
正しいスプライシングにはイントロンとエクソン境界を正確に認識する必要がある.そのための大事な目印となるのが,イントロン間で保存されている特徴的なスプライシングの信号配列である(図1).エクソンとイントロンの境界は5′スプライス部位(5′ splice site),3′スプライス部位(3′ splice site)と呼ばれる特徴的な配列によって分断されている.3′スプライス部位の数十塩基上流には,ブランチ部位(branch site)と呼ばれる配列が位置しているが,その配列保存性は酵母では高いが,脊椎動物では低い.その代わり,ブランチ部位と3′スプライス部位の間にピリミジン塩基に富んだポリピリミジン配列(polypyrimidine tract)があり,イントロンとエクソンを区別するために重要な役割を果たしている.これらの信号配列は,スプライシングの遂行装置であるスプライソソームの構成因子が動的に相互作用することで認識される.5′スプライス部位はU1 snRNPが結合し,ポリピリミジン配列と3′スプライス部位は,U2AF65/U2AF35(U2AF2/U2AF1)二量体(以下U2AF)が結合する.U2AF65は,U2 snRNPの構成タンパク質であるSF3B1と結合することで,U2 snRNPによるブランチ部位の認識を誘導する(図2A).
スプライシング機構の解明を目指した研究が盛んだった1980年代は,スプライシングによって取り除かれる本体であるイントロンが認識されて,スプライシングが起こるという概念に,まったく疑問の余地がなかった.実際にスプライシング効率のいいmRNA前駆体では,両端のスプライス部位配列を含むイントロン本体だけでスプライシング反応には必要十分である7).今日では,これを「イントロン認識モデル(intron definition model)」という.当時in vitroスプライシングで用いられたのは,比較的短い1個のイントロンに両端のエクソン配列の一部がつながっているミニ遺伝子から転写したmRNA前駆体だった.ところが1990年,Berget研究室からの報告によってブレークスルーが起きた.下流エクソンの5′スプライス部位も含めたミニ遺伝子の転写物を使うと,一見関係のなさそうな上流イントロンのスプライシングが劇的に促進されたのである.ところが,下流エクソンの長さが300塩基を超えると,この促進効果はなくなった8).これが「エクソン認識モデル(exon definition model)」誕生の先駆けとなった論文であり,あまりにも画期的だったので著者が出版に苦労したという裏話を聞いた.
スプライシングの初期段階であるスプライソソームA複合体(以下A複合体)において,イントロンの長さに応じて,イントロン・エクソンの認識機構が異なるだろう,というのが今日の確立された概念となっている9, 10).一般的に短いイントロンの場合,イントロンをまたいでmRNA前駆体に結合したU1 snRNPとU2AF/U2 snRNPが主体となるイントロン認識複合体(intron definition complex)が形成されることで,イントロンの認識とスプライシングが行われる(図2A).一方,一般的に長いイントロンの場合は,mRNA前駆体に結合したU2AF/U2 snRNPとU1 snRNPが,エクソンをまたいで相互作用したエクソン認識複合体(exon definition complex)が形成されることで,エクソンが先に認識される(図2B).この際,エクソン内のスプライシング促進配列(exonic splicing enhancer:ESE)にSRタンパク質が結合すること,さらにSRタンパク質がU2AF35と,U1 snRNPの構成因子の70Kタンパク質の両方に,タンパク質間結合できることが,エクソン認識複合体の実験的な根拠となっている11–13).形成されたエクソン認識複合体は,隣接するエクソン複合体と相互作用し,長いイントロンを湾曲させ,はみ出させる形で両端のスプライス部位が近づき,スプライシング反応が進行するというモデルが提起された(図2B).
前述したとおり,イントロンの長さは,高等真核生物になるほど長くなっていくが,ほとんどのエクソンの長さは数百塩基に満たないので,高等真核生物では,スプライシング初期に短いエクソンがまず認識されるメカニズムが確かに合理的である.
3. 長いイントロンを取り除く多段階スプライシング機構
ヒトのイントロンは3000個以上が5万塩基より長く,1200個以上が10万塩基より長い14).これらの非常に長いイントロンをスプライシングするには,エクソン認識モデルが理にかなっているのはいいとしよう.さて,エクソン認識複合体が,3次元空間で遠く離れている状態から,どのようにして隣接すべき互いを正確に見つけ出して接近し相互作用できるようになるのだろうか? さらに,RNAポリメラーゼIIの転写速度は平均して3000塩基/分なので,10万塩基長のイントロンがあった場合,すべてを転写するのに30分以上要する15, 16).その転写が完了するまでの長い時間に,mRNA前駆体は,どのようにして分解から保護されているのだろうか? このように,非常に長いイントロンのスプライシング機構は謎めいており,長年にわたる未解決問題だった.
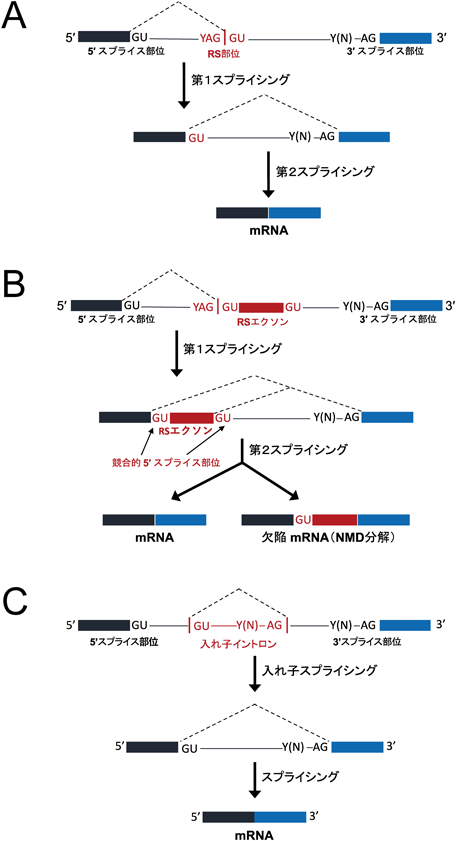
1998年,ショウジョウバエで発見された再帰的スプライシング(recursive splicing)機構は,この謎を解決する鍵となっていると思われた17, 18).これは,長いイントロンの5′側から順繰りにスプラシングを繰り返すことによって,段階的にイントロンが取り除かれていく現象である(図3A).再帰的スプライシングが起こる原因は,長いイントロンにある再帰的スプライス部位(RS部位)と呼ばれる3′スプライス部位と5′スプライス部位が直結した配列(YAG/GU)にある.本来ならこの間にエクソンが挟まっているわけで,これを0塩基エクソンと表現する論文もある.このRS部位が使われることによって,上流から順繰りに継続的なスプライシングが起こり,最終的に長いイントロンが取り除かれる仕組みである.再帰的スプライシングが最初に見つかったのはショウジョウバエのUbx遺伝子のイントロンであるが17),その後に見つかったのは,違う種類のショウジョウバエを含む数個のイントロンだけであり18),再帰的スプライシングはハエに特異的な現象ではないかと考えられて,その一般性には疑問が残されていた.
筆者らは,哺乳動物の非常に長いイントロンでも再帰的スプライシングが起こっているに違いないと考えて,ヒトのジストロフィン(DMD)遺伝子に存在するイントロン7(110,119塩基)を用いて,再帰的スプライシング現象の証拠を精力的に探索した.結果的には,イントロン7内部に,再帰的スプライシングが起こった証拠は見つからなかったが,その代わりに,入れ子状に存在する二つの内部イントロン断片が投げ縄状構造をとって存在していることを確認した.投げ縄状RNAは,スプライシング反応以外の原因で形成されることはない.この事実から,入れ子状に存在する複数の内部イントロン部分のスプライシング後に,短くなったイントロン7を取り除くスプライシングが起こっていると予想し,長いイントロンのスプライシング機構の一つとして,入れ子スプライシング仮説(nested splicing)を提唱した19)(図3C).
次世代シーケンサーを用いた網羅的な解析ができるようになり,このような多段階スプライシング現象の実体に光が当てられた.ヒトの初代内皮細胞,脳細胞,ショウジョウバエの転写産物のパターン分析から再帰的スプライシングの中間産物が検出され,2015年になってついに,再帰的スプライシングが生物種を問わず,長いイントロンにおける一般性の高いスプライシング機構であることが明らかにされた6, 20, 21).さらに,筆者らが分子生物学的解析データから存在を予想した入れ子スプライシングも,同じDMD遺伝子において,多くの長いイントロンで起こっていることが確認された22).非常に長いイントロンのスプライシング機構に,このようなイントロン内の多段階スプライシングが関わっている事実は,もう間違いないようだ.
網羅的解析された再帰的スプライシング機構を詳しくみてみよう.ショウジョウバエのイントロン内のRS部位の特定が行われ,197の機能的なRS部位が115の遺伝子で同定された21)(図3A).この197のRS部位は複数のショウジョウバエ系統間で高度に保存されていた.ショウジョウバエのRS部位の3′スプライス部位は,通常のイントロンの3′スプライス部位に比べて,ポリピリミジン配列が長く,C/U配列に富んだ配列であることも明らかになった.このことから,再帰的スプライシングは通常のイントロンよりもポリピリミジン配列を認識するU2AFに強く依存しているようだ.実際,ショジョウバエU2AFの発現抑制によって,再帰的スプライシングが強く阻害された.
ヒトの再帰的スプライシングでは,新たな事実も見つかった.ヒト脳の九つの遺伝子で同定された再帰的スプライシングは,ショウジョウバエの再帰的スプライシングと比較すると,その過程が少し異なっていた6).特定された多くのRS部位を調べたところ,RS部位は,エクソン(RSエクソンと呼ぶ)として機能しており,競合する二つの5′スプライス部位を持つ中間体を生成していた(図3B).RSエクソンが残ってしまったmRNAは,フレームシフトにより,RSエクソンの内部に終止コドンが生じてしまう場合が多く,ナンセンス変異依存的mRNA分解機構(nonsense mediated mRNA decay:NMD)によって分解され,結果的にその遺伝子発現が抑制されてしまう.したがってヒトの場合,再帰的スプライシングは,遺伝子発現の入/切の制御スイッチとしても利用されると考えられる.
DMD遺伝子を用いた解析では,通常の一段階スプライシングを受けるイントロン長は平均6400塩基(107~38,368塩基)に対し,多段階スプライシング(再帰的スプライシングと入れ子スプライシング)が起こるイントロン長は平均4万塩基(650~248,401塩基)である22).イントロン内の多段階スプライシングは,非常に長いイントロンを取り除く合理的な機構であることに疑問の余地はないだろう.しかしながら,入れ子スプライシングは,まだすべての遺伝子で解析されたわけではなく,また再帰的スプライシングにおいても,ショウジョウバエでは長いイントロンの6%,ヒトでは9個のイントロンのみで明らかにされたにすぎない.言うまでもないが,長いイントロンを取り除く未知のメカニズムの存在は否定できず,今後のさらなる研究が期待される.
4. 短いイントロンで発見された特異的なスプライシング機構
長いイントロンの問題とともに,短いイントロンが,どのようにスプライシングされるのかという問題もスプライシングの発見以来,現在になっても未解決である.
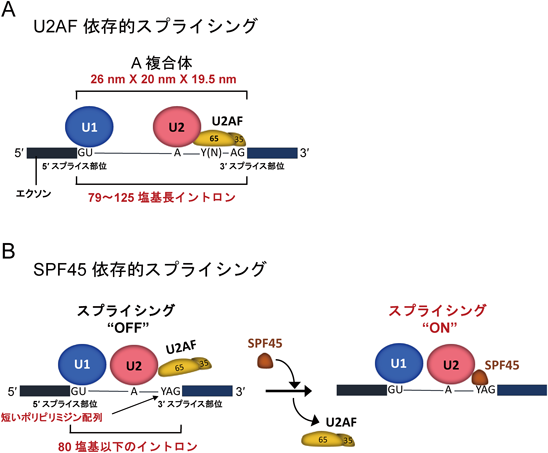
1984年に,ヒトのin vitroスプライシング系を用いて,人工的にイントロン長を短くしていく方法で,スプライシングに最低限必要なイントロン長が調べられた23).その結果,正常にスプライシングされるには,イントロンの長さが80塩基以上必要であることが示された.実際に,イントロン認識複合体であるA複合体を電子顕微鏡で観察すると,その大きさは約26 nm×20 nm×19.5 nmである24).この複合体の大きさは,約79~125塩基のRNA長に対応し,前述のin vitroスプライシングの結果と矛盾はない(図4A).しかしながら,ヒトの遺伝子には80塩基以下のイントロンも数多く存在する.実際に筆者らは,ヒトゲノムと転写産物のデータベースから85塩基より短いイントロンを4500個以上も見いだし,さらに65塩基以下のイントロンが正確にスプライシングされていることを実証した25, 26).
それでは,80塩基以下の短いイントロンはどのような機構でスプライシングされているのであろうか? 1990年代に短いイントロンのスプライシング信号配列について,注目すべき報告がある.ショウジョウバエの長いイントロンは,C/U塩基に富む長いポリピリミジン配列を持つが,短いイントロンの多くは,重要なポリピリミジン配列を欠失していた27, 28).これは,ショウジョウバエに限ったことではないようだ.近年のバイオインフォマティクス解析によって,ヒトの100塩基以下の全イントロンでは,ポリピリミジン配列が短い代わりに,ブランチ部位がより共通配列に近いという特徴が明らかになった29).ポリピリミジン配列にはU2AFが結合し,それは3′スプライス部位の認識とU2 snRNPを導入するために必須なので,その配列が短いということは,スプライシング信号配列としては弱いといえる.実際に,1995年にはポリピリミジン配列が不完全な短いイントロンでは,それを克服する異なるメカニズムがあるのではないかと予想されていた9).2009年に,ショウジョウバエで違う長さのイントロン(147塩基と62塩基)を用い,それらのスプライソソーム構成因子の比較と電子顕微鏡による解析が行われ,62塩基長イントロンではスプライソソームの形状をコンパクトにすることで,スプライシングを遂行する可能性が示唆されたが,その詳細なメカニズムは謎のままであった30).
それから10年後,短いイントロンのスプライシグに関わる因子の一つが同定された31).酵母とヒトのスプライソソームの構成因子を比較し,ヒトのスプライソソームのみに含まれる因子群の中から,Smu1とREDが注目された.Smu1とREDの発現を抑制した培養細胞の全転写物(トランスクリプトーム)解析を行うと,選択的スプライシングの変化とともに,多くの短いイントロンのスプライシングが阻害されていた.さらにSmu1–RED複合体は,5′スプライス部位とブランチ部位の距離が短い場合に機能して,スプライソソームの構造を変化させることで,短いイントロン上にスプライソソームを形成させることを示した.しかしながら,Smu1–RED複合体によるスプライシング機構においては,ポリピリミジン配列の長短は関係ない.それゆえ,一般的にポリピリミジン配列が短い特徴がある短いイントロンのスプライシングは,Smu1–RED因子では説明できず,未知のスプライシング因子が存在するに違いないと予想できた.
筆者らの長年の短いイントロンの基礎研究は,その未知なる短いイントロン特異的なスプライシング必須因子の同定にたどり着き,この疑問を解き明かす結果となった32).HNRNPH1遺伝子の短いイントロン(56塩基)をモデルとして,そのスプライシング活性を指標に154種類の核タンパク質に対するsiRNAライブラリをスクリーニングすることにより,スプライシング必須因子を探索した.その結果,選択的スプライシング調節因子として知られていたSPF45(RBM17)が同定された.SPF45を発現抑制したヒト培養細胞のトランスクリプトーム解析を行うと,多くの短いイントロンでスプライシングが抑制されていた.それらのSPF45依存的にスプライシングされる短いイントロンのポリピリミジン配列は有意に短く,既知の結合因子であるU2AFが結合していなかった.これは,前述したように,短いイントロンが一般的に,短く弱いポリピリミジン配列を持つという特徴と見事に合致する.さらに,SPF45はU2AFに置き換わってU2 snRNPの構成タンパク質SF3B1と結合し,U2 snRNPがブランチ部位に導入され,スプライシングが引き起こされることを解明した(図4B).これらの事実は,スプライシング必須因子であるU2AFの代わりにSPF45を用いることで,そのスプライシング反応を可能にしている短いイントロン群が存在することを意味する.実際にU2AFよりもSPF45の方が分子が小さいため,スプライシング初期のA複合体をよりコンパクトにでき,短いイントロンに適合しているのだろう(図4B).
短いイントロンのスプライシング機構について,未解明だった問題にようやく光が当てられてきたが,まだまだ隠された機構があると考えられる.筆者らが解析したところ,65塩基以下の微小イントロン群には,イントロン内部の配列が異常にG塩基に富んでいて,5′と3′スプライス部位およびブランチ部位が,まったく機能しない配列になっている奇妙イントロンが散在する26).それらが確実にスプライシングされているのは証明できた.一部の微小イントロン内にはスプライシング促進配列(intronic splicing enhancer:ISE)が見つかり,その関与が示唆されたが,これらの奇妙な微少イントロンのスプライシング分子機構の全体像については,まったくもって謎めいている25, 26).
5. イントロンの「長さ」を識別する機構の破綻と疾患
異常なスプライシングやスプライシング不全は,mRNA前駆体上のスプライス部位はもちろん,スプライシング・エンハンサーやスプライシング・サイレンサーなどの制御配列の変異,さらにはスプライシング因子の変異によって引き起こされ,現在,さまざまな疾患の原因として確立されている33).ここで論じてきたイントロン長に応じたスプライシング機構の破綻もまた,疾患やがんとの関連が強く示唆されている.
ヒトの内皮細胞と脳組織のトランスクリプトーム配列データから高信頼性RS部位を抽出し,ヒトの推定疾患原因変異(SNPs)のデータベースに照らし合わせてみると,それらが予想より高い頻度で,神経系疾患と循環器系疾患に関連する遺伝子に見つかった34).これらの疾患が,再帰的スプライシング過程での間違いによって引き起こされるのだろうか? 大いに興味のあるところだが,今後の研究によって明らかにされるだろう.
意義深いことに,筆者らが新しいスプライシング必須因子として同定したSPF45は,以前に抗がん剤耐性獲得細胞で高発現する遺伝子として報告されていた35).SPF45を培養細胞で過剰発現させると,少なくとも六つの抗がん剤に対する多剤耐性を獲得することが判明した36).抗がん剤の多剤耐性獲得は,抗がん剤を細胞外に排出するポンプ作用を持つトランスポーターの過剰発現が,既知の作用機序である.しかしながら,SPF45を過剰発現させた抗がん剤多剤耐性細胞は,抗がん剤が細胞内に蓄積したままであり,この事実は,新しい抗がん剤多剤耐性獲得メカニズムの存在を示唆している36).SPF45がスプライシング必須因子と判明した今,SPF45依存的スプライシング機構の解明が,抗がん剤多剤耐性の問題解決の糸口になるのではないかと大胆に予想している37, 38).
イントロンの「長さ」に焦点を当てるアプローチで,一見確立されたように思われるスプライシング機構に,いまだに新しいスプライシング機構が発見されている.「故きを温ね新しきを知る」基礎研究の醍醐味を多少は味わっていただけたと思う.教科書に書かれたスプライシング機構が対応する,ほどよい長さから大きくかけ離れた長いイントロンでは,イントロン内の多段階スプライシングが関与し,逆に短いイントロンでは短いポリピリミジン信号配列に対応できる新たなスプライシング因子SPF45が発見された(図5).イントロンが発見されて半世紀近く経っているが,イントロン集合に,既知の機構でスプライシングされない短いイントロンの部分集合が新たに存在したのである.ところが「一難問去ってまた一難問」,いまだに解明できない巨大イントロンと微小イントロンの存在がある.未知のスプライシング機構やその生理的意義のさらなる謎解き,その飽くなき挑戦に終わりはない.
引用文献References
1) Will, C.L. & Lührmann, R. (2011) Spliceosome structure and function. Cold Spring Harb. Perspect. Biol., 3, a003707.
2) Kastner, B., Will, C.L., Stark, H., & Lührmann, R. (2019) Structural insights into nuclear pre-mRNA splicing in higher Eukaryotes. Cold Spring Harb. Perspect. Biol., 11, a032417.
3) Kupfer, D.M., Drabenstot, S.D., Buchanan, K.L., Lai, H., Zhu, H., Dyer, D.W., Roe, B.A., & Murphy, J.W. (2004) Introns and splicing elements of five diverse fungi. Eukaryot. Cell, 3, 1088–1100.
4) Piovesan, A., Caracausi, M., Ricci, M., Strippoli, P., Vitale, L., & Pelleri, M.C. (2015) Identification of minimal eukaryotic introns through GeneBase, a user-friendly tool for parsing the NCBI Gene databank. DNA Res., 22, 495–503.
5) Rogozin, I.B., Carmel, L., Csuros, M., & Koonin, E.V. (2012) Origin and evolution of spliceosomal introns. Biol. Direct, 7, 11.
6) Sibley, C.R., Emmett, W., Blazquez, L., Faro, A., Haberman, N., Briese, M., Trabzuni, D., Ryten, M., Weale, M.E., Hardy, J., et al. (2015) Recursive splicing in long vertebrate genes. Nature, 521, 371–375.
7) Mayeda, A. & Ohshima, Y. (1990) β-globin transcripts carrying a single intron with three adjacent nucleotides of 5′ exon are efficiently spliced in vitro irrespective of intron position or surrounding exon sequences. Nucleic Acids Res., 18, 4671–4676.
8) Robberson, B.L., Cote, G.J., & Berget, S.M. (1990) Exon definition may facilitate splice site selection in RNAs with multiple exons. Mol. Cell. Biol., 10, 84–94.
9) Berget, S.M. (1995) Exon recognition in vertebrate splicing. J. Biol. Chem., 270, 2411–2414.
10) De Conti, L., Baralle, M., & Buratti, E. (2013) Exon and intron definition in pre-mRNA splicing. Wiley Interdiscip. Rev. RNA, 4, 49–60.
11) Wu, J.Y. & Maniatis, T. (1993) Specific interactions between proteins implicated in splice site selection and regulated alternative splicing. Cell, 75, 1061–1070.
12) Kohtz, J.D., Jamison, S.F., Will, C.L., Zuo, P., Lührmann, R., Garcia-Blanco, M.A., & Manley, J.L. (1994) Protein–protein interactions and 5′-splice-site recognition in mammalian mRNA precursors. Nature, 368, 119–124.
13) Staknis, D. & Reed, R. (1994) SR proteins promote the first specific recognition of Pre-mRNA and are present together with the U1 small nuclear ribonucleoprotein particle in a general splicing enhancer complex. Mol. Cell. Biol., 14, 7670–7682.
14) Shepard, S., McCreary, M., & Fedorov, A. (2009) The peculiarities of large intron splicing in animals. PLoS One, 4, e7853.
15) Wada, Y., Ohta, Y., Xu, M., Tsutsumi, S., Minami, T., Inoue, K., Komura, D., Kitakami, J., Oshida, N., Papantonis, A., et al. (2009) A wave of nascent transcription on activated human genes. Proc. Natl. Acad. Sci. USA, 106, 18357–18361.
16) Singh, J. & Padgett, R.A. (2009) Rates of in situ transcription and splicing in large human genes. Nat. Struct. Mol. Biol., 16, 1128–1133.
17) Hatton, A.R., Subramaniam, V., & Lopez, A.J. (1998) Generation of alternative Ultrabithorax isoforms and stepwise removal of a large intron by resplicing at exon–exon junctions. Mol. Cell, 2, 787–796.
18) Burnette, J.M., Miyamoto-Sato, E., Schaub, M.A., Conklin, J., & Lopez, A.J. (2005) Subdivision of large introns in Drosophila by recursive splicing at nonexonic elements. Genetics, 170, 661–674.
19) Suzuki, H., Kameyama, T., Ohe, K., Tsukahara, T., & Mayeda, A. (2013) Nested introns in an intron: Evidence of multi-step splicing in a large intron of the human dystrophin pre-mRNA. FEBS Lett., 587, 555–561.
20) Kelly, S., Georgomanolis, T., Zirkel, A., Diermeier, S., O’Reilly, D., Murphy, S., Langst, G., Cook, P.R., & Papantonis, A. (2015) Splicing of many human genes involves sites embedded within introns. Nucleic Acids Res., 43, 4721–4732.
21) Duff, M.O., Olson, S., Wei, X., Garrett, S.C., Osman, A., Bolisetty, M., Plocik, A., Celniker, S.E., & Graveley, B.R. (2015) Genome-wide identification of zero nucleotide recursive splicing in Drosophila. Nature, 521, 376–379.
22) Gazzoli, I., Pulyakhina, I., Verwey, N.E., Ariyurek, Y., Laros, J.F., ’t Hoen, P.A., & Aartsma-Rus, A. (2016) Non-sequential and multi-step splicing of the dystrophin transcript. RNA Biol., 13, 290–305.
23) Wieringa, B., Hofer, E., & Weissmann, C. (1984) A minimal intron length but no specific internal sequence is required for splicing the large rabbit β-globin intron. Cell, 37, 915–925.
24) Behzadnia, N., Golas, M.M., Hartmuth, K., Sander, B., Kastner, B., Deckert, J., Dube, P., Will, C.L., Urlaub, H., Stark, H., et al. (2007) Composition and three-dimensional EM structure of double affinity-purified, human prespliceosomal A complexes. EMBO J., 26, 1737–1748.
25) Sasaki-Haraguchi, N., Shimada, M.K., Taniguchi, I., Ohno, M., & Mayeda, A. (2012) Mechanistic insights into human pre-mRNA splicing of human ultra-short introns: Potential unusual mechanism identifies G-rich introns. Biochem. Biophys. Res. Commun., 423, 289–294.
26) Shimada, M.K., Sasaki-Haraguchi, N., & Mayeda, A. (2015) Identification and validation of evolutionarily conserved unusually short pre-mRNA introns in the human genome. Int. J. Mol. Sci., 16, 10376–10388.
27) Mount, S.M., Burks, C., Hertz, G., Stormo, G.D., White, O., & Fields, C. (1992) Splicing signals in Drosophila: Intron size, information content, and consensus sequences. Nucleic Acids Res., 20, 4255–4262.
28) Guo, M., Lo, P.C., & Mount, S.M. (1993) Species-specific signals for the splicing of a short Drosophila intron in vitro. Mol. Cell. Biol., 13, 1104–1118.
29) Corvelo, A., Hallegger, M., Smith, C.W.J., & Eyras, E. (2010) Genome-wide association between branch point properties and alternative splicing. PLoS Comput. Biol., 6, e1001016.
30) Herold, N., Will, C.L., Wolf, E., Kastner, B., Urlaub, H., & Lührmann, R. (2009) Conservation of the protein composition and electron microscopy structure of Drosophila melanogaster and human spliceosomal complexes. Mol. Cell. Biol., 29, 281–301.
31) Keiper, S., Papasaikas, P., Will, C.L., Valcárcel, J., Girard, C., & Lührmann, R. (2019) Smu1 and RED are required for activation of spliceosomal B complexes assembled on short introns. Nat. Commun., 10, 3639.
32) Fukumura, K., Yoshimoto, R., Sperotto, L., Kang, H.S., Hirose, T., Inoue, K., Sattler, M., & Mayeda, A. (2021) SPF45/RBM17-dependent, but not U2AF-dependent, splicing in a distinct subset of human short introns. Nat. Commun., 12, 4910.
33) Chabot, B. & Shkreta, L. (2016) Defective control of pre-messenger RNA splicing in human disease. J. Cell Biol., 212, 13–27.
34) Georgomanolis, T., Sofiadis, K., & Papantonis, A. (2016) Cutting a long intron short: Recursive splicing and its implications. Front. Physiol., 7, 598.
35) Sampath, J., Long, P.R., Shepard, R.L., Xia, X., Devanarayan, V., Sandusky, G.E., Perry, W.L. III, Dantzig, A.H., Williamson, M., Rolfe, M., et al. (2003) Human SPF45, a splicing factor, has limited expression in normal tissues, is overexpressed in many tumors, and can confer a multidrug-resistant phenotype to cells. Am. J. Pathol., 163, 1781–1790.
36) Perry, W.L. III, Shepard, R.L., Sampath, J., Yaden, B., Chin, W.W., Iversen, P.W., Jin, S., Lesoon, A., O’Brien, K.A., Peek, V.L., et al. (2005) Human splicing factor SPF45 (RBM17) confers broad multidrug resistance to anticancer drugs when overexpressed—a phenotype partially reversed by selective estrogen receptor modulators. Cancer Res., 65, 6593–6600.
37) Fukumura, K., Venables, J.P., & Mayeda, A. (2021) SPF45/RBM17-dependent splicing and multidrug resistance to cancer chemotherapy. Mol. Cell. Oncol., 8, 1996318.
38) 福村和宏,前田明(2021) スプライシングと抗がん剤耐性. 細胞,53, 900–901.
著者紹介Author Profile
 福村 和宏(ふくむら かずひろ)
福村 和宏(ふくむら かずひろ)藤田医科大学・医科学研究センター・遺伝子発現機構学研究部門 講師.博士(理学).
略歴2004年熊本大学理学部生物科学科卒業.09年神戸大学大学院自然科学研究科博士課程後期修了.同年ペンシルバニア大学医学部博士研究員.12年サーモフィッシャーサイエンティフィック フィールドアプリケーションサイエンティスト.15年藤田保健衛生大学・総合医科学研究所・遺伝子発現機構学研究部門,助教.21年より現職.
研究テーマと抱負一つの遺伝子から選択的スプライシグによって複数のタンパク質を生み出すという美しい機構に魅了されて20年.ドラマティックな研究を続けていきたいと考えています.
ウェブサイトhttp://www.fujita-hu.ac.jp/~mayeda/
趣味登山,ボルダリング.
 前田 明(まえだ あきら)
前田 明(まえだ あきら)藤田医科大学・医科学研究センター・遺伝子発現機構学研究部門 教授.医学博士.
略歴1983年埼玉大学理学部生体制御学科卒業.89年筑波大学大学院医学研究科修了,医学博士.89年日本学術振興会,特別研究員(PD).90年コールドスプリングハーバー研究所,博士研究員/准研究員/研究員.98年マイアミ大学医学部生化学分子生物学科,助教授.2007年より現職.
研究テーマと抱負スプライシング発見45年~スプライシング研究40年.驚くべきことに,その分子機構において,いまだに未解決な問題がある.時流に流されず,それらの問題を世界に先駆けて,美しく解明したい.
ウェブサイトhttp://www.fujita-hu.ac.jp/~mayeda/(研究室)
https://www.rnaj.org/index.php?option=com_k2&view=item&id=972:mayeda-4(日本RNA学会会報の継続エッセイ)
趣味音楽全般(鍵盤楽器演奏),ヨガ,ブレイキン,原始仏典研究,数学鑑賞,デザイン創作.