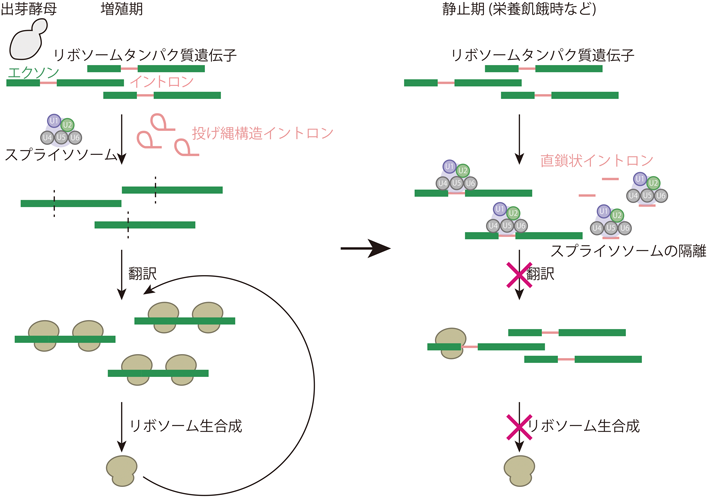
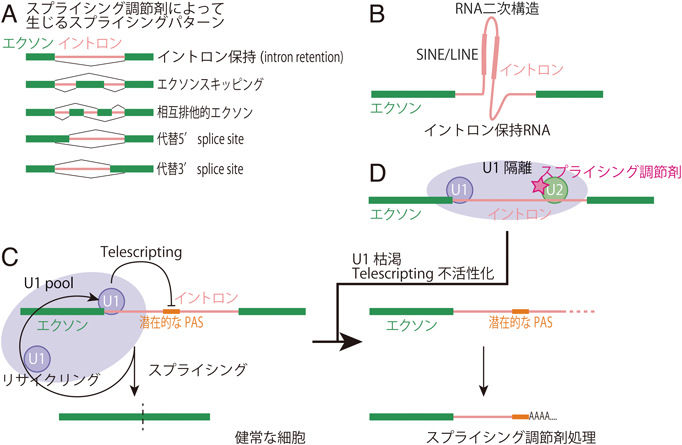
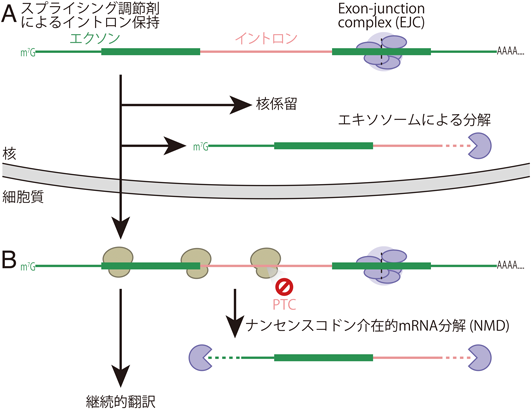
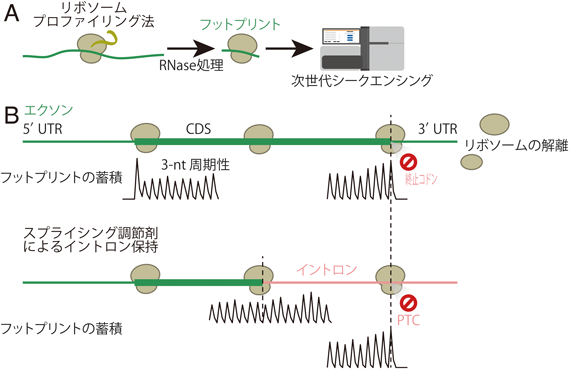
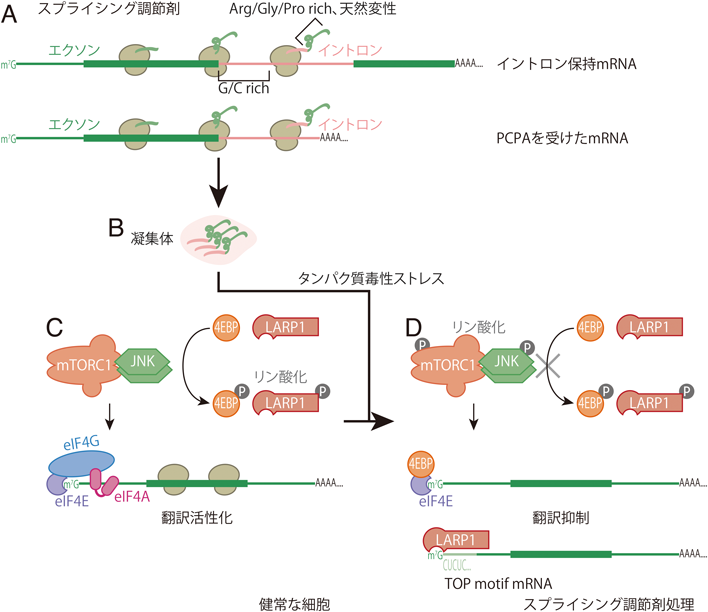
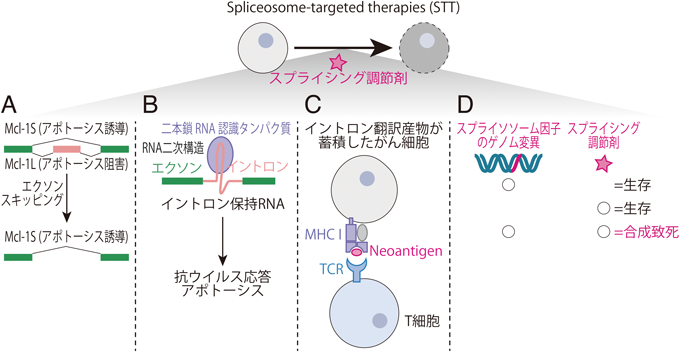
スプライシング阻害と翻訳制御Splicing modulation-mediated translational control
1 理化学研究所開拓研究本部RNA Systems Biochemistry Laboratory, RIKEN Cluster for Pioneering Research ◇ 〒351–0198 埼玉県和光市広沢2–1 ◇ 2–1 Hirosawa, Wako, Saitama 351–0198, Japan
2 東京大学大学院新領域創成科学研究科メディカル情報生命専攻Department of Computational Biology and Medical Sciences, Graduate School of Frontier Sciences, The University of Tokyo ◇ 〒277–8561 千葉県柏市柏の葉5–1–5 ◇ 5–1–5 Kashiwanoha, Kashiwa, Chiba 277–8561, Japan
3 理化学研究所環境資源科学研究センターChemical Genomics Research Group, RIKEN Center for Sustainable Resource Science ◇ 〒351–0198 埼玉県和光市広沢2–1 ◇ 2–1 Hirosawa, Wako, Saitama 351–0198, Japan
4 東京大学大学院大学院農学生命科学研究科応用生命工学専攻Department of Biotechnology, Graduate School of Agricultural and Life Sciences, The University of Tokyo ◇ 〒113–8657 東京都文京区弥生1–1–1 ◇ 1–1–1 Yayoi, Bunkyo-ku, Tokyo 113–8657, Japan
5 東京大学微生物科学イノベーション連携研究機構Collaborative Research Institute for Innovative Microbiology, The University of Tokyo ◇ 〒113–8657 東京都文京区弥生1–1–1 ◇ 1–1–1 Yayoi, Bunkyo-ku, Tokyo 113–8657, Japan