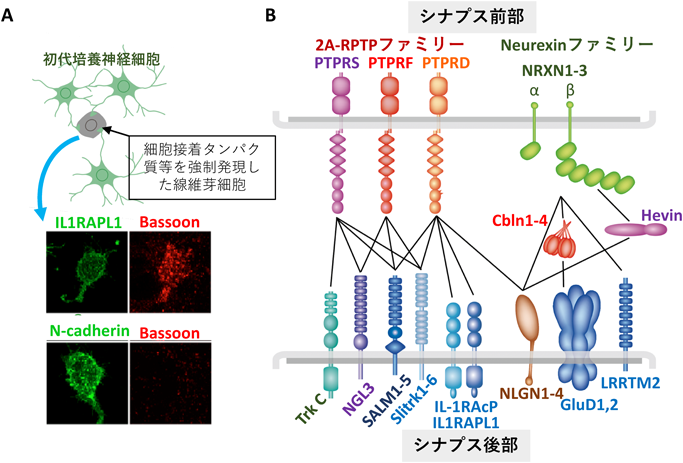
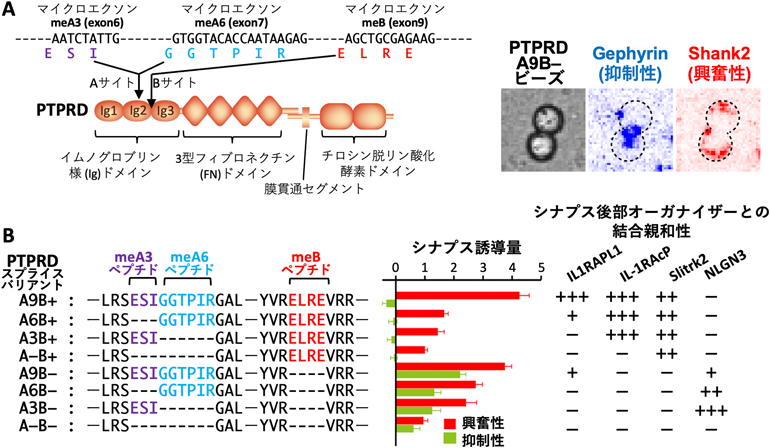
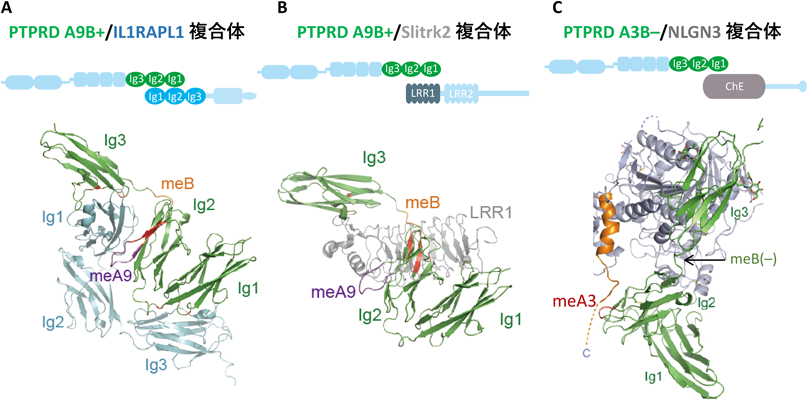
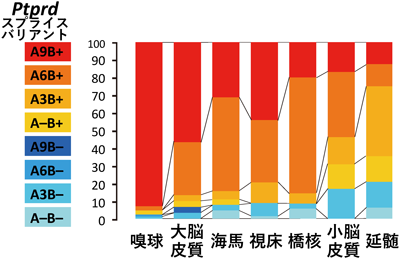
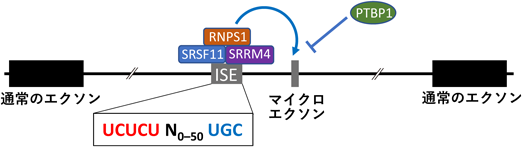
マイクロエクソンの取捨選択による中枢シナプス形成の調節Synaptic target selection regulated by Ptprd microexons’ splicing
富山大学学術研究部医学系分子神経科学講座Department of Molecular Neuroscience, Faculty of Medicine, Academic Assembly, University of Toyama ◇ 〒930–0194 富山県富山市杉谷2630 ◇ Sugitani 2630, Toyama 930–0194, Japan