1)植物のpre-mRNAスプライシング制御の特徴
pre-mRNAスプライシングは,スプライソソームと呼ばれる分子装置によって触媒される反応である.スプライソソームのコア因子はU small nuclear ribonucleoprotein(U snRNP)であり,U snRNA(Uridine-rich small nuclear RNA)とその特異的相互作用タンパク質群の複合体である1, 2).snRNPによるpre-mRNAスプライシング反応の分子機序は,酵母やヒトのpre-mRNAスプライシング研究から明らかにされており,その詳細は他著1, 2)に譲る.U snRNPはじめ,重要なpre-mRNAスプライシング因子は植物でも保存されていることから,基本的なpre-mRNAスプライシング機構は植物を含めて広く真核生物で共通していると考えられている3).
その一方で,植物ではゲノム倍化によって,pre-mRNAスプライシング因子をコードする遺伝子数が,平均的な動物種のケースの2倍程度に増加していることもわかっている3, 4).たとえば,serine-arginine(SR)タンパク質ファミリーに関しては,植物特異的なドメイン構造を持つRSサブファミリー,RS2Zサブファミリー,SCLサブファミリー,SR45サブファミリー,の四つのサブファミリーが見つかっている3, 5).シロイヌナズナSR45タンパク質は,環境応答や形態形成に重要な役割を担うことが報告されており6, 7),植物特異的なpre-mRNAスプライシング因子の重要性もわかりつつある.
また近年のゲノムワイド解析によって,植物におけるpre-mRNAスプライシング制御のゲノムワイドな特徴も明らかにされている.たとえば,選択的pre-mRNAスプライシングはエクソンとイントロンの選択形式によって分類されるが,ヒトでは特定のエクソンを飛ばしてpre-mRNAスプライシングを起こす「スキッピングエクソン型」の選択的pre-mRNAスプライシングの頻度が高い一方で,植物では「イントロン保持(intron retain:IR)型」の頻度が最も高い8–10).IR産物は一般的に新規のストップコドンを生み出す場合が多く,これはRNAの品質管理機構の一つであるナンセンス変異依存的mRNA分解機構(non-sense mediated mRNA decay:NMD)の標的になるため,その多くはmRNA段階で分解されタンパク質に至らない不良RNAと考えられてきた11).しかしながら植物においては,NMDの標的とはならないIR産物が存在すること12),選択的pre-mRNAスプライシングの産物の35%がポリソームに結合しており,翻訳されていると考えられること13)が報告されている.特に,コドンの読み枠が変わらずGC含有率がエクソン並みに高いイントロンは「Existron」として整理・注目されており,植物では特にその比率が高いことが確認されている14, 15).このように,植物では,動物とは異なる選択的pre-mRNAスプライシング制御の特徴を進化させてきたと考えられる.
2)植物におけるU snRNP生合成経路
先述のとおり,U snRNAは,スプライソソームのコア因子U snRNPを構成する重要な小分子RNAである.ヒトのU snRNA遺伝子は,ゲノム上に5~20のコピー遺伝子がタンデムに反復した状態でコードされており16),このU snRNA遺伝子座領域のクロマチン構造が緩み,カハール体(Cajal body)と結合することで,活発な転写が行われる17).これに対して,シロイヌナズナゲノムにはこうしたU snRNA遺伝子の高度なタンデムリピートは見つからず,代わりにクラスターを形成して存在していることが見いだされた4).シロイヌナズナゲノムでは,U4 snRNAの180~300 nt上流にU1 snRNAが位置するU1 snRNAとU4 snRNAのクラスター(U1–U4 snRNAクラスター)が7個見つかっており,その多くは5番染色体に位置している.また,U2-U5 snRNAクラスターや,U2 snRNAとU5 snRNAのタンデム重複も見つかっており,U snRNA遺伝子のゲノム配置は,複数箇所に集中しているものの,ヒトとはまったく異なる配置をとることがわかっている4).
哺乳類研究からは,スプライソソームを構成するsnRNPの生合成経路は,snRNAのクラスによって異なることも示されている.RNAポリメラーゼIIによって転写されるSmクラスsnRNA(U1, U2, U4, U5, U11, U12 snRNAおよびU4atac snRNA)の場合,snRNAの修飾やプロセシングは,核内と核外の両方で行われるが,一方で,RNAポリメラーゼIIIによって転写されるSm-likeクラスsnRNA(U6 snRNAおよびU6atac snRNA)は,核内ですべての過程が終結する2, 18).具体的には,SmクラスsnRNAは,核内でsnRNA特異的転写因子複合体(snRNA activating protein complex:SNAPc)によって転写され修飾を受けた後,核外へ輸送されキャップ構造のトリメチル化修飾を受ける.さらにトリミングを受け,特異的相互作用タンパク質と結合した後,再び核内に輸送され,カハール体や核小体でsnRNP構成タンパク質と結合し,成熟snRNPとなる2, 18, 19).興味深いことに,シロイヌナズナゲノムからは,これら哺乳類snRNP生合成因子について,一部しかホモログ因子が見つからない.たとえば,細胞質においてsnRNAとSmタンパク質群との結合制御に関わるsurvival motor neuron(SMN)複合体関連タンパク質のうち,シロイヌナズナではGEMIN2とAtPRMT5のみが保存されている19–22).このことから,U snRNP生合成経路については,動物と植物では一部異なっている可能性も考えられるが,詳細は不明なままである.今後の解析による実態解明が待たれる.
3)植物snRNP生合成因子のユニークな役割
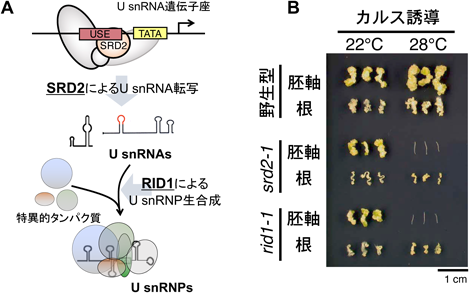
筆者らは,シロイヌナズナのU snRNP生合成関連因子の分子遺伝学的解析を進めている(図1).シロイヌナズナ突然変異体shoot redifferentiation defective 2-1(srd2-1)およびroot initiation defective 1-1(rid1-1)は,高温条件下で組織培養における器官再生不全を示す点変異型温度感受性変異体である23, 24).植物は一般的に高い再生能力を持ち,オーキシンやサイトカイニンといった植物ホルモン条件を調整することで,植物組織片から脱分化(カルス形成)および再分化(器官再生)を誘導することができる.srd2-1とrid1-1の表現型解析から,SRD2とRID1は胚軸脱分化(カルス形成)と,カルスからの新たな分裂組織形成の両方に重要な役割を持つこと,特にカルス形成初期の細胞増殖能獲得に関わることがわかった25–29)(図1).解析の結果,SRD2遺伝子はヒトSNAPcのサブユニットSNAP50のホモログをコードしており,植物細胞内でU snRNA転写制御に関与すること25),RID1遺伝子は核小体に局在しU snRNP成熟に関わる可能性があるDEAH-box型RNAヘリカーゼをコードすること26),が示された.さらには,U snRNAレベルが胚軸細胞の細胞増殖能を規定すること,細胞脱分化には分裂組織の活性維持よりもより高いレベルのU snRNAレベルが必要とされることもわかった27).以上は,U snRNP生合成制御が植物細胞の特徴的性質である細胞増殖・分化能の可塑的制御に本質的な役割を示していることを世界に先駆けて示す,重要な成果であった19, 27, 28).
srd2-1やrid1-1の芽生えでは,高温条件下で側根形成,分裂組織の確立,葉の形態形成,花器官形成に異常がみられることから,U snRNP生合成は器官再生の場面だけでなく,高温ストレス下での植物発生制御にもまた重要であると考えられる19, 26, 28, 30, 31).興味深いことに,前述のU snRNP生合成に関わるSMN複合体サブユニットGEMIN2のシロイヌナズナ変異体gemin2-1は,概日時計の撹乱や低温ストレス高感受性の表現型を示す22).またGEMIN2 mRNA自身,温度依存的な選択的pre-mRNAスプライシング制御を受け,低温条件下では機能的なアイソフォームの割合が増加する32).AtPRMT5も概日時計制御に関わり33, 34),加えて塩や乾燥といった環境ストレス応答にも重要であることが報告されている35–37).さらにU snRNAのキャップ構造をトリメチル化する酵素trimethylguanosine synthase(TGS)の機能不全は,シロイヌナズナにおいて低温耐性を低下させることもわかっている38).以上の知見(表1)は,U snRNP生合成制御は植物の環境ストレス応答においても重要であることを示している.さらに筆者たちの観察では,U snRNP生合成関連変異体それぞれが異なる環境ストレスへの耐性異常を示すこともわかりつつあり,U snRNP生合成のステップが各々異なる役割を果たしていることも推測される.
表1 環境ストレス応答に関与するスプライシング関連変異体の例| 変異体 | 原因遺伝子 | AGI | 高温 | 低温 | 塩 | 参考文献 |
|---|
| sr45 | SR45 | AT1G16610 | | | ✓ | 7) |
| srd2-1 | SRD2 | AT1G28560 | ✓ | | | 23, 35) |
| rid1-1 | RID1 | AT1G26370 | ✓ | | | 26) |
| gemin2-1 | GEMIN2 | AT1G54380 | | ✓ | | 22) |
| tgs1 | TGS1 | AT1G45231 | | ✓ | | 38) |
| cyp18-1 | CYP18-1 | AT1G01940 | ✓ | | | 46) |
| sta1-1 | STA1 | AT4G03430 | ✓ | ✓ | | 47, 48, 49, 50) |
| skip-1 | SKIP | AT1G77180 | | | ✓ | 51, 52) |
| sf1 | SF1 | AT5G51300 | ✓ | ✓ | | 58) |
2. 植物環境応答におけるpre-mRNAスプライシングダイナミクス制御
前節で述べたとおり,U snRNP生合成因子を含むpre-mRNAスプライシング制御因子の変異体がストレス応答異常を示す事例は多く蓄積しており,pre-mRNAスプライシングの環境応答における重要性を端的に示す成果となっている.また,ゲノムワイドトランスクリプトーム解析によって,環境ストレス応答時にpre-mRNAスプライシングパターンが大きく変化することや,選択的スプライシングが生じる部位はストレス種間で異なることがわかりつつある39, 40).このとき,RNAプロセシングに関連する遺伝子の発現も変化する41, 42).また,興味深いことに,pre-mRNAスプライシング阻害剤処理と環境ストレス処理時のトランスクリプトームがよく似ていることも報告されている43, 44).このように,植物環境応答とpre-mRNAスプライシング制御の間には強いつながりがあることが多角的に明らかとなってきた.
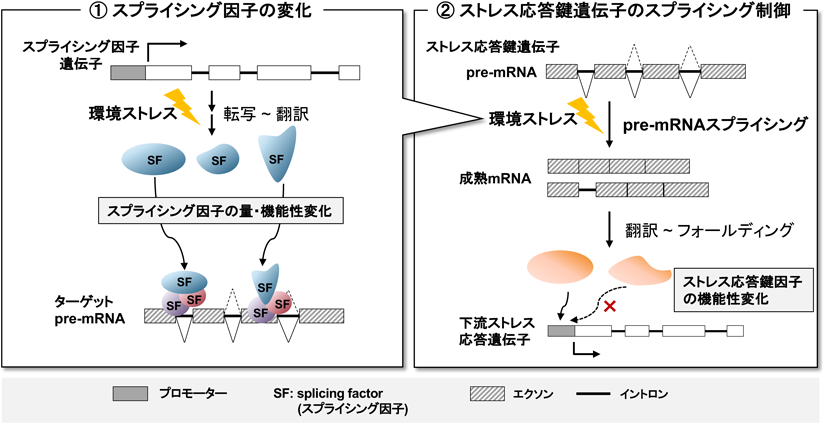
ここで,pre-mRNAスプライシングダイナミクスがどのように環境応答に貢献するのか,その分子作用機序を整理してみよう.論理的には,図2で示したような二つの道筋が想定される.すなわち,第一には,pre-mRNAスプライシング制御因子自体の発現やpre-mRNAスプライシングパターンがストレスに応答して変動し,結果としてpre-mRNAスプライシングパターンの変動をもたらす,というケース(図2の左側)である.さらには,こうしたpre-mRNAスプライシング制御因子の機能性変化によって,ストレス応答の鍵遺伝子mRNAのスプライシングパターンが大きく変動するケースである.この場合,pre-mRNAスプライシングパターン変化の結果としてストレス応答鍵因子の機能性が変化し,適切なストレス応答を誘導すると考えられる(図2の右側).以下,この二つのケースについて,実例をいくつか紹介する.
1)pre-mRNAスプライシング制御因子mRNAのストレス応答性
先述のSR遺伝子のmRNAは,その多くが,高温,低温,光の条件に応答して自身のpre-mRNAスプライシングパターンを変化させることが示されている45).また,pre-mRNAスプライシング因子をコードするCYCLOPHILIN18-1(CYP18-1)遺伝子は高温応答的にその発現が変動し,スプライソソーム構成タンパク質の脱リン酸化を制御することで,pre-mRNAスプライシング調節に関与することが示されている46).その他にも,酵母PRP6のホモログでU4/U6–U5 snRNPのアッセンブリーに関与するSTABILIZED 1(STA1)は,低温,塩ストレスでその発現が上昇する47–50).上述したSR45と相互作用するSki-interacting protein(SKIP)の発現も,塩や乾燥ストレスの影響を受ける51, 52).こうしたpre-mRNAスプライシング因子機能不全変異体は,先のsrd2-1やrid1-1の高温感受性のように,ストレスに弱くなる表現型を示すことが多い19, 26, 28, 30, 31)(表1).さらにSKIPの例では,塩ストレスによって引き起こされるpre-mRNAスプライシングパターン変化の大半がSKIPによって制御されることが示唆されており51),植物は環境ストレスに応答して積極的にpre-mRNAスプライシング制御因子のバリエーションや機能性を変化させ,スプライシングバリアントの作り替えをしていると考えられる.
2)ストレス応答鍵遺伝子mRNAのpre-mRNAスプライシングダイナミクス
ストレス応答鍵遺伝子mRNAのスプライシングダイナミクスが環境ストレス応答に大きな影響をもたらす好例として,たとえば,熱応答遺伝子Heat shock transcription factor A2(HsfA2)があげられる.HsfA2は熱応答に重要な因子であり,hsfa2変異体では熱ストレス高感受性を示す53).HsfA2 mRNAは,熱ストレス依存的なスプライシングダイナミクスを示し54, 55),興味深いことに,熱ストレスによって出現するスプライシングバリアントから作られるHsfA2タンパク質バリアントが,HsfA2遺伝子のプロモーターに結合して自身の転写を活性化するという,正のフィードバックループ転写制御が存在することが示された55).また,花成制御の重要因子である低温応答性遺伝子FLOWERING LOCUS M(FLM)も,温度条件依存的に二つのpre-mRNAスプライシングバリアントFLM-βおよびFLM-δの比が変化する.このうち,FLM-βが機能的なタンパク質をコードするスプライシングバリアントであり,ターゲット遺伝子のプロモーターに結合して花成に抑制的に働く56).それに対し,FLM-δは花成に促進的に働くことから,FLM-βとFLM-δの比が花成制御にとって重要であることが示されている57).さらに,このFLM mRNAの温度依存的なpre-mRNAスプライシング制御には,pre-mRNAスプライシング因子AtSF1が関与することも報告された58).このように,植物の環境応答戦略の一つとして,鍵遺伝子のpre-mRNAスプライシングダイナミクスが積極的に変化し,その結果,植物の環境応答・発生の方向性が決定されるという仕組みが想像される.pre-mRNAスプライシング因子自身の発現やpre-mRNAスプライシングダイナミクスを介して,どのように鍵遺伝子のpre-mRNAスプライシングが変化し,植物の環境応答や発生制御が行われるのか,今後のさらなる解析による包括的理解が期待される.
3. pre-mRNAスプライシングと光合成活性の関わり
葉緑体は植物や藻類などに特有のオルガネラであり,光合成をはじめとするさまざまな代謝反応の場である.また,葉緑体は環境を感知してその情報を核に伝える重要なオルガネラでもある59–61).この葉緑体から核へと発せられるレトログレードシグナルは,核の遺伝子発現制御を介して,光合成活性や形態形成,植物ホルモン応答などを制御することが知られている59–61).長らく葉緑体レトログレードシグナルは,転写因子やmicroRNAを介して核の転写制御に影響されると理解されてきたが,近年,pre-mRNAスプライシングもターゲットにしていることが明らかになりつつある62, 63).
Petrilloら(2014)は,地上部で生成される光条件依存的な葉緑体レトログレードシグナルが,地下部組織におけるpre-mRNAスプライシングパターン(RS31遺伝子mRNAなど)を変化させることを明らかにした62).同グループはさらに研究を進め,葉緑体レトログレードシグナルの少なくとも一部は,地下部のミトコンドリアとtarget of rapamycin(TOR)キナーゼによって,pre-mRNAスプライシング制御に影響すること,また,シグナルの分子実体の少なくとも一部は,地上部から地下部へ輸送される光合成産物である糖が担っていることを報告している63).こうした結果は,pre-mRNAスプライシングは,葉緑体が感知した地上部情報を地下部に伝える際の重要な受け口になっていることを示唆しており,非常に興味深い.筆者らも,現在,こうしたpre-mRNAスプライシング制御を介した環境ストレス応答に注目し,光合成活性依存的な側根形態制御について解析を進めている.これによって想定されてこなかった新たなpre-mRNAスプライシングの植物生理的役割がみえつつあり,稿を変えて紹介する機会を待ちたい.