真核多細胞生物におけるmRNA前駆体の選択的スプライシングは,タンパク質の多様性に寄与するのみではなく,中途の終止コドン(premature termination codon:PTC)を持つmRNAをあえて産生することによって遺伝子の発現量を制御するものがある.しかし,PTCを持つmRNAのスプライスバリアントはナンセンスコドン介在的mRNA分解(nonsense-mediated mRNA decay:NMD)と呼ばれる品質管理機構により速やかに分解されるため,一般的に不安定でほとんど検出されない.酵母や線虫を用いた遺伝学的な解析により,進化的に保存された必須のNMD因子としてUPF1, UPF2およびUPF3などが同定された.さまざまな生物で,NMDが欠損する状態で安定化されるmRNAを探索することにより,snoRNAのホスト遺伝子,偽遺伝子,長鎖非コード遺伝子,ウイルス遺伝子などの他,一部のmRNAスプライスバリアントが天然のNMD基質として同定された.多くのスプライシング制御因子では,NMDと共役した選択的スプライシングにより自身の遺伝子の発現量を負に自己制御することが知られている.
NMDと共役した選択的スプライシング制御の研究のモデルに線虫Caenorhabditis elegansを利用する利点として,NMD因子群が生存や生殖に必須でないために,天然のNMD基質を個体レベルでゲノムワイドに探索できる点があげられる.線虫のゲノムは脊椎動物などの高等動物と同様にイントロンに富んでおり,少なくとも25%以上のタンパク質遺伝子が何らかの選択的mRNAスプライシングを受ける.初期に行われたスプライスバリアント感受性マイクロアレイ解析により,線虫でNMDと共役した選択的スプライシングを受ける遺伝子が30個以上同定された.筆者らは,線虫でUPF1をコードするsmg-2遺伝子の変異体の大規模RNAシーケンシング(RNA-seq)解析を行い,リボソームタンパク質をコードする80個の遺伝子のうち8個がNMDと共役した選択的スプライシングにより発現量を負に自己制御することを報告した1).近年では,長寿の表現型を示す変異体線虫やカロリー制限により寿命が延長した線虫においてNMDと共役した選択的スプライシングが重要な役割を果たすことが示され,RNA-seq解析により長寿と関連した選択的スプライシングが同定されている.しかし,リード長が短いRNA-seq解析では,そのような天然のNMD基質となるmRNAの全長の配列を正確に再構成することが困難であった.
mRNAの転写後の塩基修飾は,遺伝子発現における新しい階層の制御機構となっている.そのような塩基修飾の一つであるN6-メチルアデノシン(m6A)修飾は,高等真核生物のmRNAの内部修飾において最も高頻度にみられるものである.一般にm6A修飾は可逆的であり,“writer”と呼ばれるメチル基転移酵素がmRNAをメチル化し,“eraser”と呼ばれる脱メチル化酵素がmRNAを脱メチル化する.mRNA中のm6A修飾のほぼすべては,methyltransferase-like 3(METTL3)/METTL14のヘテロ二量体からなるコアと他の制御サブユニットを含む複合体により形成される.m6A修飾は,RNAの局所的な構造を変化させてRNA結合タンパク質との相互作用を変化させたり,YTHDC1, YTHDF1やYTHDF2などの“reader”と呼ばれるタンパク質による特異的な認識を介してmRNAのスプライシング,核外輸送,翻訳開始や安定性に影響を与えたりする.RNA中のm6A修飾は,eraserであるFTOやALKBH5によって除去される.しかし,線虫においては上述のようなm6A writer, eraserやreaderのオルソログ遺伝子がゲノム中に見当たらず2, 3),生化学的解析でもmRNAのm6A修飾は限定的であることが示された.したがって,mRNAのm6A修飾が線虫のmRNA制御においてどのような役割を果たしているかについては不明であった.
筆者らは,線虫で発現する天然のNMD基質となるmRNAアイソフォームの全長配列を網羅的に明らかにするために,線虫でUPF1をコードするsmg-2遺伝子の変異体のL1ステージ幼虫から調製したポリ(A)+ RNAを用いて,Nanopore MinIONプラットフォームで長リード直接RNAシーケンシング解析を行った4).140万超のリードが線虫のゲノムにマップされ,少なくとも三つの長リードと以前のRNA-seqデータ1, 5, 6)に支持される12,517種類のmRNAアイソフォームが8028遺伝子から同定された.一方,比較として野生型株N2のさまざまな発生段階のポリ(A)+ RNAの直接RNAシーケンシングデータ等1, 5, 7)に同様の解析を行うと,351万超のリードがゲノムにマップされ,11,331遺伝子から18,376種類のmRNAアイソフォームが同定された.二つの株で共通に検出された6642個の遺伝子についてみてみると,smg-2変異体では解析したステージやリード数が限られているにもかかわらず,野生型株やWormBaseの予測にはない700個以上の新しいエクソン-エクソン境界リードが検出されており,smg-2変異体では通常では存在しないスプライスバリアントが安定化していることが示唆された.
複数のスプライスバリアントがあり少なくとも10リード以上が検出された2931遺伝子の8701種類のmRNAバリアントについて,smg-2変異体と野生型株で存在比率が異なるものを探索したところ,smg-2変異体で減少する208遺伝子の219バリアントと,smg-2変異体で増加する375遺伝子の420バリアントを見いだした.mRNAが最初のAUGコドンから翻訳されると仮定すると,smg-2変異体で増加するバリアントの3′非翻訳領域の長さの中央値は613塩基で,smg-2変異体で減少するバリアント(中央値194塩基)や両株で少なくとも10リード以上検出された4366遺伝子の全10,136バリアント(中央値218塩基)よりも有意に長かった.また,最下流のエクソン-エクソン境界から50塩基以上上流にある終止コドンをPTCだと仮定すると,smg-2変異体で増加するバリアントの68.8%がPTCを持つのに対して,smg-2変異体で減少するバリアントではたったの13.7%,検出されたすべてのバリアント中でも28.1%のみがPTCを持っていた.smg-2変異体で増加しPTCを持つバリアントがある12個の遺伝子について半定量的RT-PCRで解析したところ,イントロン保持型バリアントが安定化された1遺伝子を例外として,PTCを持つバリアントのsmg-2変異体における安定化が確認された.これらの結果から,PTCを持ちsmg-2変異体で増加する289個のバリアントを産生する259個の遺伝子が,線虫においてNMDと共役した選択的スプライシングにより発現量が制御される遺伝子であると強く考えられた.
上述の259遺伝子について遺伝子オントロジー解析を行うと,RNAの翻訳やプロセシングに関連した遺伝子の濃縮が明らかとなった.この結果は,哺乳類と線虫の両方においてリボソームタンパク質を含むRNA結合タンパク質の多くが自身の遺伝子発現をスプライシング段階で負に自己制御する,というこれまでの知見と整合するものである.
3. SAM合成酵素(sams)遺伝子の摂食と絶食による動的な選択的スプライシング制御
遺伝子オントロジー解析では,代謝に関連した遺伝子もNMDと共役した選択的スプライシングにより発現が制御される遺伝子として有意に濃縮されていた.そのような代謝関連遺伝子の解析の過程で,筆者らは,smg-2変異体におけるsams-3遺伝子,sams-4遺伝子とsams-5遺伝子のバリアントのうちPTCを持ちNMDで分解されるものの比率が,孵化したばかりのL1幼虫の摂食によって急上昇し,その後の絶食によって元に戻ることを見いだした.これらの遺伝子の発現が摂食状態に応じてスプライシング段階で動的に制御されることに何らかの生物学的意義があると考えられたことから,これらの遺伝子のスプライシング制御について,さらに詳しく解析することとした.
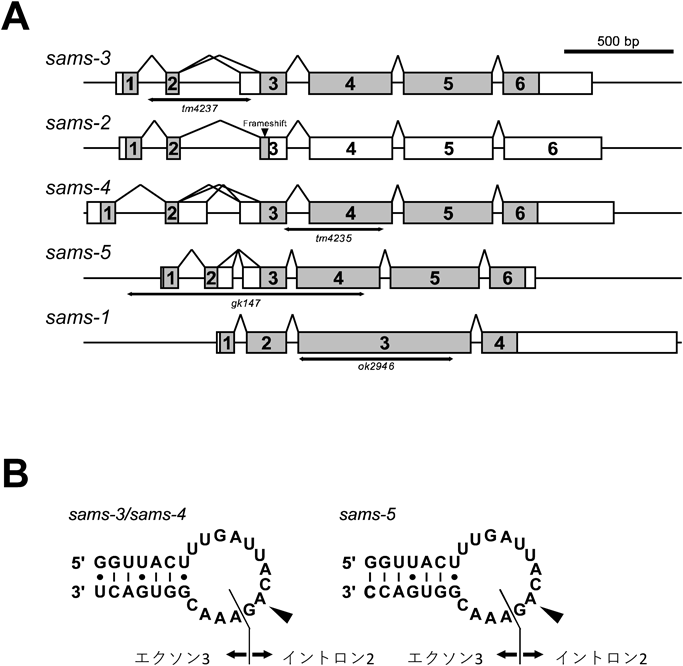
sams遺伝子はL-メチオニンとアデノシン三リン酸(ATP)からSAMを生成するSAM合成酵素をコードする遺伝子ファミリーである.線虫のゲノムには五つのファミリー遺伝子がコードされている(図1).sams-3とsams-4は互いに最も塩基配列の相同性が高く(sams-3とsams-4の間で87.5%の塩基配列同一性),エクソン構造も保存されている.sams-5もこれらとエクソン構造および選択的スプライシングパターンが保存されている.筆者らは,これら三つの遺伝子で選択的スプライシング制御を受けるイントロン2の塩基配列がイントロンとしては異例に配列が保存されていることを見いだした.対照的に,sams-1遺伝子はイントロン2を欠いており,構成的に発現すると考えられた.sams-2遺伝子はsams-3遺伝子と最も同一性が高い(ゲノム全体で94.2%の同一性)が,エクソン3にフレームシフト変異があることから偽遺伝子と考えられ,イントロン2の近位の3′スプライス部位に塩基置換変異があることから,構成的なスプライシングを受けると考えられた.
摂食によるsams遺伝子の選択的スプライシングパターンの変化にタンパク質の新規合成が必要か確かめるために,タンパク質合成阻害剤エメチンの存在下でsmg-2変異体に大腸菌を与えたところ,脂肪酸不飽和化酵素をコードするfat-7遺伝子の誘導と中鎖脂肪酸–アシルCoA合成酵素をコードするacs-2遺伝子の抑制は完全に抑圧された一方,sams遺伝子のスプライスバリアントの量と比率の変化はある程度まで誘導された.この結果から,sams遺伝子の摂食による動的な選択的スプライシング制御には,摂食に伴う新規のタンパク質合成を必要としないことがわかった.
4. SAM合成酵素(sams)遺伝子の選択的スプライシングのフィードバック制御
sams遺伝子のNMDと共役した選択的スプライシング制御の生物学的な意義に迫るため,SAM合成酵素活性がSAM合成酵素の発現を自己制御するかどうかを調べた.sams-1遺伝子は構成的に発現することから,smg-2変異体背景でsams-1遺伝子とsams-5遺伝子の二重破壊株におけるsams-3遺伝子とsams-4遺伝子の選択的スプライシングパターンを調べたところ,摂食前ではsmg-2変異体とsmg-2;sams-5;sams-1三重変異体でそれほど違いがみられなかったが,摂食後ではタンパク質をコードするバリアントの比率と量がsmg-2;sams-5;sams-1三重変異体で明らかに多くなっていた.また,SAM合成酵素の競合阻害剤であるシクロロイシンの投与によりわずかだが有意にタンパク質を産生するsams-3バリアントの比率と量が増加した.これらの結果から,SAM合成酵素活性がsams-3遺伝子とsams-4遺伝子のNMDと共役した選択的スプライシングを制御していることが明らかとなった.
SAM合成酵素をコードするバリアントの量の増加が実際にSAM合成酵素タンパク質の量の増加につながっているかどうかについて,四つのsams遺伝子産物を区別できる抗体を用いてウェスタンブロット法で確認したところ,smg-2変異体ではSAMS-3タンパク質とSAMS-4タンパク質の量が幼虫発生期を通じて一定であったのに対し,smg-2;sams-5;sams-1三重変異体ではSAMS-1とSAMS-5の欠損を補償するように幼虫発生期に次第に増加した.同様のSAMS-3とSAMS-4のmRNAとタンパク質の増加は,野生型背景のsams-1単独変異体でもみられた.これらの結果から,線虫におけるSAM合成酵素活性の恒常性は,NMDと共役したsams遺伝子の選択的スプライシング制御により維持されていることが明らかとなった.
5. METT-10が生体で摂食に応答したsams遺伝子の選択的スプライシングを制御する
SAMはタンパク質,DNA, RNA,脂質などをメチル化するさまざまなメチル基転移酵素の反応においてメチル基の主要な供与体であることから,筆者らは,SAM合成酵素活性の変動によるsams遺伝子の選択的スプライシングパターンの変動は特定の基質分子のメチル化レベルの変動を介したものと推測した.メチル化修飾されたヒストンがスプライシング制御因子をリクルートしたり転写の速度に影響したりすることで選択的スプライシングパターンが変化する例が哺乳類で知られていたことから,クロマチン免疫沈降-大規模シーケンス(ChIP-seq)法でヒストンH3のK4のジメチル化,K27のトリメチル化およびK36のトリメチル化を調べてみたが,smg-2変異体とsmg-2;sams-5;sams-1三重変異体でsams-3遺伝子座とsams-4遺伝子座での選択的スプライシングを受けるエクソンのメチル化状態に有意な差異はみられなかった.
次に,sams遺伝子のmRNA前駆体自体のメチル化を推測した.高等真核生物のmRNAの内部にみられる最も豊富な化学修飾はm6A修飾であるが,線虫ゲノムは哺乳類のmRNAのm6A修飾のほとんどを担うMETTL3, METTL14などのメチル基転移酵素複合体の構成因子のオルソログ遺伝子を欠いている2, 3).一方,哺乳類において別のメチル基転移酵素METTL16が,ヒトでSAM合成酵素をコードするMAT2A遺伝子の3′非翻訳領域の6個のヘアピン構造のループ内にあるUACAGARAAというコンセンサス配列の下線部のm6A修飾を特異的に触媒することが,近年に相次いで報告されていた8–10).筆者らは,sams-3遺伝子,sams-4遺伝子およびsams-5遺伝子のmRNA前駆体がMETTL16のコンセンサス配列をループ内に持つヘアピン構造をとりうること,しかも,m6A修飾を受けるアデニンがまさに選択的スプライシングを受けてSAM合成酵素を生成するための遠位3′スプライス部位にある不変のAGジヌクレオチドのAの位置に当たることに気がついた.そこで,METTL16の線虫オルソログであるMETT-10が生体でsams-3遺伝子,sams-4遺伝子およびsams-5遺伝子の選択的スプライシング制御に関与するか調べた.すると,摂食により誘導されるNMD基質となるスプライスバリアントの量は,smg-2単独変異体と比べてsmg-2;mett-10二重変異体で大きく減少していた.この結果は,METT-10がsams遺伝子の選択的スプライシングを,5’スプライス部位からみて遠位にありタンパク質をコードするバリアントを産生するための3′スプライス部位から,近位にありNMDで分解されるバリアントを産生するための3′スプライス部位へ摂食に応答してシフトさせるのに必須であることを示している.野生型背景のmett-10単独変異体においても,sams-3遺伝子およびsams-4遺伝子でタンパク質をコードするmRNAスプライスバリアントの量が増加し,それに応じてSAMS-3タンパク質およびSAMS-4タンパク質も野生型と比べて増加していた.これらの結果から,METT-10がsams遺伝子の選択的スプライシングの摂食に応答した負のフィードバック制御に必須であることが確認された.
6. METT-10は試験管内でsams遺伝子の遠位3′スプライス部位を特異的にm6A修飾する
筆者らは次に,線虫のMETT-10がsams遺伝子のmRNA前駆体を直接かつ特異的にメチル化できるか調べた.sams-3/sams-4遺伝子のイントロン2からエクソン3にまたがる127塩基の領域(両者の配列は同一)のRNAを試験管内転写で調製して,組換え全長METT-10タンパク質または組換えMETTL16メチルトランスフェラーゼドメインタンパク質を用いて試験管内メチル化反応を行ったところ,予期したとおり,両組換えタンパク質は遠位3′スプライス部位のAGのアデニンをSAM依存的に特異的かつ効率的にメチル化した.
7. 内在性のsams遺伝子mRNAは遠位3′スプライス部位にm6A修飾を持つ
最後に,筆者らは内在性sams遺伝子mRNAのm6A修飾について調べた.潜在的なm6A修飾部位はイントロン2の終端にあるため酵素をコードするバリアントではスプライシングで除去されてしまうが,NMD基質となるバリアントでは遠位3′スプライス部位は選択されずに成熟mRNA上に残っているはずである.m6A修飾特異的な抗体を用いたRNA免疫沈降と半定量的および定量的なRT-PCRの結果は,sams-3遺伝子,sams-4遺伝子,sams-5遺伝子のすべてにおいて酵素をコードするバリアントが相対的に減少し,NMDの基質となるバリアントが濃縮されていたことから,sams遺伝子mRNAのNMD基質バリアントに特異的な領域にm6A修飾部位が存在する,という仮説と合致した.
内在性sams遺伝子mRNAのm6A修飾についてさらに調べるため,先に述べた筆者らのRNA直接シーケンシングのデータを活用した.比較のために,161塩基のsams-3/sams-4遺伝子RNAおよび163塩基のsams-5遺伝子RNAを試験管内転写で調製し,さらに組換えMETTL16による試験管内メチル化反応で遠位3′スプライス部位のアデニンを特異的にm6A修飾したものも調製した.これら4種類の試験管内調製RNAをプールして,RNAがナノ小孔を通過する際に流れる電流を記録するNanopore社の直接RNAシーケンシング解析11)にかけた.標準的な塩基決定アルゴリズムとマッピングによりsams-3/sams-4遺伝子RNAとsams-5遺伝子RNAの配列は区別されたが,それぞれをm6A修飾したものと未修飾のものでは,振動する電流の平均値や標準偏差,通過時間は,期待したほどにははっきりと区別できなかった.そこで,13種類のアルゴリズムを使用して試験管内調製RNAのm6A修飾状態を分類器に機械学習させ,内在性sams遺伝子のNMD基質となるスプライスバリアントを分類させたところ,教師に用いたRNAの分類の正確性と同程度に相当する73~100%のリードがm6A修飾型であるとほとんどのアルゴリズムで判定された.これらの結果は,内在性sams遺伝子mRNAのNMD基質となるスプライスバリアントの,すべてとはいわないまでもほとんどのmRNAにおいて,遠位3′スプライス部位がm6A修飾されていることを示している.
8. AGジヌクレオチドのm6A修飾による3′スプライス部位の認識の阻害
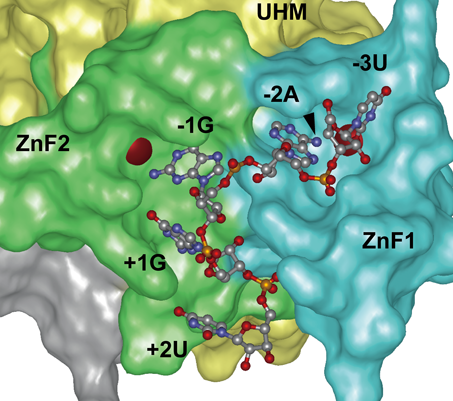
本研究成果は,3′スプライス部位の不変のジヌクレオチドであるAGのアデニンがm6A修飾されることで選択的スプライシングが制御されることを実験的に示した,すべての生物を通じて最初の例である.3′スプライス部位のAGは,タンパク質のヘテロ二量体からなる進化的に保存されたU2補助因子(U2 auxiliary factor:U2AF)の小サブユニットによって認識される.線虫では,3′スプライス部位のコンセンサス配列UUUUCAGが大サブユニットUAF-1と小サブユニットUAF-2からなるU2AFにより認識される.U2AF小サブユニットによる3′スプライス部位の認識機構については,分裂酵母Schizosaccharomyces pombeにおけるオルソログであるU2AF23と大サブユニットU2AF59の一部分からなる複合体によるRNA認識の結晶構造が解かれているのみであった12, 13).また,3′スプライス部位を模した配列5'-UUAGGU-3'の-2位のm6A修飾(UUm6AGGU)により,U2AF23複合体との親和性が劇的に減少することが試験管内の実験で示された12).3′スプライス部位のAGジヌクレオチドのm6A修飾が線虫UAF-2による3′スプライス部位の認識にも同様に影響するか明らかにするため,UAF-2と分裂酵母のU2AF23のアミノ酸配列相同性に基づいて,UAF-2による5'-UAGGU-3'配列認識の3次元構造のモデリングを行った.m6A修飾によりメチル化される−2位のアデニン塩基のアミノ基は,UAF-2においてもZnフィンガードメイン1の表面ポケットに埋め込まれており,分裂酵母のU2AF23でポケットを構成するのと同一のアミノ酸残基により密接に取り囲まれていた12)(図2).このモデルは,m6Aのreaderタンパク質が存在しない線虫でも3′スプライス部位の不変のAGジヌクレオチドのm6A修飾により3′スプライス部位としての認識を阻害できる,という我々の遺伝学的実験結果とよく合致した.哺乳類のmRNA前駆体のm6A修飾による選択的スプライシングの制御については相反する結果が報告されているが,3′スプライス部位の不変のAGジヌクレオチドのm6A修飾については,まだ報告例がない.
9. 線虫におけるSAM合成酵素の恒常性制御のモデル
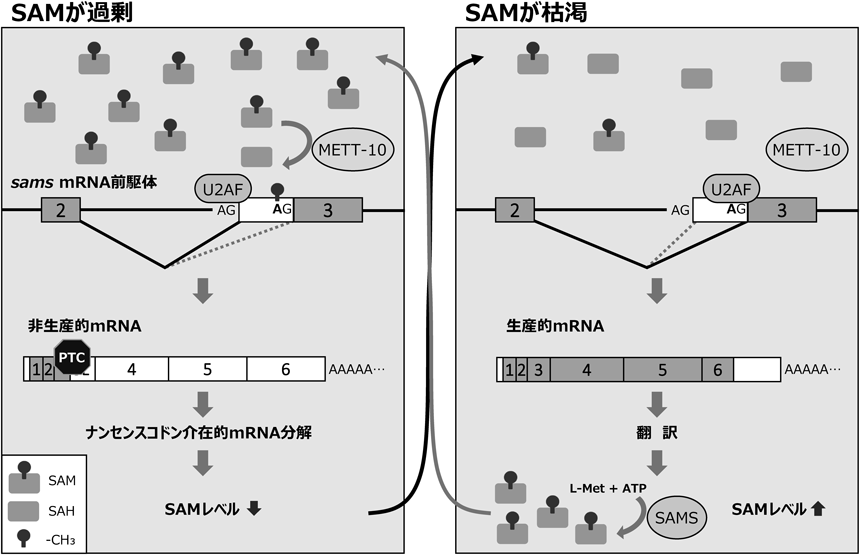
図3に,我々の実験結果に基づく線虫sams遺伝子のNMDと共役した選択的スプライシングによる恒常性維持機構のモデルを示す.SAMの濃度が低いときには,イントロン2の遠位の3′スプライス部位が未修飾のまま優先的に選択され,酵素をコードするmRNAが産生されて活性があるSAM合成酵素タンパク質に翻訳され,SAMレベルの上昇につながる.SAMの濃度が過剰なときには,メチル基転移酵素METT-10が遠位3′スプライス部位を特異的にm6A修飾することでスプライシングが阻害されて近位の3′スプライス部位が選択され,NMDの基質となるスプライスバリアントが産生される.
筆者らのこの研究と同時期に,スイスUniversity of GenevaのRamesh Pillaiらの研究グループは,筆者らとは別のアプローチで,しかし,実質的にはまったく同じ結論に達した14).彼らは抗m6A抗体を用いたRNA免疫沈降法により線虫でm6A修飾を持つRNAを濃縮し,mett-10変異体背景と野生型背景で異なる濃縮を示す部位としてsams-3遺伝子,sams-4遺伝子およびsams-5遺伝子の遠位3′スプライス部位を同定した.そして,mett-10変異体ではこの遠位3′スプライス部位を選択するmRNAが増加することから,METT-10による遠位3′スプライス部位のAGのアデニンのm6A修飾によりスプライス部位の選択が阻害されることを示した.また,試験管内でのメチル化反応により,METT-10によるこの遠位3′スプライス部位の認識にはRNAの局所的な二次構造が必要であることを示した.食餌である大腸菌の量によってもスプライスバリアントの比率が変化することを示し,大腸菌量が少ない場合でもメチオニンやビタミンB12の添加によりスプライスバリアントの比率が変化しうることも示した.m6A修飾により分裂酵母のU2AFとの親和性が大きく低下すること,m6A修飾によりヒト培養細胞株抽出液による線虫sams遺伝子の試験管内スプライシング反応が阻害されることなども示した.彼らの論文ではNMDとスプライシング制御の関係については解析しておらず,筆者らが行った実験とはほとんど重複がないものの,実質的に同じ結論を支持しており,不変の3′スプライス部位のm6A修飾による選択的スプライシングの制御という新奇の機構が独立の研究でそれぞれ生化学的・遺伝学的に明らかにされた点で大変興味深い.
10. 線虫におけるSAM合成酵素の恒常性制御のモデル
ヒトの培養細胞では,先に述べたように,SAM合成酵素をコードするMAT2A遺伝子のmRNAレベルが,SAMの濃度により2種類の機構で間接的にフィードバック制御されている.一つは,METTL16がMAT2A mRNAの3′非翻訳領域のヘアピン構造に結合することで,スプライシングされにくい最下流のイントロンのスプライシングを促すというもので,METTL16の酵素活性に依存せずヘアピンへの結合時間が延びることでSAM合成酵素をコードするmRNAの産生が促進される8).もう一つは,SAM過剰時に3′非翻訳領域に位置する6個のヘアピン構造がMETTL16によりm6A修飾されることで,核内のm6A “reader”タンパク質であるYTHDC1を介したmRNA分解が促進される9, 15).SAMはさまざまなメチル化反応において主要なメチル基の供与体であることから,さまざまな生物においてSAMレベルの維持が生物学的に重要であることが報告されている.今回の筆者らの論文を含め,さまざまな生物でSAM合成酵素の発現がSAMのレベルにより間接的にフィードバック制御を受けていることは,その恒常性維持の重要性を裏書きしている.
引用文献References
1) Takei, S., Togo-Ohno, M., Suzuki, Y., & Kuroyanagi, H. (2016) Evolutionarily conserved autoregulation of alternative pre-mRNA splicing by ribosomal protein L10a. Nucleic Acids Res., 44, 5585–5596.
2) Arribere, J.A., Kuroyanagi, H., & Hundley, H.A. (2020) mRNA Editing, Processing and Quality Control in Caenorhabditis elegans. Genetics, 215, 531–568.
3) Cunningham, F., Achuthan, P., Akanni, W., Allen, J., Amode, M.R., Armean, I.M., Bennett, R., Bhai, J., Billis, K., Boddu, S., et al. (2019) Ensembl 2019. Nucleic Acids Res., 47(D1), D745–D751.
4) Watabe, E., Togo-Ohno, M., Ishigami, Y., Wani, S., Hirota, K., Kimura-Asami, M., Hasan, S., Takei, S., Fukamizu, A., Suzuki, Y., et al. (2021) m6 A-mediated alternative splicing coupled with nonsense-mediated mRNA decay regulates SAM synthetase homeostasis. EMBO J., 40, e106434.
5) Son, H.G., Seo, M., Ham, S., Hwang, W., Lee, D., An, S.W., Artan, M., Seo, K., Kaletsky, R., Arey, R.N., et al. (2017) RNA surveillance via nonsense-mediated mRNA decay is crucial for longevity in daf-2/insulin/IGF-1 mutant C. elegans. Nat. Commun., 8, 14749.
6) Kuroyanagi, H., Watanabe, Y., Suzuki, Y., & Hagiwara, M. (2013) Position-dependent and neuron-specific splicing regulation by the CELF family RNA-binding protein UNC-75 in Caenorhabditis elegans. Nucleic Acids Res., 41, 4015–4025.
7) Roach, N.P., Sadowski, N., Alessi, A.F., Timp, W., Taylor, J., & Kim, J.K. (2020) The full-length transcriptome of C. elegans using direct RNA sequencing. Genome Res., 30, 299–312.
8) Pendleton, K.E., Chen, B., Liu, K., Hunter, O.V., Xie, Y., Tu, B.P., & Conrad, N.K. (2017) The U6 snRNA m6A Methyltransferase METTL16 Regulates SAM Synthetase Intron Retention. Cell, 169, 824–835.e14.
9) Shima, H., Matsumoto, M., Ishigami, Y., Ebina, M., Muto, A., Sato, Y., Kumagai, S., Ochiai, K., Suzuki, T., & Igarashi, K. (2017) S-Adenosylmethionine Synthesis Is Regulated by Selective N6-Adenosine Methylation and mRNA Degradation Involving METTL16 and YTHDC1. Cell Rep., 21, 3354–3363.
10) Doxtader, K.A., Wang, P., Scarborough, A.M., Seo, D., Conrad, N.K., & Nam, Y. (2018) Structural Basis for Regulation of METTL16, an S-Adenosylmethionine Homeostasis Factor. Mol. Cell, 71, 1001–1011.e4.
11) Garalde, D.R., Snell, E.A., Jachimowicz, D., Sipos, B., Lloyd, J.H., Bruce, M., Pantic, N., Admassu, T., James, P., Warland, A., et al. (2018) Highly parallel direct RNA sequencing on an array of nanopores. Nat. Methods, 15, 201–206.
12) Yoshida, H., Park, S.Y., Sakashita, G., Nariai, Y., Kuwasako, K., Muto, Y., Urano, T., & Obayashi, E. (2020) Elucidation of the aberrant 3′ splice site selection by cancer-associated mutations on the U2AF1. Nat. Commun., 11, 4744.
13) Yoshida, H., Park, S.Y., Oda, T., Akiyoshi, T., Sato, M., Shirouzu, M., Tsuda, K., Kuwasako, K., Unzai, S., Muto, Y., et al. (2015) A novel 3′ splice site recognition by the two zinc fingers in the U2AF small subunit. Genes Dev., 29, 1649–1660.
14) Mendel, M., Delaney, K., Pandey, R.R., Chen, K.M., Wenda, J.M., Vagbo, C.B., Steiner, F.A., Homolka, D., & Pillai, R.S. (2021) Splice site m6A methylation prevents binding of U2AF35 to inhibit RNA splicing. Cell, 184, 3125–3142.e25.
15) Bresson, S.M., Hunter, O.V., Hunter, A.C., & Conrad, N.K. (2015) Canonical Poly(A) Polymerase Activity Promotes the Decay of a Wide Variety of Mammalian Nuclear RNAs. PLoS Genet., 11, e1005610.
著者紹介Author Profile
 黒柳 秀人(くろやなぎ ひでひと)
黒柳 秀人(くろやなぎ ひでひと)琉球大学大学院医学研究科生化学講座教授.博士(理学).
略歴1994年東京大学理学部卒業.99年同大学院理学系研究科修了.2000年東京医科歯科大学助手.03年同講師.08年同准教授.21年より現職.
研究テーマと抱負真核生物の遺伝子発現制御機構,特に,mRNA前駆体の転写後プロセシングを制御する個体レベルでの「細胞暗号」の解明などの基礎研究と,遺伝子変異や遺伝子発現制御機構の破綻に起因する疾患の病態発現機序の解明および治療法の開発を目指して研究を展開している.
ウェブサイトhttp://biochem.med.u-ryukyu.ac.jp/
趣味街歩き.