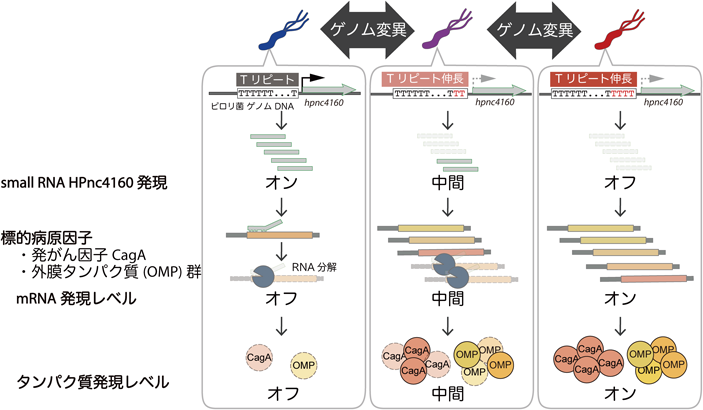
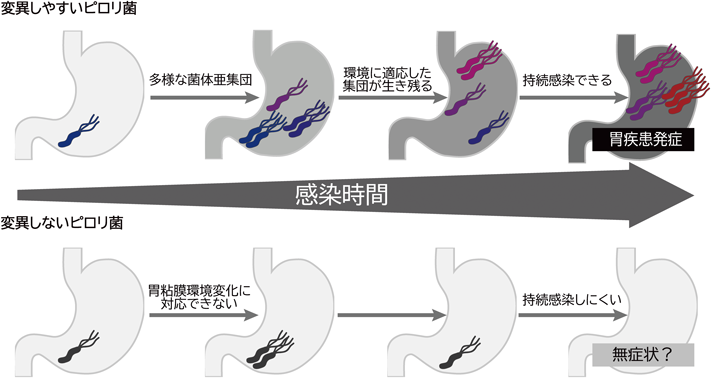
ピロリ菌の持続感染メカニズムPersistent infection mechanism of Helicobacter pylori
大分大学グローカル感染症研究センターゲノムワイド感染症研究部門Division of Genome-wide Infectious Microbiology, Research Center for GLOBAL and LOCAL Infectious Diseases, Oita University ◇ 大分県由布市挾間町医大ヶ丘1–1 ◇ 1–1, Hasama machi, Yufu city, Oita, Japan