ヒトの消化管には約1000菌種,数百兆個もの腸内細菌が生息しており,腸内細菌叢(腸内細菌の集団)は宿主と相互作用することで,複雑な腸内生態系を形成している.これまでに腸内細菌叢はヒトの健康に関係することが知られているが,その菌叢バランスの変化がさまざまな疾患に関連していることも次々に明らかになってきている.
一方,腸内細菌は哺乳類とは異なる代謝反応によって,生体内の分子もしくは食事などの外来由来の分子を構造変換することで,多種多様な代謝物を腸管内で産生している.たとえば,腸内細菌が産生する有機酸やアミノ酸に代表される親水性の一次代謝物や,フラボノイドなどの二次代謝物の分解産物が生理活性分子として,宿主の腸管における免疫,上皮バリア,ウイルス感染などの制御に関与することが近年報告されている1–4).
また,脂質代謝物については,腸内細菌によって一次胆汁酸が二次胆汁酸に代謝されて腸肝循環することは古くから知られているが,その他にも食事中の不飽和脂肪酸が哺乳動物の代謝系で生成しない分子構造に変換されることで,宿主の生体恒常性の維持や疾患の発症に関わることが明らかになってきている5, 6).さらに,腸内細菌膜の脂質分子の一部には,宿主受容体との相互作用によって機能調節に関与するなど7),腸内細菌自体だけでなく腸内細菌から産生されるさまざまな活性分子を介して,宿主に全身的な影響を与えるという新たなパラダイムシフトが生まれつつある.
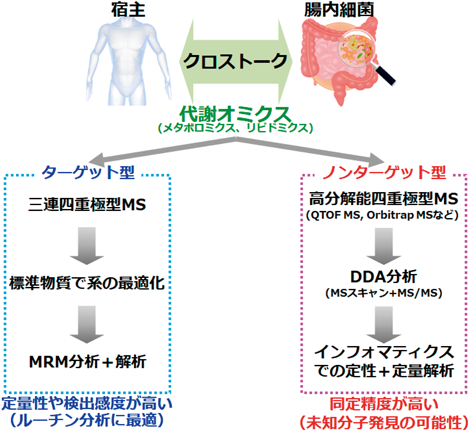
こうした研究の進展には,次世代シーケンサーを用いたメタゲノム解析や,無菌動物に特定の菌を定着させてその機能を解析するノトバイオート技術に加え,質量分析計(MS)や核磁気共鳴(NMR)等を用いた低分子の代謝物質の分析技術の進展が大きな役割を果たしている.このような背景のもと,腸内細菌と宿主との共生関係から生まれる代謝クロストークの総体を分子レベルで捉えようとする代謝オミクス研究(メタボロミクス,リピドミクス)が注目されている(図1).本稿では,腸内細菌の産生する代表的な代謝物質や,これらの分子について解析するオミクス技術を紹介する.
代謝オミクス(メタボロミクス,リピドミクス)は,生体内・外に存在する分子を網羅的かつ定量的に解析して,代謝変化の背後に関わる分子の抽出を行い,表現型や生体機能との関連性を明らかにする研究である.代謝物は物性の違いによって,親水性と脂溶性の分子に大別され,前者を対象とする研究はメタボロミクス,後者はリピドミクスに分類される.
さらに,これらのオミクスの解析アプローチとしては,主にターゲット(フォーカス)型とノンターゲット(ノンバイアス)型に大別される(図1).ターゲット法では,対象分子について選択性の高い分離・分析法や最適化された前処理法を組み合わせることによって,生体中に微量(数nMあるいはそれ以下)に存在する代謝物の包括的な検出や,標的パスウェイの代謝物群の高感度な探索に適している(補足説明1参照).
一方でターゲット解析は,分析前にあらかじめパラメーター設定した代謝物のみが解析対象となって探索範囲が限られるために,代謝物質の複雑性の高い腸内細菌叢で網羅的に探索する目的には不十分である.これとは対照的に,ノンターゲット解析ではあらかじめ対象分子を絞り込まず,代謝物の定性および定量的なデータを同時的に取得して,インフォマティクス技術を駆使して未知分子を含めた同定と網羅的な代謝変動のスクリーニングが可能になってきている(図1)(補足説明2参照).
以上のように,代謝オミクス研究を進める際には,対象とする代謝物の範囲を設定するとともに,それらの性質や存在量に基づいて,解析アプローチを選択することが重要となる.このため,以降では腸内細菌や宿主との共生代謝を探索する際に適用されるメタボロミクスやリピドミクスの代表的なプラットフォームについて概説する.
補足説明1 ターゲット解析とは
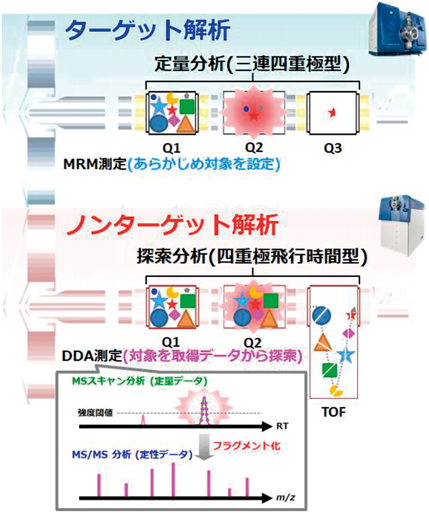
ターゲット解析では,三連四重極型(Triple-Q型)MSを用いたmultiple reaction monitoring(MRM)と呼ばれる分析法が適用されることが多い(図2上).この手法では,最初の四重極(Q1)で,分析対象の分子イオンを選択的に通過させ,次の四重極(Q2)でcollision-induced dissociation(CID)により生じたフラグメントイオンのうち,その中から特定の部分構造を持つイオンを最後の四重極(Q3)で選択的に通過させ検出することで,その分子構造特異的な検出を行うことが可能である.このような対象分子の選択性の高い分析によって,ターゲット解析は検出感度や定量性が優れており,生体中に微量で活性を発揮するような脂質メディエーターなどの深度の高い探索に適している.
補足説明2 ノンターゲット解析とは
ノンターゲット解析では,四重極飛行時間型(Q-TOF)や四重極オービトラップ型(Q-Orbitrap)型などの高分解能なMSが用いられ,分子検出の網羅性や分子構造の探索性に優れている(図2下).よく用いられる分析方法としては,最初にサンプル中に含まれる分子の一斉検出(MSスキャン分析)を行い,次に設定した強度閾値を超えたものについて自動的に構造分析(MS/MS分析)が適用される.たとえば,このdata dependent acquisition(DDA)モードと呼ばれる分析法をマウス便に応用した場合,膨大なMSスキャンデータ(約10,000個)およびMS/MSデータ(約6000個)が1回の測定で入手可能である8).
短鎖脂肪酸は,食物繊維を基質として腸内細菌の発酵によって産生される代表的な代謝物であり,中でもプロピオン酸や酢酸などは短鎖脂肪酸受容体のGPR41やGPR43のリガンドとして,宿主のエネルギー代謝恒常性に関与している9–11).また,酪酸は未成熟T細胞のFoxp3遺伝子領域のヒストンアセチル化を促進して遺伝子の発現をオンに切り替えることによって,制御性T細胞への分化を促して腸管の炎症を抑制するなど,免疫機能さらにはエピゲノム制御にまで影響を及ぼすことが知られている2, 3).
アミノ酸代謝に関しては,腸内細菌によってトリメチルアミン(trimethylamine:TMA)が産生され,腸管で吸収されて肝臓へ移行した後に,アテローム性動脈硬化を促進するトリメチルアミンN-オキシド(trimethylamine N-oxide:TMAO)に変換されることが明らかとなっている(腸肝連関)12, 13).また,トリプトファンは腸内でインドールへと変換され,その後に肝臓でシトクロムP450(CYP2E1やCYP2A6)によってインドキシルが生成され,さらに硫酸転移酵素(SULT1A1)でインドキシル硫酸が産生される.このインドキシル硫酸は,代表的な尿毒素の一つであり,インドールおよびインドキシル硫酸が腎不全の進行促進の原因の一つとして考えられている(腸腎連関)14).
植物二次代謝物であるフラボノイドは,腸内細菌によってデスアミノチロシン(desaminotyrosine:DAT)に分解され,このDATがI型インターフェロン依存的にインフルエンザウイルスに対する抵抗性を高めていることが明らかとなっている15).
以上のように,メタボロミクスで対象となる腸内細菌由来の親水性代謝物は,腸管での機能のみならず,一部は吸収されて血中に移行して全身を巡るために,メディエーターとしての役割を通じて宿主の多臓器間ネットワークの解明が今後さらに進むと考えられる.
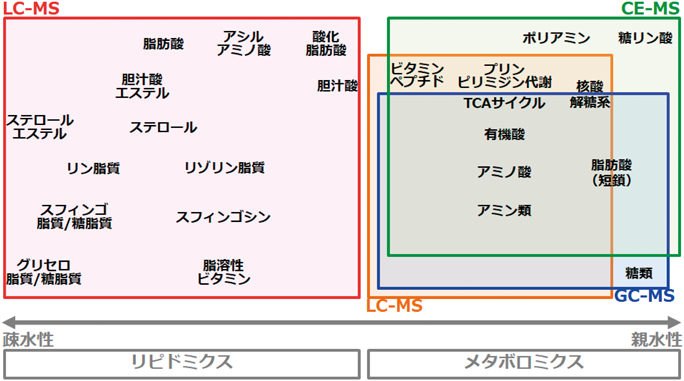
メタボロミクスは,リピドミクスに比べて個々の代謝分子の標準物質が比較的整備されており,ターゲット解析が主流となっているが,分析装置自体の選択肢が多いために,実験目的や対象に適したアプローチを選ぶ必要がある(図3右).この分析装置の種類としては,ガスクロマトグラフ質量分析計(GC-MS),液体クロマトグラフ質量分析計(LC-MS),キャピラリー電気泳動質量分析計(CE-MS),核磁気共鳴(NMR)が主に使われる.中でも,MSは非常に高感度であるために,メタボロミクス研究において現在主流の検出法であるが,対象の物性や対象範囲によって前段の分離装置部(LC, GC, CE)を選択する必要がある.
GC-MSは,分析システム上の堅牢性や保持時間の再現性も高く,短鎖脂肪酸やアミノ酸などの親水性の一次代謝物のような揮発性分子の測定には優れているが,糖リン酸類のようなリン酸基を持つ分子の検出は技術的に難しい.また,短鎖脂肪酸を含めて一次代謝物を探索する場合,メタボロミクスで包括的な検出に用いられことが多いMSTFA(N-methyl-N-trimethylsilyl-trifluoroacetamide)誘導体化よりは,前処理の安定性でMTBSTFA[N-methyl-N-(tert-butyldimethylsilyl)trifluoroacetamide]などが適している16).
LC-MSについては,高感度で幅広い極性分子の解析が可能であり,親水性もしくは疎水性相互作用によって,順相系もしくは逆相系でLC分離する場合が多い17).また,これらで用いるカラムの充填剤・官能基によっても対象範囲が絞られるため,広範囲な極性分子の探索を行う場合(たとえば,一次および二次代謝物を網羅的かつ高深度に調べたい場合)には,複数の分離条件を組み合わせる必要がある.さらに,LCの移動相には酢酸やギ酸を添加することが多いために,短鎖脂肪酸も対象の一部とする場合は,あらかじめ3-ニトロフェニルヒドラジン(3-NPH)などで誘導体化後にLC分析したり18),GC-MSを別途併用したりするなどの工夫が求められる.
CE-MSは非イオン性分子に不向きであるものの,腸内細菌が産生する短鎖脂肪酸,ビタミン類,ポリアミンなどが一度に幅広く測定できる利点がある19, 20).一方で,分析システム上で堅牢性や再現性が他の手法より低いために,データ集積の際に注意が必要である.
NMRに関しては,MSと比較して検出感度は低いが,MSではあまり適さない非イオン性分子を含めた幅広い分子の探索ができるだけでなく,非侵襲的に細胞などを生きたまま測定できる利点がある3).また,装置の安定性やスペクトルの再現性が高いため,継続的な分析や大規模なデータ集積にも適している21).
なお,腸内細菌が産生する代謝物ではなく,腸内細菌自体の帰属種・近縁種を同定する技術として,マトリックス支援レーザー脱離イオン化質量分析計(MALDI-MS)が活用されており,菌体から約2000~20,000 Da相当のタンパク質に由来するスペクトルをデータベースと照合することで,低コストかつ短時間で判別が可能になってきている.
脂質は生体膜を構成する細胞の基本構成要素であり,エネルギー源としての役割に加え,シグナル分子として働くなど多彩な機能を担っている.腸内細菌はグリセロリン脂質,スフィンゴ脂質,アシルグリセロール,ステロールなどの多様な極性分子から構成されており(図3左),脂肪酸自体の結合様式や,その分子内に含まれる二重結合数や位置なども多岐にわたる.
また,食事由来の脂質の一部は,腸内細菌によって異なる脂質へと構造変換されて,新たな働きをすることが知られている(脂質分子の機能の詳細については有田の稿「腸内細菌が産生する脂質代謝物の構造と機能」を参照されたい).たとえば,乳酸菌は飽和化反応を利用して,不飽和脂肪酸から哺乳動物の代謝系で生成しない水酸化脂肪酸,オキソ脂肪酸,共役脂肪酸などを産生することがわかっている5).中でも,リノール酸由来の水酸化脂肪酸であるHYA(10-hydroxy-cis-12-octadecenoic acid)は,GPR40やGPR120を介して腸管上皮細胞のバリア機能の増強効果やインスリン抵抗の改善効果などを持つことが明らかになってきている6).
胆汁酸に関しては,大腸の特定の腸内細菌によって一次胆汁酸から二次胆汁酸へと変換され,中でもデオキシコール酸は肝臓のがん化促進や病原性細菌の排除に関わっていることが知られている22).また,T細胞において3-oxo-LCAがTh17細胞の分化を抑制し,isoalloLCAは制御性T細胞の分化を促進するなど,二次胆汁酸は腸管免疫の恒常性維持にも寄与している23).
さらに,腸内細菌膜の脂質分子の一部には,宿主受容体との相互作用によって機能調節に関与するなど24, 25),腸内細菌自体もしくは腸内細菌から産生されたさまざまな活性代謝物を介して,宿主に全身的な影響を与えるという発見が,リピドミクス技術を通じて生まれてきている.
1)分析基盤技術
腸内細菌がさまざまな脂肪酸の構造異性体や複雑な脂質複合体なども産生することが明らかとなってきている一方で,これらの脂質分子の多くについて標準物質を入手するのは困難である.このような背景から,腸内細菌と宿主との複雑な脂質代謝クロストークを網羅的に捉えるには,従来のようなあらかじめ入手可能な脂質の標準物質にターゲットを絞り込んだアプローチでは技術的に困難なため,未知を含めた構造スクリーニングも可能なノンターゲット解析が不可欠である.
筆者らは宿主に加えて腸内細菌の脂質代謝を捉えられる探索性に優れたリピドミクス技術の開発や社会実装化を進めている8, 26, 27).このリピドミクスには,高分解能のLC-MSが用いられ,取得した測定データから遡って解析対象の脂質代謝物を網羅的にスクリーニングするというノンターゲット型のリピドミクスが基盤技術として用いられ,「前処理」・「分離」・「分析」・「解析」という四つの主要な技術要素から構成されている26).
ノンターゲット解析では,探索範囲を広くするために,対象サンプルからできるだけ多くの種類の脂質を回収する必要がある.一方,脂質はさまざまな極性を持つために,代表的な脂質の二層分配抽出法であるBligh & Dyer法では,脂肪酸代謝物などの高極性分子は油層でなく水層に一部移行するなど総脂質を一斉回収するのが困難である.このため,総脂質の調製法として,一層系の抽出溶媒(たとえば,クロロホルム,メタノール,水の体積比率が1 : 2 : 0.2)を用いている.
得られた総画分には高~低極性の脂質分子が含まれているため,分離能が高い微粒子径(1.7 µm)のオクタデシルシリル(ODS)カラム(例:ACQUITY UPLC BEH, 130 Å, 2.1 mm×50 mm/Waters社)など用いて,逆相系の超高速液体クロマトグラフィー(HPLC)システム(例:ACQUITY UPLC/Waters社)で一斉分離を行う必要がある.その際に,より幅広い極性の脂質の分離に対応した移動相およびグラジェントの条件を設定することが求められる.
一斉分離した脂質は,高分解能なMS[例:Q TOF-MS(Triple TOF 6600システム/SCIEX社)]を用いたDDAモード測定を行い,MSスキャン分析で設定ピーク強度閾値を超えた分子について,同時的にMS/MS分析でフラグメント化をして,部分構造の情報(MS/MSスペクトル)の入手を行う.
2)データ解析基盤技術
ノンターゲット解析においては,このように得られた膨大なMS/MSデータから,いかに網羅的かつ高精度に分子構造の帰属を進めるかが重要となる.この構造スクリーニングには,インフォマティクス技術によるハイスループットな脂質同定システムが有効かつ効率的である.しかしながら,脂質の場合は構成する脂肪酸のバリエーションなどにより膨大な分子種が存在し,それぞれの標準物質を入手することは困難なために,同定精度や網羅性の高い自動的な探索システムを構築や活用が重要となる.
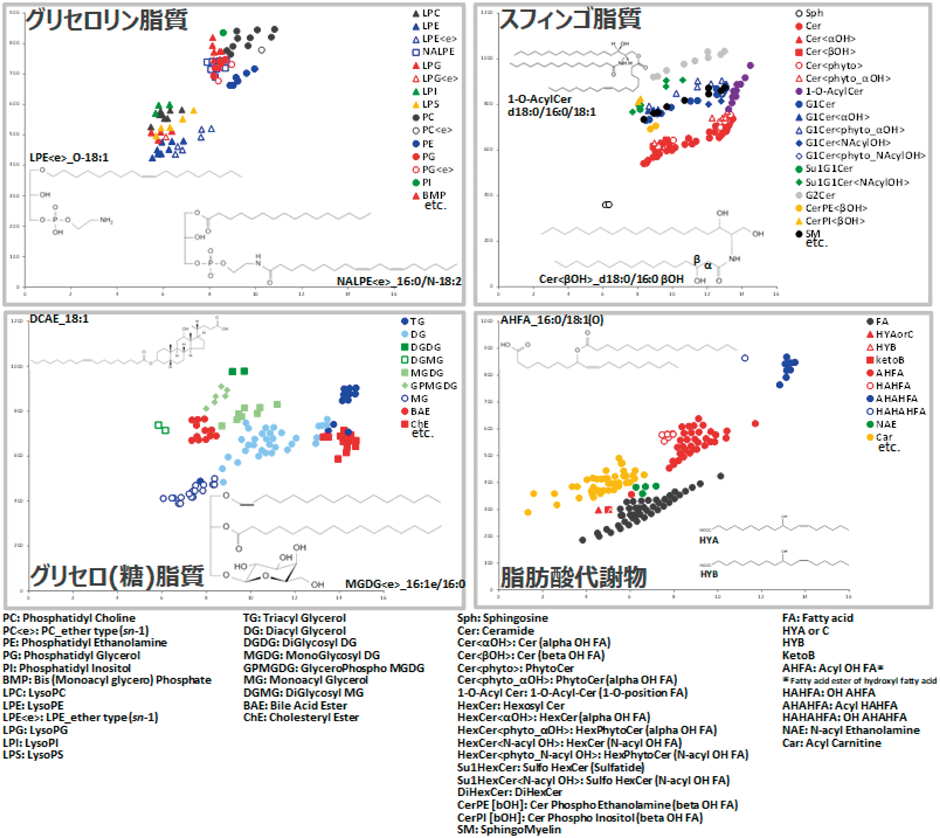
現在,このようなノンターゲット解析に志向に対応したMS-DIALなどのソフトウェアの開発も進んでおり,脂質分子の網羅的な同定やプロファイリング情報を一括処理して行うことが可能になっている26).また,従来のMS/MSサーチでは,同定精度の低いin silicoの仮想的なMS/MSデータベースが用いられてきたが,MS-DIALなどでは腸内細菌等の実測データに基づいたin silicoのデータベースや,スペクトルデータの重みづけ理論を適用することで,精度の高い同定が可能になってきている.たとえば,マウスの便のノンターゲット解析では約850分子が同定され,その中で抗生物質投与によって検出量が10分の1以下に減少するような腸内細菌由来の代謝物であることが強く示唆されるものが約230分子見つかっている8).これらの分子には,エーテル結合含有グリセロ脂質,極性頭部にホスホエタノールアミンやホスホイノシトールなどの修飾を持つスフィンゴ脂質,脂肪酸がアミノ酸で抱合されたN-アシルアミドなどの多様な構造の脂質分子群が含まれていることが明らかとなっている.また,ヒトの便のノンターゲット型リピドミクス解析では,これらの分子を含んだ約1000分子が共生総体として検出できており,2Dマッピング(横軸:LC保持時間,縦軸:m/z)から,主にグリセロリン脂質,グリセロ(糖)脂質,スフィンゴ脂質,脂肪酸代謝物から構成されていることがわかる(図4).
一方,既存のデータベースではいまだ腸内細菌によって産生される未同定分子が数多く残されており,この解決策として未知の脂質分子の構造推定には,MS/MSフラグメントの類似度でクラスタリングするfeature-based molecular spectrum networking(FBMN)や,他のデータベースと情報連携したMS-FINDERのような構造推定ツールが有用である28, 29).たとえば,FBMN解析から腸内細菌由来の新規代謝物として,酪酸などの短鎖脂肪酸がα位にヒドロキシ基のある極長鎖脂肪酸とエステル結合したacyl alpha-hydroxy fatty acid(AAHFA)が発見されている8).
3)構造解析基盤技術
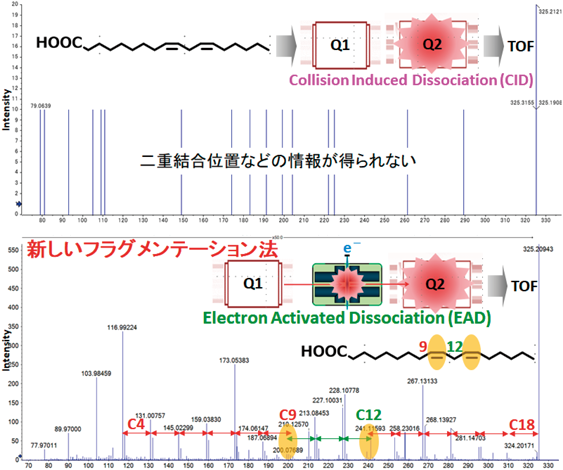
このような解析プラットフォームの拡充の一方で,今後さらに腸内細菌由来の脂質分子の同定効率を上げる方法として,MSでの新規の分子フラグメンテーション技術が注目されている.従来のCID法によるMS/MSスペクトルのデータでは,脂肪酸の二重結合や水酸基などの分子内の保有数はわかるが,これらの位置情報について得ることが困難である(図5上).腸内細菌は種類によって二重結合位置や数が異なる分子が産生され,生理活性の評価上でも詳細な構造情報が重要となるが,近年では,二重結合位置に特異的な解離を引き起こすoxygen attachment dissociation(OAD)が開発されている30, 31).さらに,シークエンシャル的な開裂によって二重結合や水酸基の位置などが判別できるelectron activated dissociation(EAD)(図5下)やultraviolet photodissociation(UVPD)の開発も進み,細菌細胞膜の主成分の一つであるグリセロリン脂質中の脂肪酸の二重結合や水酸基などの位置のみならず,シクロプロパン環や新規のヘッドグループの構造情報を得ることも可能になってきている32–34).現状では,これらの解析技術には従来法に比べて多くのサンプル量を必要とするが,今後高感度化が進めば腸内細菌研究において強力なツールとなると考えられる.
4)ターゲット解析を組み合わせる重要性
これまでリピドミクスについてノンターゲット解析の重要性について言及したが,MSのダイナミックレンジ(検量線の直線範囲)はおおよそ4桁前後であり,生体サンプル中に存在する分子の濃度域と比べると,MSの検出可能域が十分でない.このため,あらかじめ対象分子の標準物質の入手が可能で,サンプル中に極微量しか存在しない場合は,最適な前処理や分離条件の下でのターゲット解析が適している.特に,腸内細菌から代謝される胆汁酸や脂肪酸のように構造異性体が多く,微量な分子も含まれている場合は,ターゲット解析でより高感度かつ選択的に検出を行うことが望ましいと考えられる.