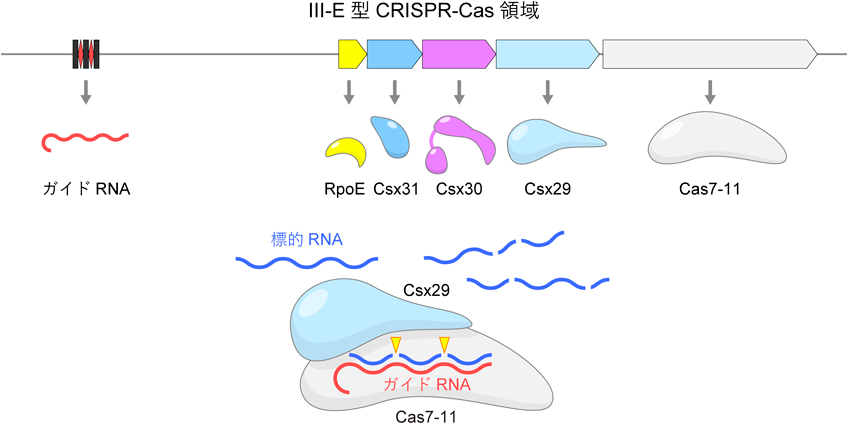
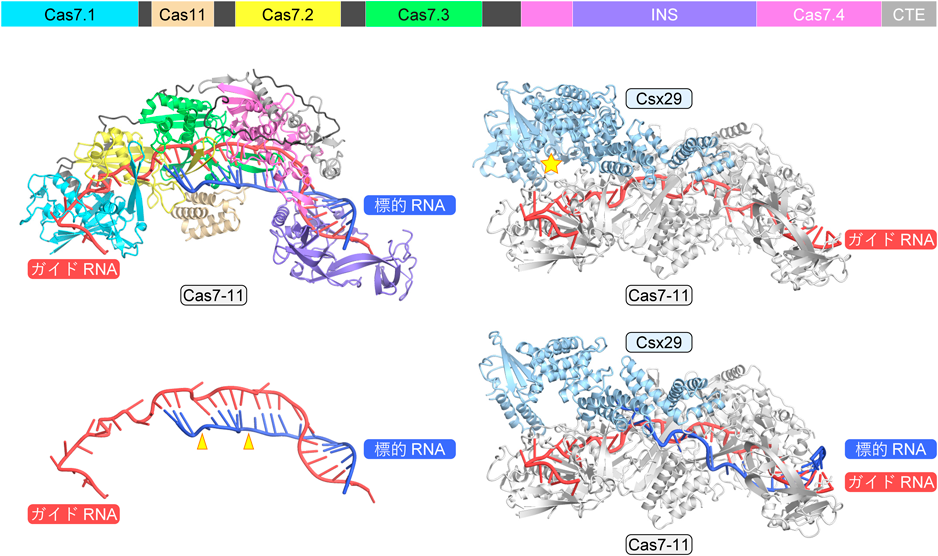
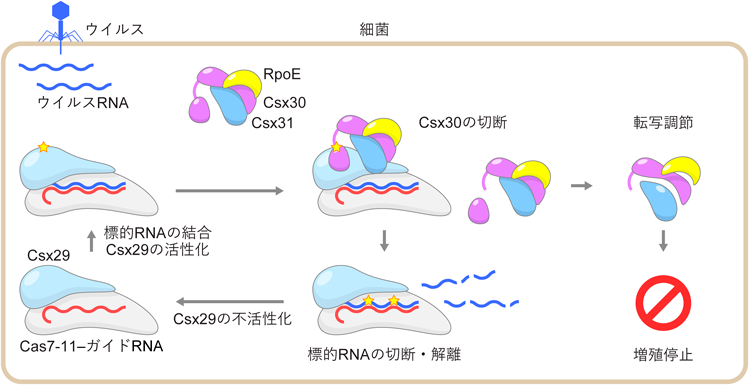
Cas7-11–Csx29ヌクレアーゼ–プロテアーゼ複合体の構造,機能,応用Structure, function, and engineering of Cas7-11–Csx29 nuclease–protease complex
1 東京大学先端科学技術研究センターStructural Biology Division, Research Center for Advanced Science and Technology, The University of Tokyo ◇ 〒153–8904 東京都目黒区駒場4–6–1 ◇ 4–6–1 Komaba, Meguro-ku, Tokyo 153–8904, Japan
2 東京大学大学院工学系研究科化学生命工学専攻Department of Chemistry and Biotechnology, Graduate School of Engineering, The University of Tokyo ◇ 〒113–8656 東京都文京区本郷7–3–1 ◇ 7–3–1 Hongo, Bunkyo-ku, Tokyo 113–8656, Japan