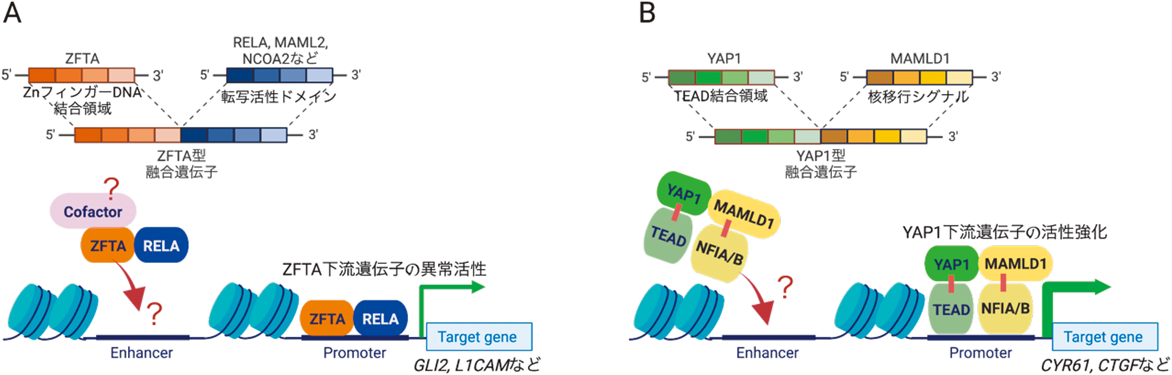
小児脳腫瘍における融合遺伝子の発がん制御Oncogenic regulation of supratentorial ependymoma-specific fusion genes
1 国立研究開発法人 国立精神・神経医療研究センター病態生化学研究部・細胞生化学研究室Department of Biochemistry and Cellular Biology, National Center of Neurology and Psychiatry (NCNP), National Institute of Neuroscience ◇ 〒187–8502 東京都小平市小川東町4–1–1 神経研究所 ◇ 4–1–1 Ogawahigashi-cho, Kodaira, Tokyo 187–8502, Japan
2 信州大学大学院・総合医理工学研究科Department of Biomedical Engineering, Graduate School of Medicine, Science and Technology ◇ 〒390–8621 長野県松本市旭3–1–1 ◇ 3–1–1 Asahi, Matsumoto, Nagano 390–8621, Japan