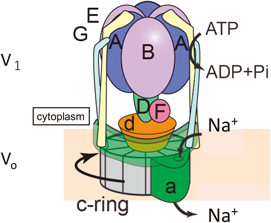
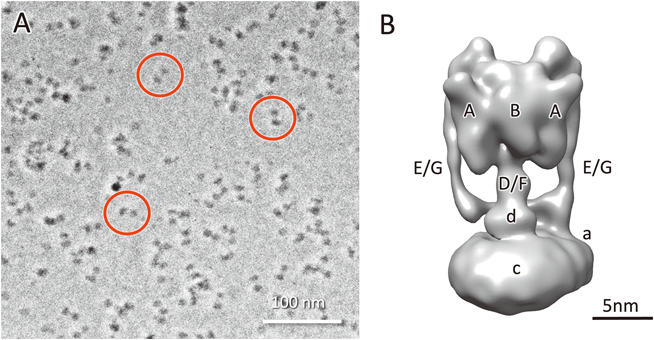
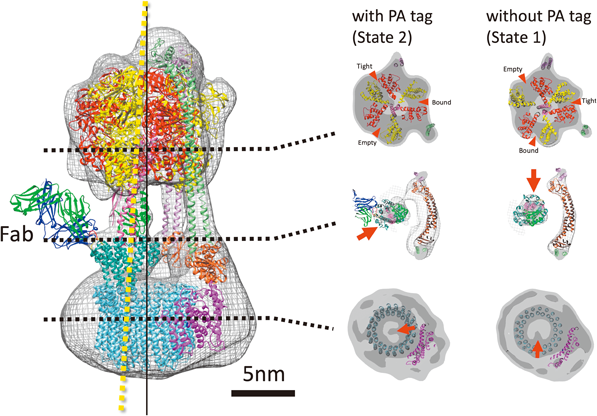
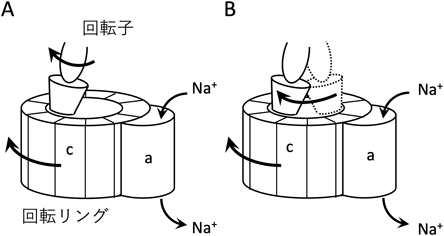
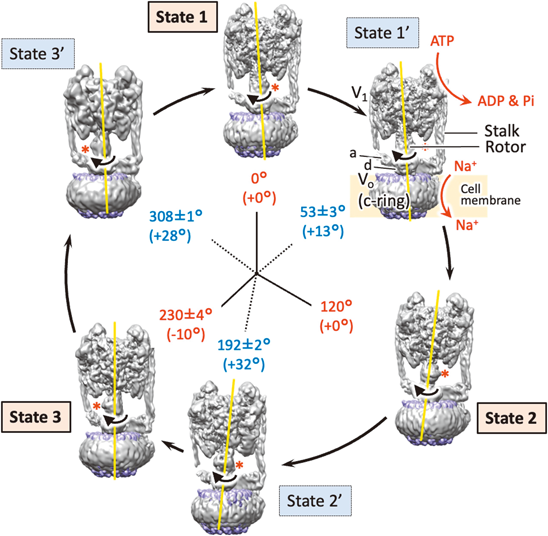
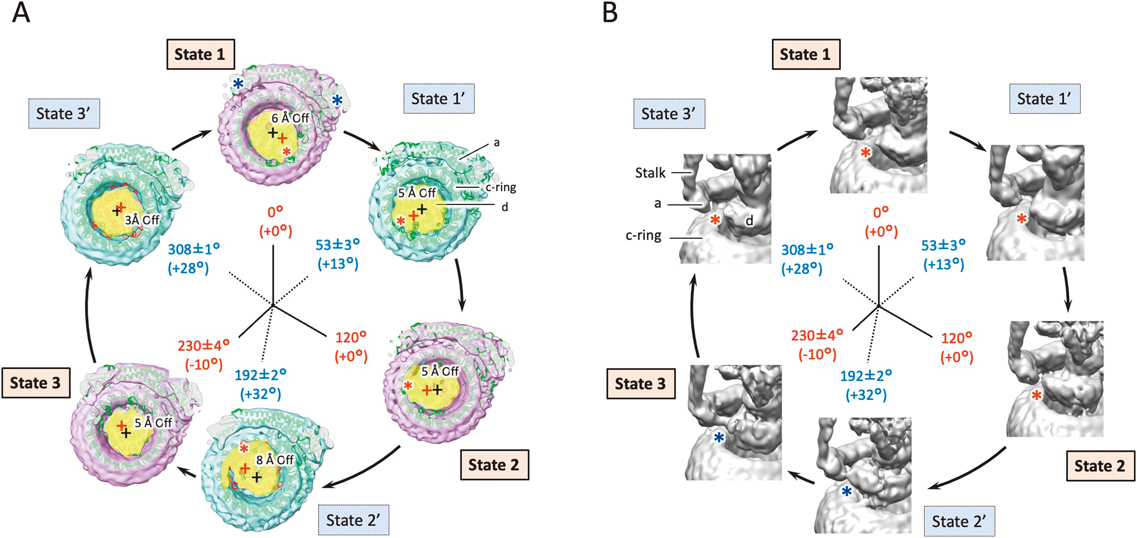
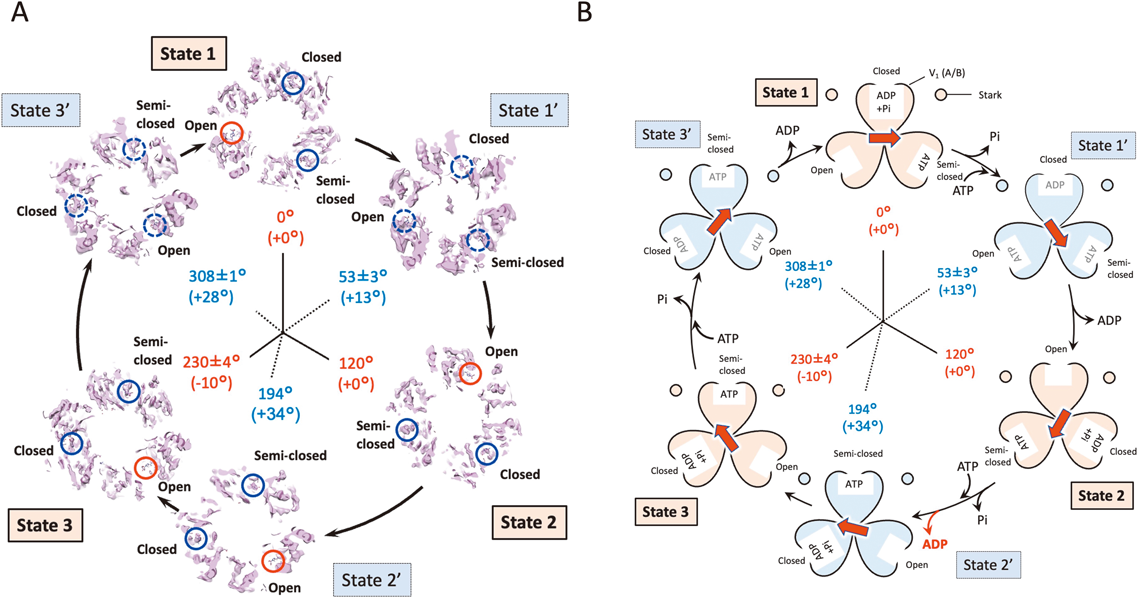
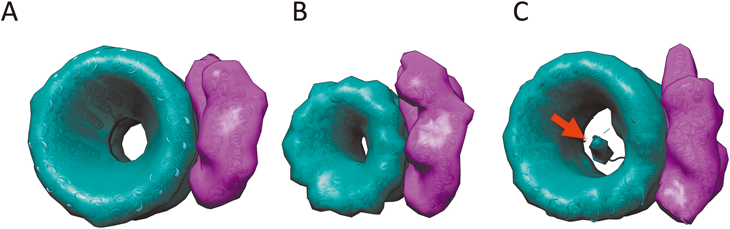腸球菌V型ATPアーゼ反応過程の構造解析Structural analysis of the reaction process of Enterococcus hirae V-type ATPase
自然科学研究機構生命創成探究センターExploratory Research Center on Life and Living Systems (ExCELLS), National Institutes of Natural Sciences (NINS) ◇ 〒444–8585 愛知県岡崎市明大寺町字西郷中38 ◇ 38 Nihigonaka, Myodaiji, Okazaki, Aichi 444–8585, Japan