2. 翻訳品質管理機構RQCによる衝突リボソームの認識と解消機構
1)RQCの概要
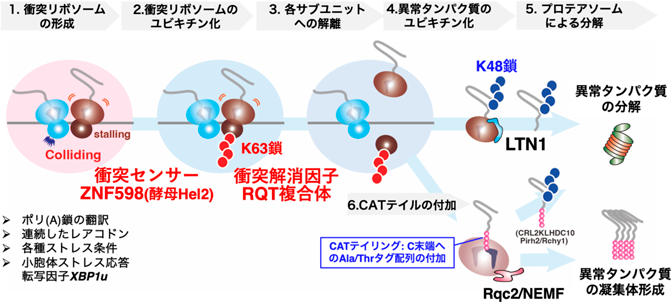
翻訳品質管理機構RQCは,異常な翻訳を監視し異常タンパク質を合成途中で分解除去する翻訳品質管理機構であり,タンパク質恒常性維持のごく初期段階としてきわめて重要な役割を果たす1–3).RQC機能不全マウスは進行性神経変性の表現型を示すことや,若年性神経筋疾患と自閉症患者にRQC関連因子の変異が報告されていることから,特に神経系におけるRQCの重要性が示唆されている.RQCにおいて,異常翻訳の認識から異常タンパク質分解までの各段階を担う因子は真核生物で広く保存されている.翻訳伸長中のリボソームがmRNA上で長期間停滞すると,後続のリボソームが追いつき,先に停滞していたリボソームに衝突する.衝突リボソームセンサー(酵母Hel2,ヒトZNF598)はE3ユビキチン化酵素であり,このリボソームどうしの衝突を異常な翻訳とみなし,先に停滞したリボソームのuS10リボソームタンパク質にK63型のポリユビキチン鎖を導入する.その後,RQT(RQC-Trigger)複合体がK63鎖を検出し,先に停滞したリボソームを40Sと60Sの各サブユニットへ解離する.解離後の60Sサブユニット上の異常タンパク質は,60Sサブユニットに特異的に結合するE3リガーゼであるLTN1によってユビキチン化され分解されるだけでなく,60Sサブユニットに結合してアミノアシルtRNAをリクルートするNEMFによるCATテイリング(C末端へのAlaもしくはThr残基の付加反応)を受ける(後述の3-1)の項を参照).
2)RQT複合体による衝突リボソーム認識と解消機構
以前我々は,衝突リボソームを生じるSDD1として知られる内在性の翻訳停滞配列を利用することで,RQCが起こる際に観察される各段階,1)翻訳停滞,2)三つのリボソームによるトライソーム形成,3)衝突リボソーム特異的ユビキチン化反応,4)ユビキチン化停滞リボソームのRQT複合体による各サブユニットへの解離反応,を試験管内翻訳反応で再構築した4)(図1).この系を利用し,内在性の翻訳停滞配列で形成される衝突リボソームであるトライソームの構造をクライオ電子顕微鏡による単粒子解析で明らかにし,レアコドン連続配列等で形成されることが知られていたダイソームと呼ばれる衝突リボソームの構造と比較した.衝突の結果生じる二つの80Sリボソームの40Sサブユニット間で形成される特殊な構造が,衝突リボソームセンサーHel2が認識する特異的構造であり,異常翻訳の実体であることが強く示唆された.
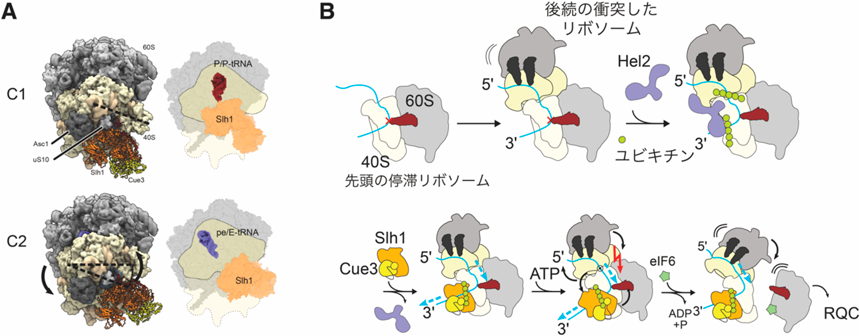
詳細は以下に示すが,我々は,衝突リボソームとそれに結合したRQT複合体をクライオ電子顕微鏡を用いて可視化し,RQT複合体がmRNAに引っ張り力を加え,リボソームの40Sサブユニットの構造を不安定化させ,最終的にサブユニットを解離させることを明らかにした5).まず,試験管内で翻訳停滞によるリボソームの衝突と衝突リボソームのユビキチン鎖の付加を再現し,衝突リボソームに結合したRQT複合体の構造を明らかにした.RQTは先頭の停滞したリボソームの40Sサブユニットに結合し,二つの構造(C1状態とC2状態)を切り替えていた(図2A).C1からC2への遷移は,転位反応における40Sの動きと逆になっており,この動きが,RQT依存の解離メカニズムの鍵となる可能性が考えられる.ATPase欠損変異Ski2様ヘリカーゼ1(Slh1)を含むRQT複合体や,3′側にはみ出したmRNAを欠く衝突リボソームでは,RQTによる衝突リボソームの解離反応が起きず,C2状態も存在しなかった.したがって,C2状態を獲得するには,mRNAに対するATP依存性ヘリカーゼ活性が発生する必要があり,この状態への移行は先頭の停止リボソームの解離反応が重要であると結論した.このように,RQTのSlh1サブユニットが停滞したリボソームを不安定化させ,最終的にサブユニットの解離を引き起こすことを提案するに至った(図2B).
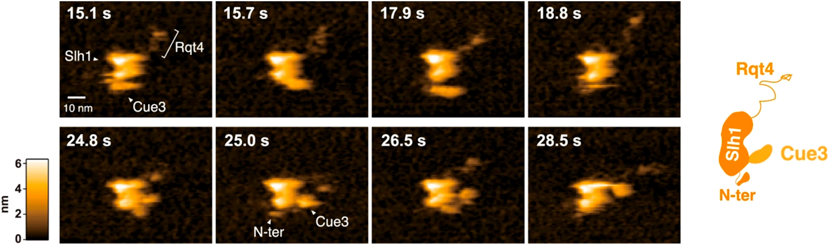
クライオ電子顕微鏡を用いる解析では,動的領域の可視化は困難である.我々は,1分子レベルの動態を可視化できる高速AFMを用いて,RQT複合体の動き,特に運動性の高い天然変性領域を可視化し,リボソームの異常な交通渋滞を選別する仕組みを解明した6).まず,RQT複合体の構成タンパク質であるCue3とRqt4が,衝突リボソームに形成されたK63型のユビキチン鎖を識別することを発見した.さらに,RQT複合体のアクセサリータンパク質であるRqt4は天然変性領域に富んだタンパク質であり,触手のように動くことで,ユビキチン鎖を捜索する様子が観察された(図3).Cue3とRqt4の二つのユビキチン識別サブユニットが協調的に相互作用して,効率よくユビキチン化衝突リボソームを認識すると考えられる.
3)衝突リボソームを認識し解消する分子機構の普遍性
哺乳類においては,出芽酵母Hel2のホモログであるZNF598によるuS10およびeS10のモノユビキチン化がRQCを誘導する報告がある.しかし,出芽酵母のRQCではK63型ポリユビキチン修飾を必要とすることや,RQT複合体のヒトホモログ(human-RQT複合体)がポリユビキチン結合活性を有することから,哺乳類細胞でも衝突リボソーム上にK63鎖が形成され,RQT複合体で認識されることが強く示唆されていた.我々は,ユビキチン修飾を介した翻訳停滞の解消を試験管内で再現し,哺乳類での衝突リボソームに対処する仕組みを明らかにした7).
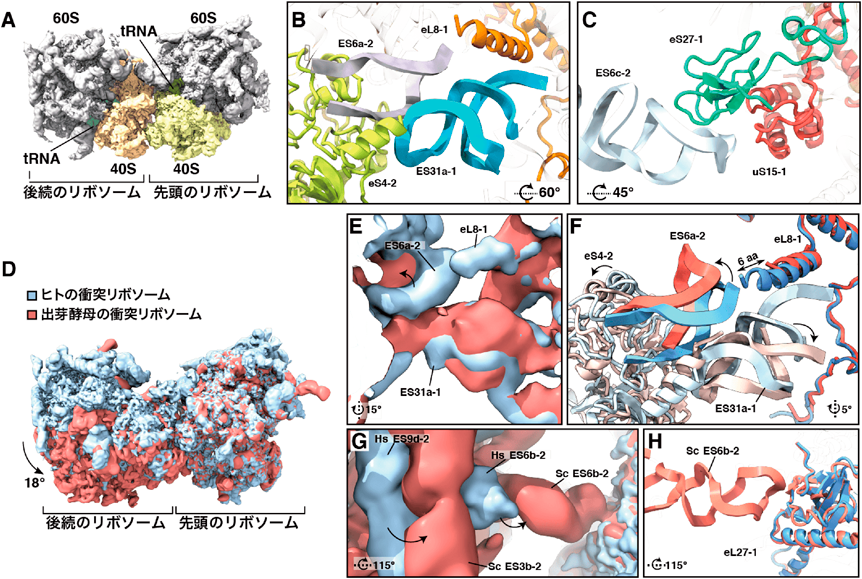
我々はまず,内在性の翻訳停滞配列であるXBP1u mRNA上で形成されたヒトの衝突リボソームの立体構造をクライオ電子顕微鏡により解析し,衝突リボソーム間の新規相互作用領域を2か所発見した(図4A).一つ目は60S–40Sの境界部分(図4B),二つ目は40S–40Sの境界部分(図4C)であった.興味深いことに,出芽酵母の衝突リボソームと比較すると,先頭のリボソームに対して衝突しているリボソームが18°回転しており(図4D),主に60S–40Sの境界領域の相互作用の違いが示唆された(図4E~H).RQC誘導に関与するユビキチン修飾は,出芽酵母ではuS10のみ,ヒトではeS10とuS10の両方で起こるという違いは,出芽酵母とヒトにおける衝突リボソームの構造の違いから説明できる可能性がある.
次に我々は,RQCの誘導過程である衝突リボソームの形成,リボソームのユビキチン化,ヒトRQT複合体によるサブユニット解離までの一連の反応を,内在性の翻訳停滞配列であるXBP1u mRNAを用いて試験管内で再現し,K63型ポリユビキチン鎖形成を初めて観察した.ヒトRQT複合体はモノユビキチンには結合せず,K63型ポリユビキチン鎖に特異的に結合し,K63型ポリユビキチン化されたuS10を持つリボソームを特異的にサブユニットへと解離した.一方,ヒトRQT複合体を介したサブユニット解離反応へのeS10のユビキチン修飾の寄与は確認されなかった.したがって,ヒトRQT複合体は,uS10に形成されたK63型ポリユビキチン修飾を目印として衝突リボソームのサブユニット解離を行うモデルが提唱された(図1).以上の結果は,衝突リボソーム上にK63鎖が形成され,RQT複合体で認識される仕組みが,出芽母から哺乳類まで広く保存されていること示している.
4)NGDにおける切断酵素Cue2の基質認識機構
衝突リボソームは,RQCだけでなく,mRNA分解機構であるNGDを誘導する.出芽酵母におけるNGDの活性化は,衝突リボソームセンサーHel2が触媒するリボソームタンパク質uS10およびeS7Aのユビキチン化によって惹起される.近年,衝突リボソーム上のmRNAを切断する酵素としてCue2が同定された.我々は,Cue2が,ユビキチン化リボソームを認識しmRNAの切断部位を決定する仕組みを最近明らかにした8).Cue2のN末端領域に存在するユビキチン結合ドメインは,eS7Aがユビキチン化された衝突リボソームを認識し,衝突リボソーム上流でのmRNA切断に必須であった.一方で,衝突リボソームがRQT複合体で解離する反応に共役して起こるmRNA切断には,122番目のトリプトファン残基が必須であった.これらのCue2によるmRNAの切断部位の決定機構は,対応するtRNAの量が少ないレアコドンの連続配列による衝突リボソームのみでなく,新生ペプチド鎖とリボソームトンネル内腔との立体障害による翻訳停滞で誘導されるNGDでも観察されており,一般的な異常翻訳に起因するmRNA切断機構と考えられる.
5)衝突リボソームとストレス応答
衝突リボソームはシグナル伝達と細胞死も誘起する.翻訳伸長阻害剤やアミノ酸飢餓,紫外線(UV)照射等のストレス条件時に,衝突リボソームRSR(Ribotoxic Stress Response)とGCN2を介した統合ストレス応答(Integrated Stress Response:ISR)の二つのストレス応答を活性化するモデルが提唱された9).MAPKKKであるZAKが衝突リボソームセンサーとして機能し,SAPK(p38/JNK)とGCN2シグナル伝達経路の両方を活性化すると考えられる.ZAKは,衝突リボソームの最小単位であるダイソーム上で,特異的に自己リン酸化を行い,GCN2による統合ストレス応答ISRを活性化すると考えられる10).リボソーム衝突はRQCとISRの両方を活性化し,かつ一方の経路の不活性化は他方の過剰活性化をもたらすモデルが提唱された.しかしながら,GCN2とHel2(ヒトZNF598)の認識する基質は異なっており,GCN2はアミノアシルtRNAの細胞内量の低下の結果,空になったリボソームのA部位を認識するモデルが示されてきた.一方で,Hel2(ヒトZNF598)は衝突リボソームの40Sどうしの相互作用部位を識別する.したがって,GCN2による統合ストレス応答と異常翻訳品質管理機構RQCの相互の関係を明らかにすることが,今後の重要な課題である.
3. RQCによる異常タンパク質の分解機構と生理機能
1)非典型翻訳反応CATテイリングにおける翻訳因子eIF5Aの新規機能と生理機能
RQT複合体による解離反応後に60Sサブユニットに存在する合成途上の異常タンパク質は,E3ユビキチン化酵素LTN1によって分解の目印となるK48型のポリユビキチン化を受ける12–15).ユビキチンプロテアソーム依存分解系の主要因子Cdc48とUfd3が,ユビキチン化異常タンパク質をプロテアソーム依存の分解に導く15, 16).出芽酵母において,LTN1によるユビキチン化が起きない場合に,60Sサブユニットの異常新生タンパク質のカルボキシ末端(C末端)に,アラニンとトレオニンからなるCATテイル(C-terminal alanine-threonine tail)と呼ばれるタグ配列がmRNA非依存的に付加される17).CATテイルの形成には酵母Rqc2が必須であり,試験管内で再構成されているが18),その形成効率を制御する機構はいまだ不明である.RQCを引き起こす配列として,対応するtRNAの量が少ないレアコドンや,連続した塩基性アミノ酸配列が同定されている.トリプトファンの連続配列もRQCを引き起こすが,Rqc2依存のCATテイルの形成を阻害する19).CATテイルの形成効率の制御機構の理解には,60Sサブユニット中での新生ポリペプチド鎖とリボソームトンネルとの相互作用の詳細や,Rqc2によるアミノアシルtRNAの識別機構を明らかにする必要がある.
通常の翻訳伸長反応とは異なり,mRNAやエネルギー源となるGTPaseが存在しないにもかかわらず,Rqc2は合成途中のペプチド鎖のC末端側にCATテイルと呼ばれるアラニンとトレオニンのみからなるランダムなペプチド配列を付加する.Rqc2はCATテイルを新生ペプチド鎖のC末端側に付加することでLTN1がユビキチン化を行いやすくし,新生ペプチド鎖の分解効率を向上させる働きを担う.一方で,CATテイルは,異常タンパク質の分解促進とは相反する,凝集体の蓄積を誘導することも報告されている20).LTN1欠損マウスでは進行性の神経変性と運動機能障害が観察された21).しかしながら,神経変性が発症する原因がCATテイルによる凝集体の蓄積であるかは不明であった.最近我々は,RQCの機能不全による細胞障害の原因に,CATテイル化タンパク質の蓄積が関わることを明らかにした.LTN1依存の異常タンパク質分解が機能不全に陥ると,CATテイル化されたタンパク質が凝集体を形成し,神経細胞の形態形成の阻害やプログラム細胞死を引き起こす22).さらに最近,E3ユビキチン化酵素複合体CRL2KLHDC10と,C末端ルール依存の分解に関与する新規のE3ユビキチン化酵素であるPIRH2/RCHY1が,C末端のAlaテイル(Ala残基のみを含むCATテイル)を持つRQC産物に直接結合して分解することが報告され,新規のRQC経路として注目されている23).
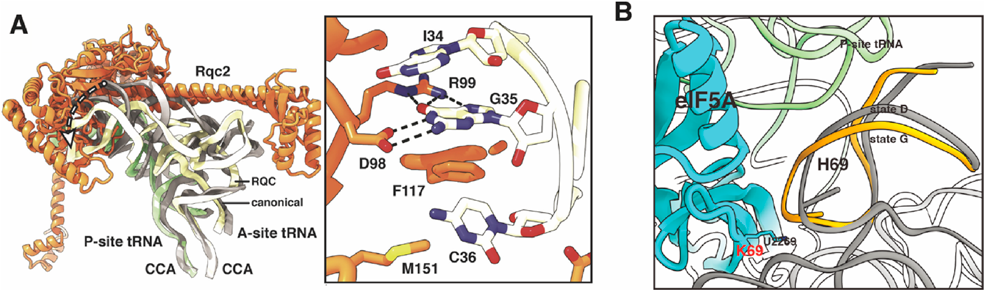
CATテイリング反応は従来の翻訳形態とは異なるペプチド合成反応である.通常の翻訳伸長反応では,リボソームの構造変化が常時行われており,翻訳伸長因子EF-GによるGTP加水分解が転位反応に必要である.Rqc2は解離したリボソームの大サブユニットに結合し,mRNAや翻訳伸長因子非依存に,合成途中のタンパク質のC末端側にCATテイルを付加する.最近我々は,クライオ電子顕微鏡を用いた構造解析と遺伝学的手法による機能解析によってeIF5AがCATテイリング反応の促進因子としての役割を担うことを発見し,eIF5Aの結合がCATテイリング反応に必須なリボソームの構造変化の引き金として働くことを見いだした24)(図5).LTN1欠損下でのCATテイル形成は,神経突起伸長を阻害し,神経細胞死を誘導する22).またRqc2のヒトホモログであるNEMF遺伝子の変異は神経筋疾患との関連が示唆されている25).疾患発症の理解の創薬基盤の構築には,NEMFによるCATテイリング反応の分子機構の解明が必須である.
2)小胞体膜上でのRQC基質の分解機構
UFM化は,ユビキチン化と類似のタンパク質のリシン残基への修飾である.UFM化はUFM1タンパク質の活性化酵素と結合酵素,そして連結酵素を介して細胞内タンパク質にUFM1との共有結合を形成するシステムであり,UFM1連結酵素が基質特異性を決定する.最近我々は,UFM1連結酵素であるUFL1と小胞体に局在するUFBP1とCDK5RAP3がUFM1連結酵素複合体を形成し,小胞体上で翻訳が停止したリボソームのRPL26にUFM1修飾を導入することを明らかにした26).さらに,このUFM1連結酵素複合体は,UFBP1に含まれるUFM1結合モチーフを介してUFM化修飾を受けたRPL26に結合することが,小胞体における合成途中のタンパク質分解に必要であることを明らかにした.小胞体膜上でのRQC基質の分解機構におけるUFM化の機能はほとんど不明であるが,今後多くの新規制御機構が明らかになることが期待される.
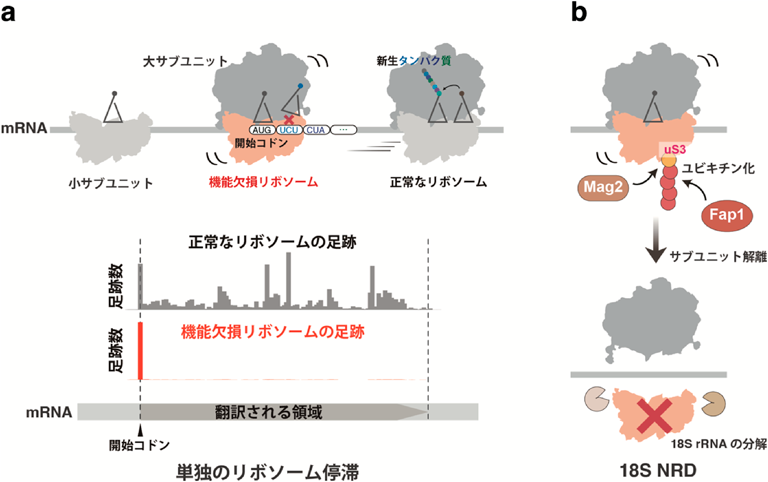
4. 機能欠損リボソームを分解する品質管理機構NRDとリボソーム量制御
リボソームは,正確なコドン認識と高効率なペプチド結合を担うタンパク質合成の中心装置であるだけでなく,さまざまな因子と相互作用し遺伝子発現に必須な機能を果たす.一方で,リボソーム合成は細胞のエネルギーの多くを消費し数百の因子が関与する細胞内の主要な反応である.その合成途上で生じた異常リボソームはさまざまな発現異常の原因となるため,細胞は機能欠損リボソームを認識し排除する品質管理機構NRDを保持している.最近我々は,すべての生物種で保存されている18S rRNA内の塩基の置換変異により正確なコドン識別能を欠損したリボソームの品質管理を解析し,リボソームの活性中心への変異導入が,出芽酵母における単独のリボソーム停滞の効率的な誘導と解析に有用であることを発見した27, 28)(図6a).また,リボソームプロファイリング法により,遺伝暗号解読活性を失ったリボソームがmRNA上の開始コドンで単独で停滞することを明らかにした.18S NRDにおけるuS3のユビキチン化はMag2とFap1という二つの因子が順に触媒する二段階の反応であることも見いだした28)(図6b).
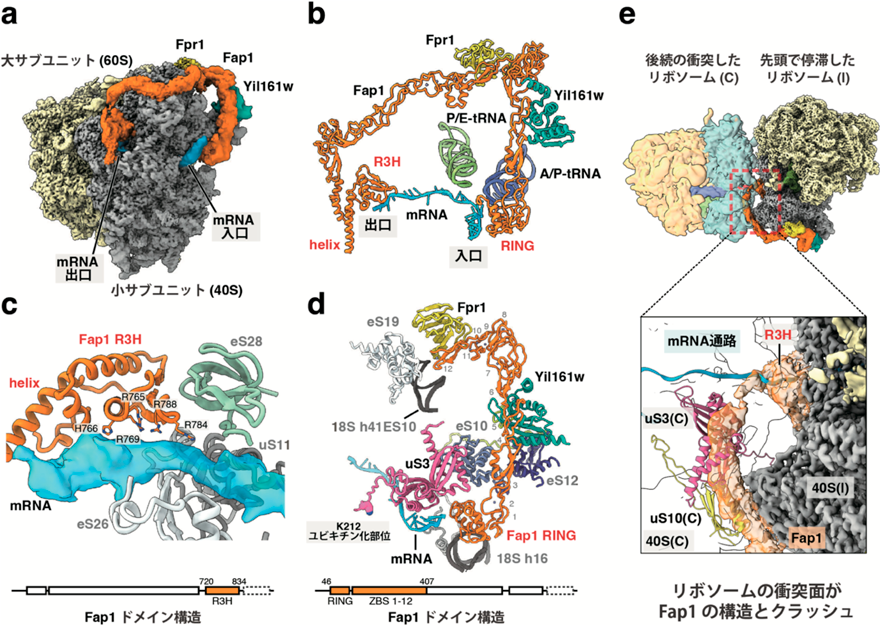
これらの因子が認識するリボソームの特徴をリボソームプロファイリング法で解析した結果,Mag2が翻訳速度の遅いリボソームに全般的に結合する可能性が示唆された.またFap1は隣接リボソームの出現頻度の低い位置に存在するリボソームを好んで結合することが示された.FAP1がリボソームに結合している立体構造をクライオ電子顕微鏡で明らかにし,Fap1は複数の亜鉛結合部位からなる細長い構造を介してリボソーム小サブユニットの頭部にアーチをかけ,両端のRINGドメインとR3Hドメインがリボソーム上のmRNAの入口,出口それぞれと相互作用する構造を明らかにした(図7a–d).翻訳伸長中のリボソームの構造変化によるmRNAの相対的な動きがこれらの相互作用を破壊し,リボソームが静止状態になったときのみFAP複合体が活性化する可能性が示唆された(図7e).これらの結果から,Fap1はリボソーム両側のmRNAの動きの停止を感知することで,衝突を伴わない停滞をリボソームの機能異常の信号として提示し,サブユニット解離に伴う停滞の解消およびリボソーム自身の分解を誘導することが明らかになった(図7).