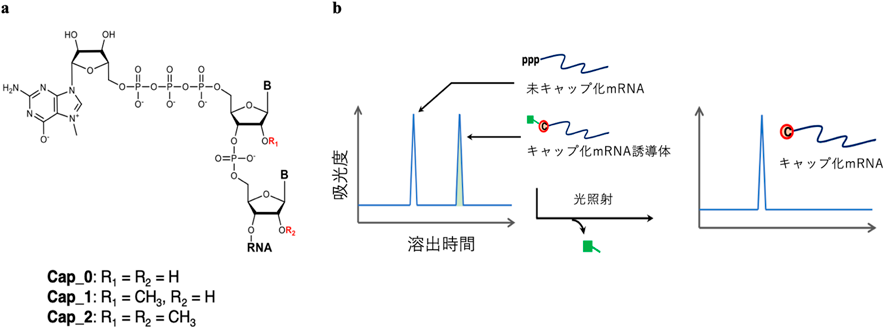
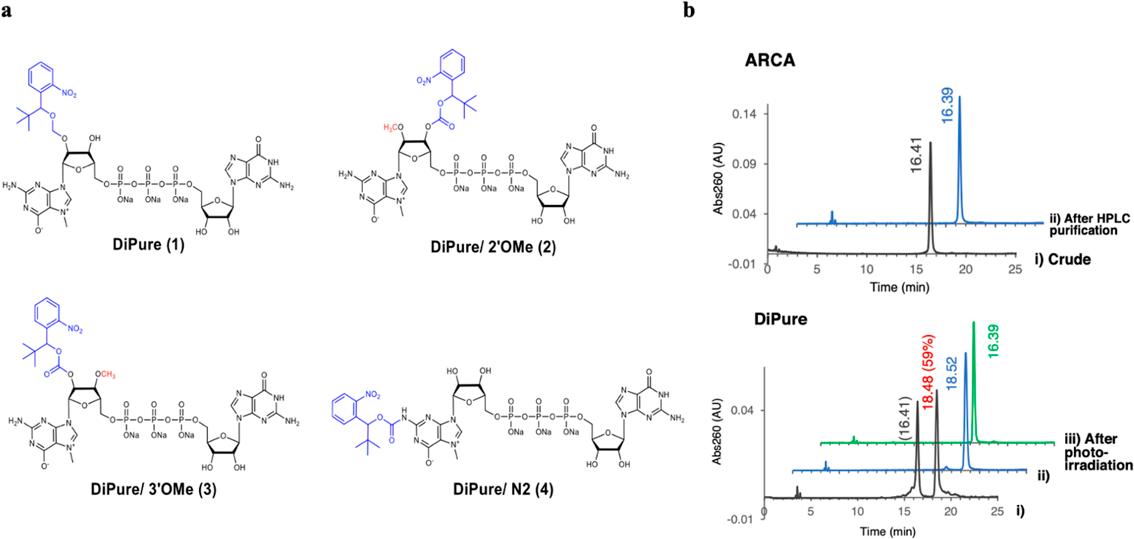
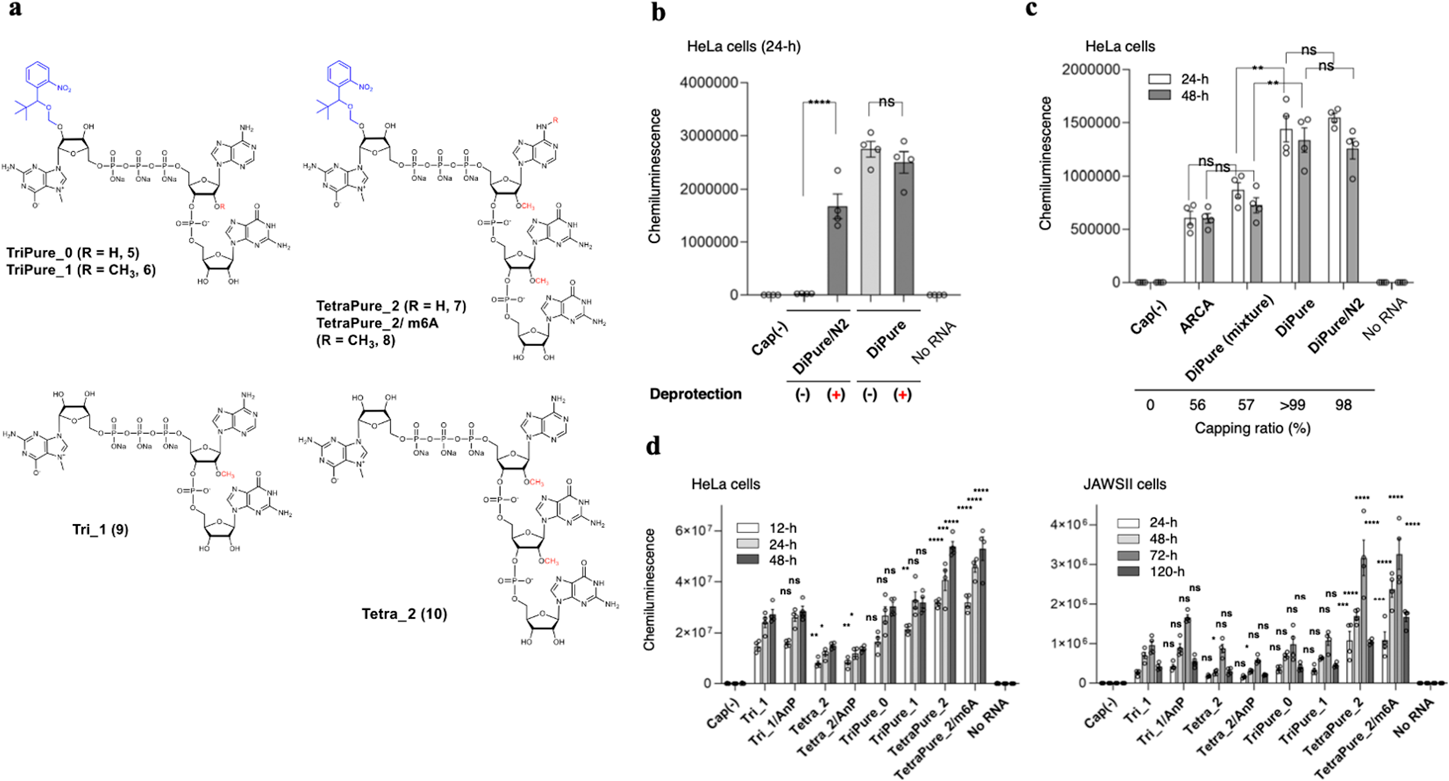
高純度キャップ化mRNAの調製を可能とするPureCap法の開発Development of PureCap method for preparation of capped mRNA in high purity
名古屋大学大学院理学研究科理学専攻生物有機化学研究室Bio-Organic Chemistry Lab, Graduate School of Science, Nagoya University ◇ 〒464–8602 愛知県名古屋市千種区不老町 ◇ Furo-cho, Chikusa-ku, Nagoya, Aichi 464–8602, Japan