従来の生命科学では,機能性,酵素活性,薬効などを持つ単一の遺伝子,タンパク質,代謝産物が研究のターゲットとされていた.しかし,1990年以降に,モデル生物のゲノム配列が続々と決定されるとゲノムのみならず,大規模に遺伝子,タンパク質,代謝産物などの生体分子を測定し,得られた大量のデータから生命事象を俯瞰して理解しようとする研究が主流となってきた.
代謝産物を網羅的に測定するメタボローム解析は,代謝産物の機能や代謝経路,代謝調節機構,未知遺伝子やタンパク質の機能,生体分子と代謝産物の相互作用,低分子マーカーなどを解き明かそうとする方法論であり,この技術の発展に伴って,代謝産物や代謝に関する新たな知見が次々と報告されるようになった.
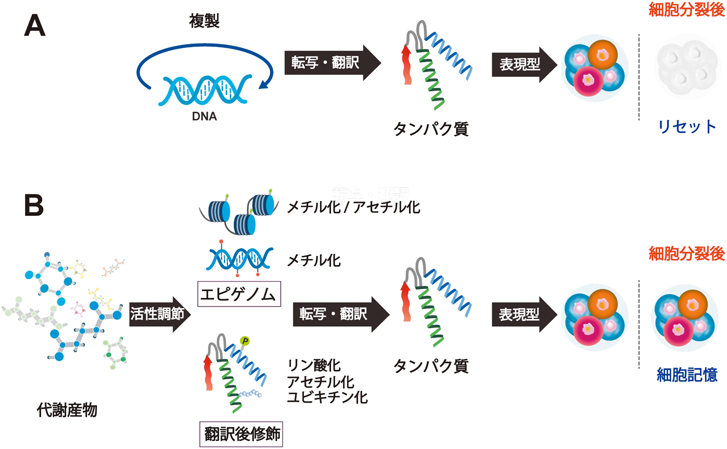
タンパク質とともにゲノム情報の最下流に位置する代謝産物は,化学反応に利用されたのち不要であれば捨てられる運命にあるとみなされていた.ところがオミクス解析の発展に伴い,代謝産物がエピゲノム反応をつかさどる酵素活性やタンパク質翻訳後修飾を介して遺伝子の転写・翻訳を調節していることが見いだされた.一般に遺伝子発現は一過的で,遺伝子情報の表現型は細胞分裂によってリセットされる(図1A).しかし,DNAやヒストンのエピゲノム修飾は,細胞分裂が起きても保存されるため1, 2),表現型は世代を超えて細胞に記憶され続ける(図1B).
このことは,エピゲノム修飾を誘導する代謝産物は,細胞の分化,がん化,アレルギー,免疫応答などのあらゆる生理機能・生命現象を制御することを示唆する.ゲノム情報が変化しなくとも,生体は代謝産物からの分子情報を介して栄養,微少環境,腸内細菌叢,代謝異常などの内外環境の変化に対する表現型の多様性を生み出し,それによって順応性・可塑性を獲得し恒常性を維持することができる.生活習慣病やがんの発症などは,食事,腸内細菌,肥満,運動,喫煙,炎症,ストレスなどの環境の影響を受けるといわれているが,代謝産物が原因で引き起こされている可能性があり,今後の研究の進展により多くのブレークスルーが生み出されるのではないかと期待されている.
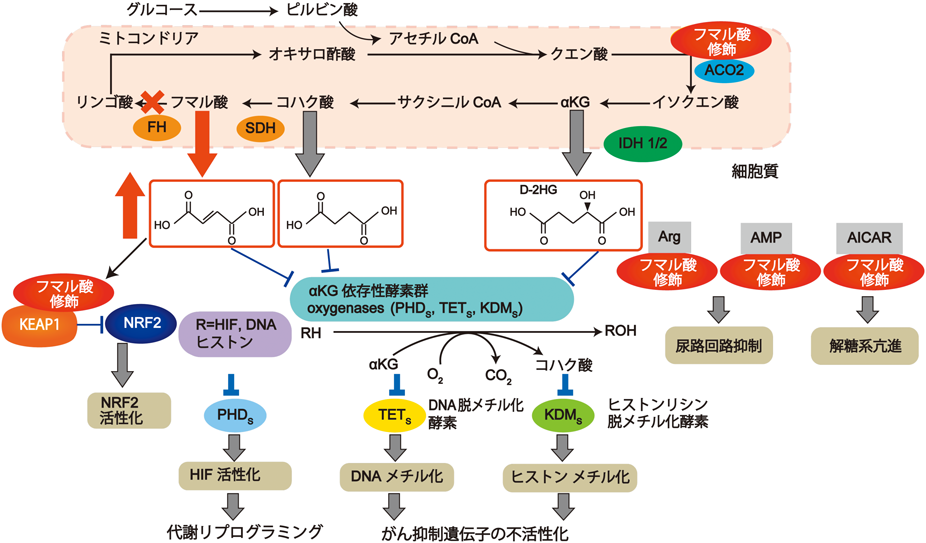
それに応えるように近年,がん細胞や免疫細胞を制御している代謝産物が発見された.発がんやがんの進展に関与する代謝産物(オンコメタボライト)としてD-2-ヒドロキシグルタル酸(D-2-hydroxyglutaric acid:D-2HG),フマル酸,コハク酸などが報告されている3, 4).D-2HGはイソクエン酸脱水素酵素(isocitrate dehydrogenase:IDH)5–7),フマル酸はフマル酸ヒドラターゼ(fumarate hydrase:FH)8, 9),コハク酸はコハク酸脱水酵素(succinate dehydrogenase:SDH)10, 11)の変異により細胞内に蓄積する(図2).これらの代謝産物はα-ケトグルタル酸(α-ketoglutaric acid:αKG)と構造が類似するため,競合的にα-ケトグルタル酸依存性ジオキシゲナーゼ酵素群の活性を阻害する4, 12).この酵素群には,プロリン水酸化酵素(prolyl hydroxylase:PHD),DNA脱メチル化酵素(ten-eleven-translocation:TET)やヒストンリシン脱メチル化酵素(histone lysine demethylase:KDM)などが存在する.PHDの活性が抑制されると低酸素誘導因子(hypoxia inducible factor 1α:HIF-1α)のユビキチン・プロテアソーム系による分解が抑制されて安定化し,血管新生,細胞増殖,アポトーシス,糖代謝に関与する遺伝子を発現する.また,TETやKDMの活性が抑制されるとDNAおよびヒストンのメチル化が促進し,その結果,クロマチン構造が変化し,その領域にある遺伝子の発現が変化することによってがん化に寄与する3).
IDH1/2の変異によって急性骨髄性白血病13),グリオーマ5),FHの変異によって腎細胞がん14),SDHの変異によって傍神経節腫15)や褐色細胞腫10)が発症することが報告されている.
一方,がん細胞が産生する代謝産物(乳酸,キヌレニン,アデノシン,スペルミジンなど)が,T細胞の分化や活性化を阻害することでがん細胞が免疫細胞の攻撃から逃れていることも明らかにされている16–18).また,肥満になると二次胆汁酸を産生する腸内細菌が増加し,これにより細胞老化や肝細胞がんが促進されるということも報告されている19).さらに,細菌感染などでM1マクロファージの解糖系が亢進し産生された乳酸が外界に放出され,ヒトやマウスの細胞のヒストン(H3K23)を乳酸修飾し,ホメオスタシス関連遺伝子群を発現する20),など,がん細胞や免疫細胞のみならず代謝産物が生命事象を制御する例は枚挙にいとまがなく,近年,代謝産物の重要性が注目を浴びている.
現在,代謝産物の機能や代謝機構の解明にメタボローム解析は不可欠な技術となっているが,本稿では,メタボロミクスの黎明期から筆者らが開発してきたメタボローム測定技術とその最新技術,さらにがん研究を中心とした生命科学への応用例を紹介したい.
4種類のヌクレオチドからなるゲノム解析やトランスクリプトーム解析,20種類のアミノ酸からなるプロテオーム解析とは異なり,物理的化学的性質が異なった数千種類の代謝産物を一斉に測定しようとするメタボローム解析の技術開発は難しく,いまだにすべてのメタボロームを測定できる方法論は確立されてはいない.
2000年代に入ると核磁気共鳴分析(nuclear magnetic resonance:NMR)21),ガスクロマトグラフィー質量分析計(gas chromatography mass spectrometry:GC/MS)22, 23),液体クロマトグラフィー質量分析計(liquid chromatography mass spectrometry:LC-MS)24)を用いたメタボローム測定技術が誕生した.
NMRは試料を直接測定できるという利点はあるものの,感度と分離能が乏しく,測定可能な代謝産物はせいぜい数十種類である.
GC/MS法は高感度,高分離能を有する汎用性の高い気体成分の測定技術である.しかし,代謝産物の多くは不揮発性であり,これらの物質を気化させるための誘導体化反応が必要になる.また,誘導体化を行っても気化しない代謝産物も多く,限られた代謝産物しか測定することができない.
これに対してLC-MS法は,幅広い範囲の代謝産物を測定できる分析手法である24).LC-MSで一般に用いられる逆相系の分離カラムは,脂質のような疎水性の高い代謝産物の測定には適している一方,中心炭素代謝経路に多く存在する親水性の代謝産物の分離を苦手としている.近年,親水性の代謝産物の分離に適した親水性相互作用カラムも開発され,LC-MS法でカバーできる代謝産物の種類は増えてきた25).ただ,代謝産物の種類に応じて分離カラムや測定条件を選ばなければならないことや,質量分析計で問題となるイオンサプレッションの影響により定量性が乏しいことなど,LC-MS法もメタボローム解析においてはいくつかの課題が残っている26, 27).
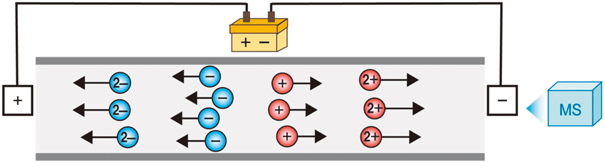
筆者らは,解糖系,クエン酸回路,アミノ酸生合成,核酸生合成などの経路に存在する代謝産物のほとんどがリン酸基,カルボキシ基,アミノ基などのイオン性官能基を有していることに着目し,イオン性化合物に対して高分離能を有するキャピラリー電気泳動(capillary electrophoresis:CE)に質量分析計を接続させたCE-MS法によるメタボローム測定技術を独自に開発した(図3)28–30).
この方法では,試料を細長い中空のキャピラリーに注入後,電圧を印加すると,各代謝産物は,その物質が持つ電荷/水和イオン半径の比に基づいた速度で電気泳動し,キャピラリー出口に接続された質量分析計によって検出される.陽イオン性代謝産物はすべて陰極方向に,反対に陰イオン性代謝産物はすべて陽極方向に電気泳動するため,2種類の測定条件で数千種類のイオン性代謝産物の一斉分析が可能である.
CE-MS法は,中性や脂溶性代謝産物は測定できないといった欠点はあるものの,分離能がきわめて高い(理論段数数十万段)ため,イオン性代謝産物であれば構造異性体も含めて分離できることが大きな特長である.これまで筆者らは,主に共同研究を通じて,本法を微生物28, 31),植物32),動物29, 33)からヒト34–42)に至るさまざまな種類の検体のメタボローム解析に応用し成果を報告している.
メタボローム解析技術の誕生により,新規の低分子バイオマーカーが数多く発見されている.しかし,バイオマーカーを臨床検査会社が実用化するためには,1検体1分以内の測定時間が求められる.既存のメタボローム解析技術では,1検体の測定に数十分を必要とするため,筆者らはハイスループットメタボローム解析を可能にする多検体同時測定CE-MS技術を新たに開発した43).
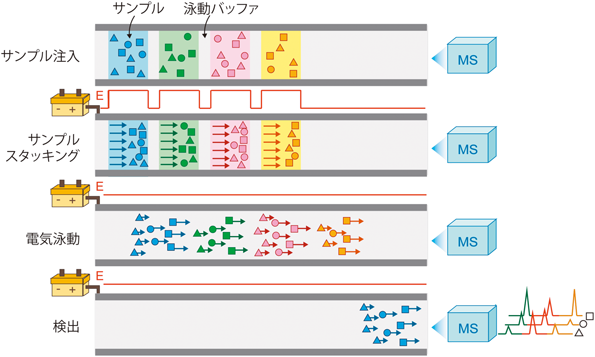
GC/MS, LC-MS, CE-MSなどの分離分析装置と質量分析計を組み合わせた測定技術は,分離を行うカラムやキャピラリーに一つのサンプルを注入後,各成分を測定する.しかし,多検体同時測定CE-MS技術は,キャピラリーに数十検体を注入後一斉に電気泳動を行って各検体の成分を一挙に質量分析計で検出する.
図4に多検体同時分析CE-MS法の測定原理を示した.まず,測定サンプルと泳動バッファを交互にキャピラリーに注入する(図4上段).その後,電圧を印加するが,サンプルの導電率が泳動バッファの導電率より低ければオームの法則に従い,印加される実際の電圧(E)は赤線で示したように,サンプルゾーンでは高く,泳動バッファゾーンでは低くなる(図4二段目).したがって,各成分は,サンプルゾーンでは速く移動し,泳動バッファゾーンに入ると減速する.その結果,各検体の成分はサンプルと泳動バッファの境界にいったん濃縮される(図4二段目).その後,サンプルと泳動バッファが混合し,両方の導電率が同じになると電位が一定になるため,電気泳動が開始され(図4三段目),1番目の検体の成分から順次質量分析計(MS)で検出される(図4下段).本法により,40検体の代謝産物を40分以内で測定することが可能になった43).
一方,ほとんどフラグメントイオンを生成しない飛行時間型質量分析計(TOFMS)を用いたCE-TOFMSによる多検体同時測定法では,各検体の代謝産物のピークが数珠つなぎに検出される.したがって,サンプル中に目的代謝産物の異性体が存在し,その泳動時間が異なると,そのピークが他の検体の代謝産物のピークと重なってしまい正確な定量値が得られなくなってしまう.この問題を解決するため,筆者らは,CEに三連四重極質量分析計を接続してフラグメント(断片)イオンを生成させ,目的の代謝産物しか検出しないフラグメントイオンをモニターした43).多検体同時測定CE-MS法は,ターゲットメタボローム解析の高速化を可能にしたきわめて有用な手法であると思われる.
4. CE-MSメタボローム測定法の生命科学への応用
筆者のラボではCE-MSをはじめとするメタボローム解析装置が60台以上稼働しており,広範囲にわたる研究を行っている.ここでは,がん研究に関するいくつかの例を紹介する.
1)オンコメタボライト:フマル酸によるがんの新たな代謝制御
遺伝性平滑筋腫症-腎細胞がん症候群(hereditary leiomyomatosis and renal cell carcinoma:HLRCC)の原因は,フマル酸ヒドラターゼ(fumarate hydratase:FH)の変異が原因で発症することが知られている.そこで,オックスフォード大学との共同研究によってフマル酸ヒドラターゼ(Fh1)を欠失させたノックアウト(KO)マウスを作製し,フマル酸が発がんに関与する機序を見いだした14).
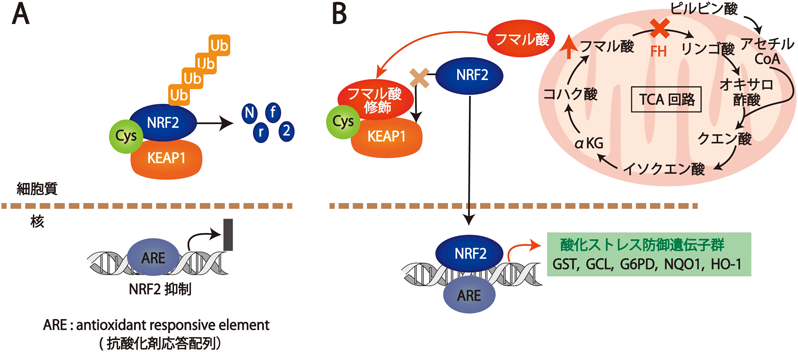
通常,酸化ストレス応答転写因子であるNRF2(nuclear factor erythroid 2-related factor 2)は,KEAP1(Kelch-like ECH-associated protein 1)タンパク質に結合することによってユビキチン・プロテアソーム系で分解される(図5A).しかし,Fh1 KOマウスでは,蓄積したフマル酸がKeap1タンパク質のシステイン残基にコハク酸修飾(フマル酸の二重結合が開いてコハク酸状態で共有結合すること)するためNrf2がKeap1に結合できなくなる.その結果,Nrf2は分解されずに安定化し,核に移行して酸化ストレス防御遺伝子群の転写を誘導し,酸化ストレス耐性を獲得する(図5B)14).
また,図2に示したように,蓄積したフマル酸が代謝産物であるアルギニン(arginine:Arg)やAMP(adenosine 5′-monophosphate)にもコハク酸修飾し,アルギノコハク酸,アデニロコハク酸を過剰に生成することによって尿素回路を抑制したり44),フマル酸がプリン生合成経路のAICAR(5-aminoimidazole-4-carboxamide ribonucleoside)にコハク酸修飾してSAICAR(5-amino-4-imidazole-N-succinocarboxamide ribotide)を生成し,その結果,解糖系を亢進したり45),ミトコンドリアの代謝酵素であるアコニターゼ2(aconitase 2:ACO2)にコハク酸修飾することによってACO2の活性を阻害してTCA回路を抑制したりすることも発見した46).
このように,HLRCCでは,FHの変異で蓄積したフマル酸がKEAP1に結合して酸化ストレス応答転写因子NRF2の安定化とその下流の酸化ストレス防御遺伝子群の発現,さらには,解糖系,TCA回路,尿素回路,プリン生合成経路などの代謝をリプログラミングすることによってがん化に深く関与していることが推測された4).
2)大腸がんの代謝解析
哺乳動物は酸化的リン酸化反応を用いてATP(adenosine 5′-triphosphate)を産生しているが,がん組織は解糖系を亢進させてATPを産生していること(ワールブルグ効果)が古くから知られている47).近年,解糖系のみならず,がん特異的な代謝経路48, 49)が見つかっているが,どのような機序でがんが代謝をリプログラミングするか不明であった.筆者らは,275例の大腸がん患者からの手術検体の生体内分子を網羅的に解析し,がんが代謝を切り換える分子機序の解明に取り組んだ50).
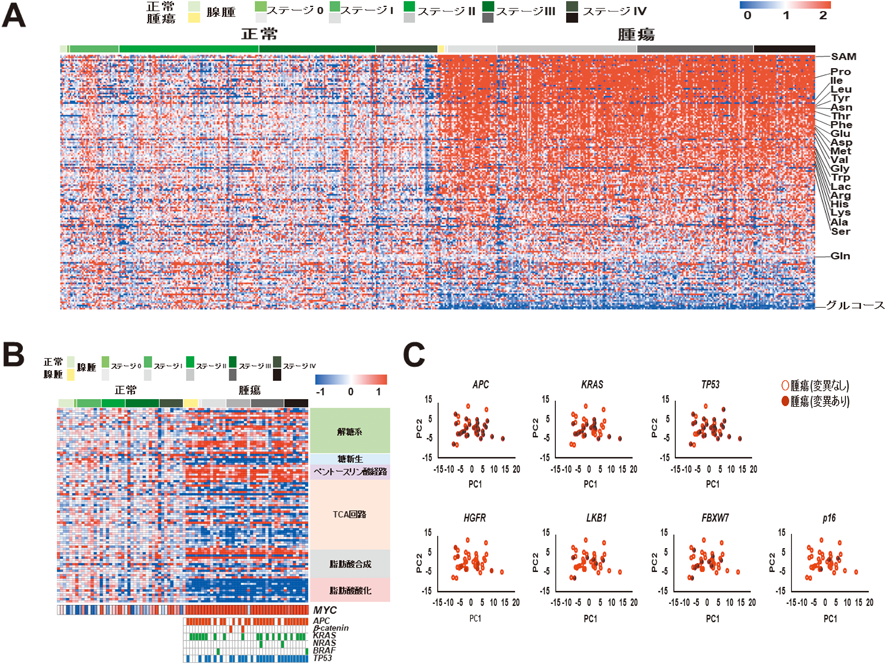
大腸がん患者の正常/腫瘍組織のペアを用いて,CE-MS法によるメタボローム解析,DNAマイクロアレイ解析を実施した.その結果,代謝産物の濃度(図6A)や代謝酵素遺伝子の発現(図6B)は,良性腫瘍ですでに変化しており,がんのステージによらず一定であった50).次に,腫瘍組織のがん遺伝子,がん抑制遺伝子のホットスポットの変異解析を行い,メタボローム解析データを用いて主成分分析を行った(図6C).その結果,APC, KRAS, TP53, HGFR, LKB1, FBXW7, p16など腫瘍組織で見つかった遺伝子の変異では,代謝産物の濃度は変化しないことが判明した50).
腫瘍組織を透過型電子顕微鏡で撮影すると,すべての腫瘍組織で,ミトコンドリアの膨潤化,クリステ,マトリックスの消失などのミトコンドリア異常が観察された.腫瘍組織では,ミトコンドリアの生合成に関与する転写活性化因子PGC-1α(peroxisome proliferator activated receptor gamma coactivator 1),ミトコンドリアのメンテナンスに重要な働きを示すPINK1(PTEN-induced kinase 1),オートファジーのマスターレギュレーターの転写因子TFEB(transcription factor EB)の遺伝子発現が有意に低下していた50).この結果から推察すると,腫瘍組織では,新規のミトコンドリアは生合成されず,またオートファジーも抑制されているため,異常なミトコンドリアは除去されずに蓄積しているのであろう.
次に,正常および腫瘍組織でPGC-1αとPINK1に相関して発現が変動する代謝関連遺伝子を探索したところ,がん遺伝子MYCとヒストンメチル化酵素であるEZH2が非常に高い相関を示した50).EZH2の発現はMYCによって制御されている51)ことから,MYCに着目したところ,大腸正常組織に対してがん組織では,MYC遺伝子が7倍以上発現していた.
MYCは種々のがんにおいて転写が亢進している転写因子であり,全遺伝子の15%のプロモーターに結合し52, 53),多くのターゲット遺伝子の発現を制御することが知られている54, 55).そこで大腸の正常および腫瘍組織の遺伝子発現結果を解析したところ,MYCの発現量は,約500種類の代謝制御遺伝子の発現量ときわめて高い正または負の相関を示していることを見いだした50).
これらの代謝制御遺伝子の発現がMYCによって制御されているか確認するため,大腸がん細胞株HCT116を用いてMYCをノックダウンしたところ50),腫瘍組織で増加している遺伝子の発現は減少し,反対に減少している遺伝子の発現は増加し,腫瘍組織でみられたほとんどの代謝制御遺伝子の発現が抑制された.このことは,これらの代謝制御遺伝子の発現はMYCによって制御されていることを示した.この実験結果より,MYCが39種類のトランスポーター,ほとんどのプリンおよびピリミジン生合成経路の代謝酵素,多くのone-carbon metabolism,脂肪酸代謝,解糖系,糖新生など121種類の代謝酵素の遺伝子の発現を介して,少なくとも215の代謝反応を制御していることが判明した.
続いて,MYCによって代謝産物の濃度も制御されているか検討した.HCT116細胞を用いて,MYCをノックダウンすると,グルコースは増加し,乳酸は減少した50).つまり,腫瘍組織でみられたワールブルグ効果(解糖系の亢進)が抑制された.さらに腫瘍組織で増加していたSAM(S-adenosylmethionine)や多くのアミノ酸が,MYCのノックダウンにより減少し,腫瘍組織でみられた多くの代謝産物の濃度変化がリセットされることが判明した50).これらの結果は,MYCが代謝関連遺伝子の発現のみならず代謝産物の濃度も制御していることを示す.
最後に,MYCによって制御されている代謝酵素群が大腸がんの治療標的となるか検証した50).大腸がん細胞HCT116を用いてMYCをノックダウンすると細胞増殖は抑制された.続いて,MYCによってほとんどの遺伝子の発現が制御されているピリミジンおよびプリン生合成経路の代謝酵素遺伝子をノックダウンした.予想どおり,ピリミジン生合成経路の代謝酵素をノックダウンすると細胞増殖は抑制された.しかし,プリン生合成路の代謝酵素をノックダウンしても細胞増殖の変化はみられなかった50).この理由は不明であるが,プリンにはサルベージ経路も存在するため,生合成経路を阻害してもサルベージ経路によって代謝産物が補充されるため細胞増殖には影響がないのかもしれない.
今回の研究で,大腸がんでは,糖新生とβ酸化以外の代謝経路はすべて亢進しており,がん細胞の増殖や進展に必要なDNA, RNA,タンパク質,脂質,リン脂質の前駆体を活発に産生していることやMYCが大腸がんの代謝リプログラミングに中心的な役割を担っていることが判明した.またMYCによって制御されているピリミジン生合成経路が大腸がんの有望な治療標的候補になることも示された50).
3)多検体同時分析CE-MSによる唾液中のがんマーカー測定
古くから,がん患者の尿でポリアミン生合成経路のスペルミン,スペルミジンおよびそれらのN-アセチル化体などの代謝産物が高値を示すことが報告されている56).各種のがんでは,遺伝子変異,遺伝子増幅,増殖因子やシグナル伝達の活性化,炎症やホルモンの影響によってMYCが高発現あるいは活性化している50).MYCはターゲット遺伝子であるポリアミン合成経路の律速酵素ODC1遺伝子の転写を誘導するため57),各種のポリアミン類が生合成される.ポリアミンは正に帯電しており,DNAやRNAに結合するため,遺伝子発現,翻訳,細胞増殖,膜安定性,器官形成などの生体反応に影響を与える58, 59).近年の研究によって大腸がん,膵臓がん,乳がんなどの患者の唾液でもポリアミン類が高値を示していることが見いだされ,唾液ががん患者のスクリーニングに有用であることが示された60, 61).しかし,臨床検査などで実用化するためには膨大な数の検体を短時間に処理できる測定法が必要であるが,ポリアミンなどの低分子は抗体の作製が難しく,ELISA(enzyme-linked immunosorbent assay)などの迅速測定手法の開発が困難であった.
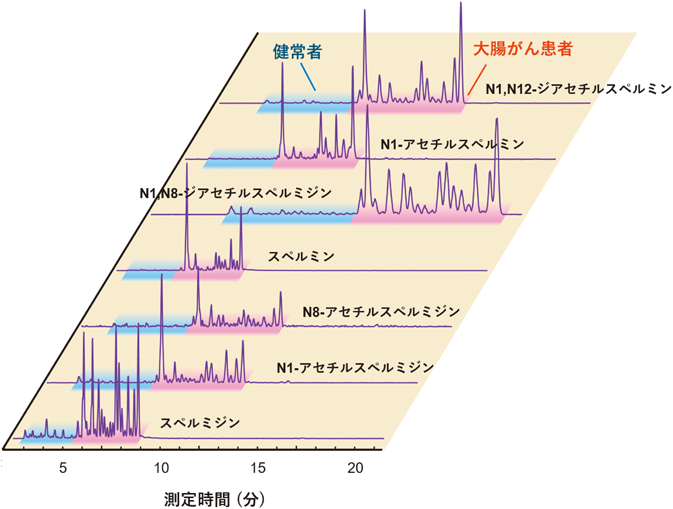
そこで筆者らは,前述の多検体同時分析CE-MS法を唾液中のポリアミン類の測定に応用した43).図7に健常者(青で示した前半の20例)と大腸がん患者(赤で示した後半の20例)から採取された唾液中の7種類のポリアミンを測定した結果を示した.健常者からはポリアミン類のピークはほとんど検出されていないのに対して,大腸がん患者からは高濃度のポリアミンが検出された.また,40検体を40分以内で測定した.
本法を用いて,359例(健常者57例,大腸良性腫瘍26例,大腸がん276例)の唾液中のポリアミン類を測定したところ,N1-アセチルスペルミジン,N1-アセチルスペルミン,N1,N12-ジアセチルスペルミジンなどのポリアミンは,大腸がん患者では高値を示した.N1-アセチルスペルミンを用いると83.4%の精度で大腸がん患者を健常者と大腸良性腫瘍から区別した43).本法のスクリーニング精度は,既存の大腸がんのタンパク質マーカーであるNSE(neuron-specific enolase:76.6%),CEA(carcinoembryonic antigen:68.2%),CA19-9(56.0%)などよりも優れた結果を示した.
ここでは,唾液による大腸がんのスクリーニングを示したが,膵臓がん,肺がん,乳がん,口腔がんでも唾液中のポリアミン類が増加する.バイオベンチャーのサリバテック社では,すでにいくつかのポリアミンを組み合わせることでこれらのがんのリスクを検査するビジネスを展開している.
本稿で示した多検体同時分析CE-MS法による唾液中のポリアミン類の測定は,1検体を1分以内に測定できる迅速な分析手法であるばかりでなく,スクリーニングの精度も優れており,今後有望ながんのスクリーニング手法になると思われる.