これまでリボソームや一般的な翻訳制御因子に対する阻害剤はほぼすべてのmRNAの翻訳に一様に影響すると考えられ,mRNA特異的な効果は多くの場合見逃されてきた.これは,これまで多くの研究が生化学的ないし分子生物学的手法により,特定のreporter mRNAの翻訳動態を計測するにとどまっており,多くの種類,配列多様性を持ったmRNAを同時並行的に実験することが困難であったことによる.
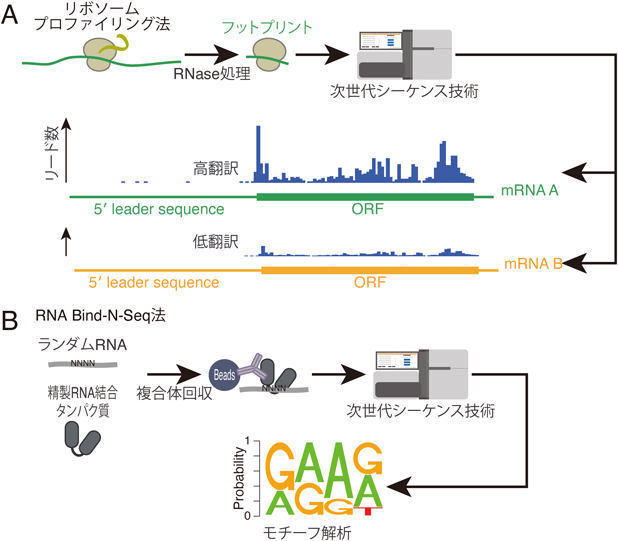
この状況はリボソームプロファイリング法(またはRibo-Seq法とも呼ぶ)の登場によって大きく様変わりした4, 5)(図1A).リボソームプロファイリング法は,次世代シーケンス技術を利用したものである.翻訳中,リボソームはmRNAに結合し,その上を3′方向へ移動しながらコドンを読み取っていく.このようなmRNA-リボソーム複合体に対し,RNaseで処理すると,リボソームが結合していない大部分の領域は途端に分解してしまうが,リボソームが直接結合するmRNAの領域はRNaseの分解から保護される(図1A).最終的に残ったRNAの断片をリボソームフットプリントと呼ぶ.これらを回収し,次世代シーケンサーで解析することにより,どのmRNAのどのコドンにどの程度リボソームが存在したのか,ということを網羅的にかつ定量的に議論することが可能である.
この技術を応用することで,翻訳阻害剤が与える影響をつぶさに理解することができるようになった.比較的新規の翻訳阻害剤の影響のみならず,古典的なリボソーム結合型の阻害剤であっても見逃されていた特異性を見出されている.これまで汎用で利用していた化合物に関してもその効果をあらためて見直すことが重要である.
リボソームプロファイリング法に加え,生化学と次世代シーケンサーを組み合わせた他の手法も有用である.たとえばあるRNA結合タンパク質に対し,その特異性を網羅的に検証する手法としてRNA Bind-N-Seqがある.この手法は,精製したRNA結合タンパク質と,ランダムな配列を持つRNAをin vitroで反応させ,RNA結合タンパク質に結合したRNA画分だけを単離,次世代シーケンサーで解析する6)(図1B).この実験はランダム配列を持つRNAを用いているので,1回の実験ですべてのRNAの配列の組合わせを検証することができる.この手法をeIF4Aおよび化合物が作用した前後で解析することで,化合物がeIF4AのRNA選択性に影響を与えるか,という点を網羅的にかつ定量的に計測することができる7–10).
1)Rocaglateの薬理学的効果
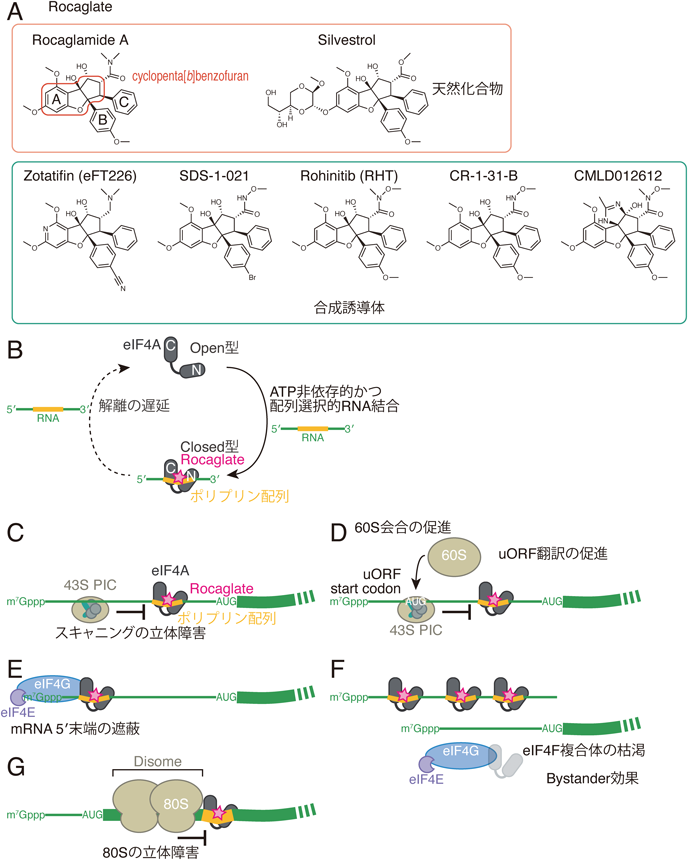
Rocaglateはcyclopenta[b]benzofuran(シクロペンタ[b]ベンゾフラン)構造をコアとする低分子化合物の総称である(図3A).この化合物は真核生物のeIF4Aを標的とする翻訳阻害剤として同定されてきた21–23).
Rocaglateは,抗がん作用を持つことから大きな関心を集めている24).現在までに,200を超えるRocaglate誘導体が天然に単離,または合成されている24–27)(図3A).Rocaglateは,リンパ腫21, 28),白血病29–32),腺がん24, 33),神経芽細胞腫34),骨髄腫35),あるいはBRAF(V600)変異等による抗がん剤耐性がん細胞36–38)など,さまざまなタイプのがんに対して効果を示す.抗がん作用に加え,マクロファージの極性化39, 40),抗原虫活性41),抗真菌活性42, 43),抗ウイルス活性44–47)など,多様な疾患の治療や予防に関しても有望な活性も示す.特に合成誘導体であるZotatifin(ゾタチフィン,eFT226としても知られる)27)(図3A)は固形腫瘍とCOVID-19の治療薬として臨床試験に入っており,将来の臨床応用が期待される.
2)Rocaglateの作用機序
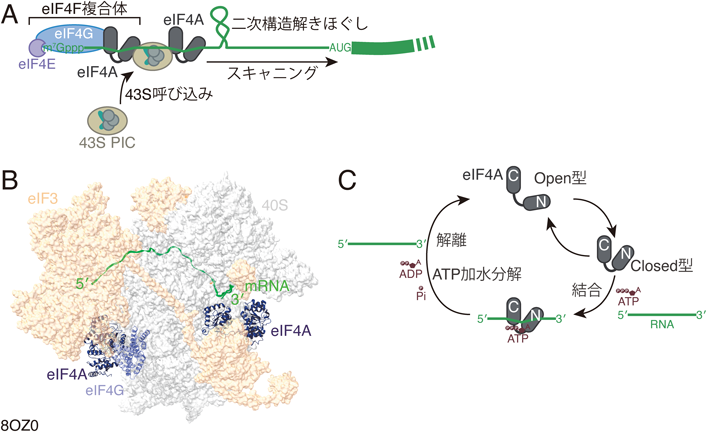
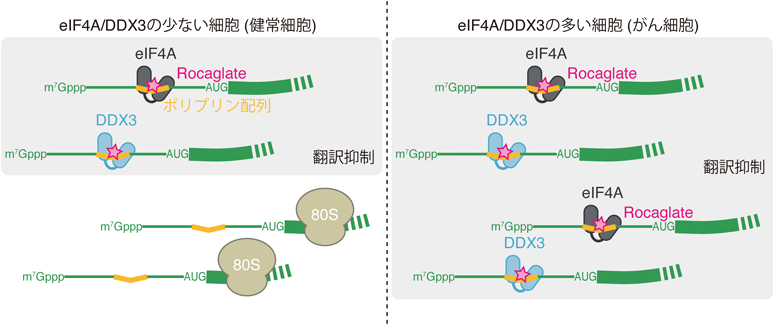
上述のようにeIF4Aは通常,ATP依存的RNA結合を介し,翻訳を活性化させる因子として機能する.しかし,RocaglateはこのようなeIF4Aの機能を一変させる.RocaglateがeIF4Aに作用することにより,A/G塩基の反復配列を持つRNA(polypurine,ポリプリン)に選択的に結合するようになる7–9, 25, 26, 48, 49)(図3B).この配列選択的RNA結合は,eIF4Aが持つATP依存性を無視し,ATPの存在の有無にかかわらず,誘導される7–9, 25, 26, 49).つまり,RocaglateによりeIF4Aの性質がATP依存的かつ配列非依存的RNA結合タンパク質からATP非依存的かつ配列依存的RNA結合タンパク質へと大きく変容するということを意味している(図3B).細胞の中で,mRNAとeIF4Aとの結合はeIF4E, eIF4Gと協調するが,Rocaglateはこのような因子とは独立に作用し,eIF4AをmRNAに直接結合させる7).このような配列特異性の俯瞰的探索にはRNA Bind-N-Seq法が奏功している7, 9).
このようないわば人工的なRNA選択性の獲得により,eIF4Aは翻訳の活性化因子ではなく,翻訳の抑制因子として機能するようになる7).実際にリボソームプロファイリングにより解析すると,特定のmRNAがより選択的に翻訳抑制されることが報告されている7, 50, 51).その翻訳抑制誘導のメカニズムとして以下の五つのモードがある.
i.スキャニングの立体障害
たとえば,ポリプリン配列がmRNAの5′ leader sequenceにある場合,長期間eIF4Aが結合したままになることで43S PICのスキャニングの立体障害となり,mRNAからのタンパク質合成を,翻訳開始段階で阻害する(図3C)7).5′ leader sequence上にポリプリン配列が多ければ多いほど,効果が高くなり,実際,リボソームプロファイリングを行うと配列数依存的な翻訳抑制効果が観察される.
ii.uORF翻訳の誘導
mRNAの5′ leader sequenceには通常翻訳開始因子としては使われにくい翻訳開始コドンがある.これらはKozak配列が適切ではない,あるいはAUGではなくCUG/GUGといったsub-cognate配列を持っているなどの理由で通常翻訳開始コドンとして認識されない.このような5′ leader sequenceに存在するORFはupstream ORF(uORF)と呼ばれる.しかしながら,最適でない配列周辺にRNA結合タンパク質や強力なRNA二次構造などがあり,立体障害によって43S PICのスキャニングが遅れると,開始コドンとして認識する確率が高まることが知られている52–56).RocaglateによってmRNAに強固に結合したeIF4Aも立体障害になるので,5′ leader sequenceから翻訳が開始されやすくなる(図3D).すると,上流で翻訳が完結してしまい,下流のORFからの翻訳に必要なリボソームの供給が抑えられる(図3D).この機構により,Rocaglateによる翻訳開始抑制の効果をさらに強める7).
iii.mRNA 5′末端の遮蔽
mRNAの5′末端にはm7Gキャップ構造があり,eIF4E–eIF4G–eIF4A三量体複合体(eIF4F複合体)が結合する.ポリプリン配列がm7Gキャップ構造の直下に存在するような場合,eIF4F複合体全体がRocaglateによりつなぎ止められてしまい,m7Gキャップ構造が次の翻訳開始に使われなくなってしまうことで,翻訳開始抑制を誘導する26)(図3E).
iv.bystander(バイスタンダー)効果
RocaglateがmRNA上にeIF4Aを長時間つなぎ止めてしまうことで,eIF4F複合体として参加できるeIF4Aの総量は減少する.これにより,翻訳開始の効率全体が減少する.この効果はmRNA上のポリプリン配列の有無にかかわらずmRNA全体に起きることから,bystander(バイスタンダー,傍観者)効果と呼ばれる26)(図3F).
v.リボソーム停滞の誘導
i~ivについては翻訳の開始に影響を与えるものであるが,翻訳の伸長過程にもRocaglateは影響を及ぼす.RocaglateはeIF4AをmRNAのORF中に存在するポリプリン上にも結合させ続け,最終的に翻訳伸長中のリボソームの立体障害にもなる57)(図3G).停滞したリボソームに次にやってくるリボソームが衝突し,リボソームの渋滞が生じる.この複合体をdi-ribosome(disome)と呼ぶ.
以上のように,RocaglateはeIF4Aのloss-of-function型の一般的阻害剤ではなく,gain-of-function型の特殊な化合物として働く.Rocaglateは単にeIF4A阻害剤と紹介されることもあり,eIF4Aの機能を抑える(loss-of-function)と単純化されて考えられがちであるし,そのような文脈で使用している論文も数多く見受けられる.しかし,実際はRocaglateはeIF4Aのgain-of-function型薬剤として機能し,上記のような多様な作用メカニズムで効果を発揮する(図5参照).
その特殊性は,その効果がドミナントネガティブ効果を示すことからもうかがい知れる.通常の阻害剤であれば,標的のタンパク質(この場合eIF4A)が多ければ多いほど,阻害剤の効果が拮抗し,弱まるはずである.しかし,Rocaglateの場合はeIF4Aが多ければ多いほど,ポリプリン配列に結合するeIF4Aも増加し,より阻害剤の効果が強くなる7, 9).
これらの翻訳抑制機構に加え,Rocaglateの長期投与によってより複雑な状況が誘導されることが示されている.たとえば,一部のmRNAの翻訳がむしろ選択的に上昇するということが,最近のプロテオミクス研究で報告されている58).
3)RocaglateとeIF4A–RNAとの結合様式
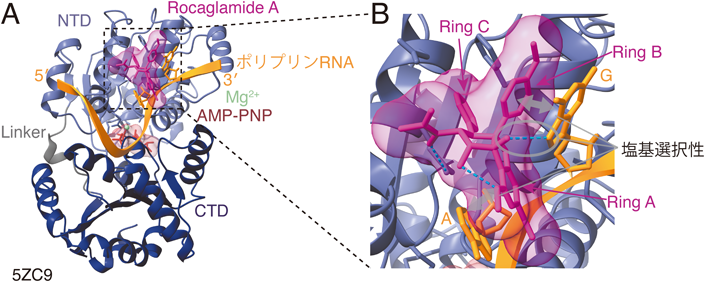
eIF4A1・ポリプリンRNA・RocA(Rocaglamide A,代表的なRocaglate)・AMP-PNP(非加水分解性ATPアナログ)の結晶構造から,その配列選択性の分子メカニズムが明らかになっている8).eIF4A上に結合したRNAはその途中で鋭く湾曲する.RocAはその湾曲部とeIF4Aが形成するポケットにはまり込む(図4A).RocAはタンパク質側,RNA側両側と相互作用するが,その中でRocAはAおよびG塩基と水素結合およびπ-πスタッキングを介して直接相互作用する(図4B).特に水素結合はピリミジン塩基とは形成できないことから,RocaglateによってeIF4Aにポリプリン配列選択性が課されることが説明できる.
4)Rocaglateの抗がん作用を決定する因子
ではなぜ,Rocaglateはがん細胞選択的に増殖を抑えることができるのであろうか? これまでに複数の説明がなされており,これらが複合的に作用していると考えられる.
i.がん原因遺伝子の翻訳阻害
たとえば,MYC, MYB, NOTCH, CDK6, BCL2などのがん原因遺伝子の翻訳が特にRocaglateに影響を受けやすいということが報告されている51).これは偶然にもこれらのmRNAにポリプリン配列が多く,Rocaglateによる翻訳抑制を受けやすいため,と説明される.
ii.ドミナントネガティブ効果
これとは独立に,Rocaglateの標的タンパク質の発現量が細胞の感受性を決めるという説明もある.上記のように,Rocaglateはドミナントネガティブに働くことから標的タンパク質が多ければ多いほど,翻訳抑制効果が大きくなる.近年,eIF4Aに加えてDDX3と呼ばれる別のDEAD-box型RNA結合タンパク質がRocaglateの標的として同定された9).DDX3はeIF4Aと同様に翻訳開始因子として働くことが知られている59–64).DDX3に対するRocaglateの影響は,eIF4Aに対する影響と同様であり,ポリプリン配列上にDDX3をつなぎ止めるという効果が生じ,翻訳を抑制する.がん細胞は一般的に高い増殖能を支えるために翻訳が亢進しており,一部のがん細胞ではeIF4AおよびDDX3が過剰発現している.実際にeIF4A/DDX3の発現量とRocaglateによる細胞増殖抑制効果は正の相関関係を示すことが報告されており9),ドミナントネガティブ効果が細胞感受性を決定する一要因として働くだろう(図5).
iii.その他の要因
さらに,ABCB1に代表される多剤排出型トランスポーターの発現レベルやその調節などがRocaglateの細胞増殖抑制活性の決定因子としても報告されている65, 66).
5)Rocaglate産生植物と菌との間の生存競争
i.AglaiaにおけるRocaglateに対する自己防衛
Rocaglateは元来Aglaia(和名 樹蘭)と呼ばれる東南アジア(日本では沖縄)で生育する植物の二次代謝物として同定された67).そのため,Rocaglateはこの植物にとっての抗菌薬・抗虫薬として働いているのではないか,と考えられてきた.しかし,この植物もeIF4AおよびDDX3を利用して翻訳をしているはずであり,阻害剤とその標的が植物体内に共存する状況にある.このことから何らかの方法でAglaiaは自分自身への攻撃を回避しているはずである.
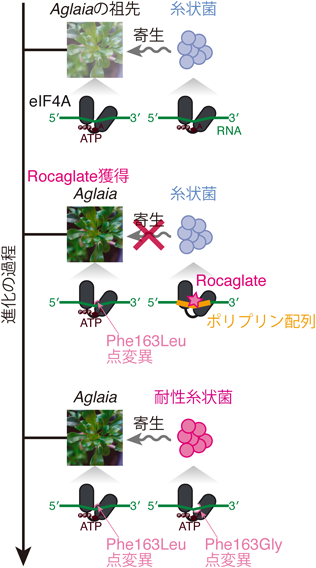
Aglaiaのtranscriptome de novo assemblyからAglaiaが持つeIF4Aのアミノ酸配列を解析すると,進化的に比較的新しい点変異が複数獲得されていることが明らかになった(Phe193LeuおよびIle199Met,ヒトeIF4Aでのアミノ酸番号)8)(図6).これらの点変異は構造上,eIF4AのRocaglate結合ポケットに導入されていた.特にPhe163Leuはポケットの穴を埋めるような変異であり,この結果,RocaglateがAglaiaではまったく作用できないよう進化していることが明らかになった.
ii.Aglaiaに寄生する真菌のRocaglate耐性
Rocaglateはあらゆる真核生物に保存されたeIF4Aを阻害するため,抗菌薬として機能し,原理的にはAglaiaには真菌/カビは生えないはずである.しかしながら,この前提に反し,Aglaiaに寄生する糸状菌が発見された(Ophiocordyceps sp. BRM1株)49).この糸状菌はアリなどに感染しゾンビ化させることで知られる冬虫夏草の仲間である.この寄生糸状菌のtranscriptome de novo assemblyからそのeIF4Aの配列を解析するとPhe163Gly(ヒトeIF4Aでのアミノ酸番号)という点変異が導入されていた(図6).これはAglaiaで見つかっているRocaglate抵抗性点変異が導入されているのとまったくおなじアミノ酸残基に当たる.AglaiaのPhe163Leuがポケットの穴を埋めるのに対し,寄生糸状菌のPhe163Glyはポケットの穴を大きくする方向に働く.このように同じ位置の点変異であるが,その構造に対する効果は真逆の方向に抵抗性が獲得されてきたことが興味深い.寄生糸状菌には,Aglaia上では他の競合菌が存在しないため(Rocaglateによって死滅する),植物からのリソースを専有できる優位性が発生する.
このように,植物が抗菌薬としてRocaglateを獲得すると同時に,菌側もRocaglate抵抗性を獲得する,という化合物を中心とした生物間相互作用が生じている点は非常に興味深い(図6).なお,AglaiaではDDX3も点変異を獲得し,Rocaglate耐性になっているが9),寄生糸状菌のDDX3には耐性変異は導入されておらず49),現在も糸状菌側が最適化の過程にある可能性がある.
1)Pateamine Aの薬理学的効果
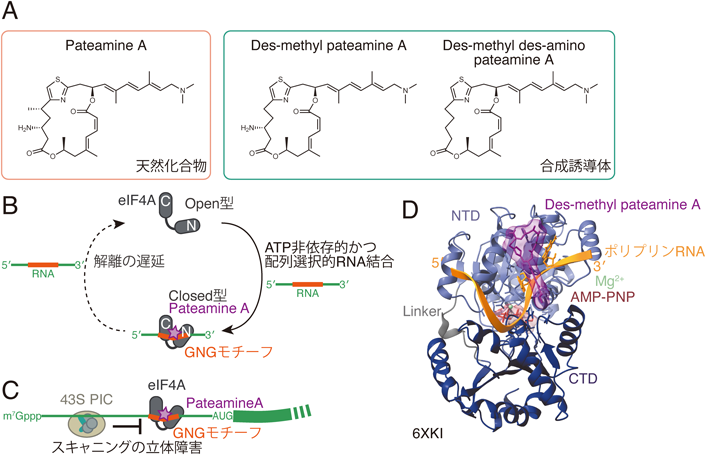
Pateamine Aは海綿(Mycale sp.)に共生する細菌群が合成する二次代謝物である68–70).Pateamine Aはメラノーマ(悪性黒色腫),BRAF(V600)変異等による抗がん剤耐性がん細胞37)をはじめとする多様ながん細胞の増殖を特異的に抑制することが知られる71, 72).Pateamine Aの化学合成の難しさから,より合成が簡便なdes-methyl pateamine Aやdes-methyl des-amino pateamine Aも多く用いられる(図7A)(本稿ではまとめて単純にPateamine Aと呼称する).
2)Pateamine Aの作用機序
Pateamine AはeIF4Aを標的とする翻訳阻害剤として機能することが知られていたが,その作用メカニズムについては諸説あり,明確になっていなかった72–79).近年,RNA Bind-N-Seqを用いた解析から,Pateamine AがRocaglateと同様に,eIF4AをRNA配列選択的かつATP非依存的RNA結合タンパク質へと変容させることが報告されている10)(図7B).RocaglateがA/Gの連続配列であるポリプリンに選択的であるのに対し,Pateamine Aの場合,GNG(NはどのA/U/G/C塩基でもよい)モチーフを選り好む.その一方で,eIF4Aを5′ leader sequenceに係留させ,43S PICの立体障害として翻訳開始を抑制する点はRocaglateと同様である10)(図7C).実際にリボソームプロファイリング法で解析すると,GNGモチーフ依存的にmRNA選択的翻訳抑制が誘導される.
Pateamine AはRocaglateと同様にDDX3に対しても作用し,GNGモチーフ選択的かつATP非依存的RNA結合能を与えることが報告されている10).
3)Pateamine AとeIF4A–RNAとの結合様式
結晶構造解析からPateamine AのeIF4AおよびRNAとの結合様式が報告されている79).RocaglateとPateamine Aはその化学構造上まったく別物の化合物であるのにかかわらず,eIF4AとRNAが形成するまったく同じポケットを利用する(図7D).RocaglateとPateamine Aはそれぞれ植物,海綿動物(に共生する細菌群)に由来するが,それぞれが収束進化として同様のポケットを標的する点で非常に興味深い.
この構造を基盤に,分子動力学計算とフラグメント分子軌道法による量子計算により,Pateamine Aのtertiary amine基が持つ正電荷と,近接するG塩基O6の負電荷の相互作用によりGNG選択性が生じることが示されている10).
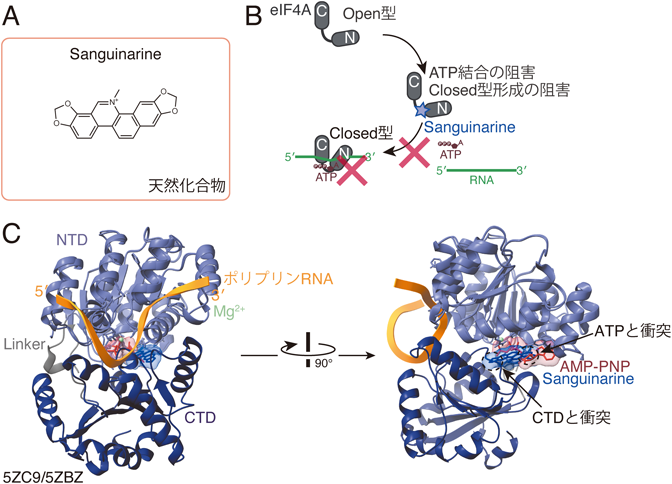
6. Hippuristanol(ヒップリスタノール)
1)Hippuristanolの薬理学的効果
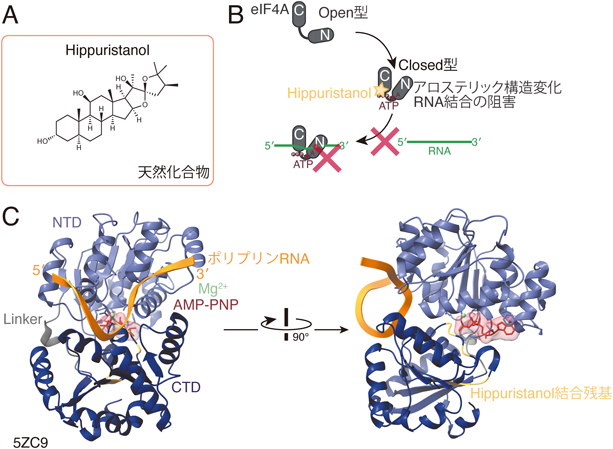
Hippuristanolは軟体サンゴ(Isis hippuris)から単離された化合物である20, 80–83)(図8A).リンパ腫80, 84),BRAF(V600)変異等による抗がん剤耐性がん細胞36, 37)などのがん細胞の増殖を抑えることが報告されている.
2)Hippuristanolの作用機序
HippuristanolはeIF4AとRNAと間の結合を弱めることが報告されている20, 81–83)(図8B).つまり,HippuristanolはeIF4Aのloss-of-functionに近い効果を表すと考えられる.リボソームプロファイリングによる解析から,一群のmRNA,特に5′ leader sequenceの長いmRNAや5′ leader sequenceに二次構造を多く含むmRNAがよりHippuristanolに高い感受性を示すことが判明している7, 20).
3)HippuristanolとeIF4Aの結合様式
HippuristanolとeIF4Aの結合を示す構造解析はいまだ報告がないが,NMR解析によりCTDに結合することが示唆されている82)(図8C).この領域はATP結合部位に相当する一方で,HippuristanolはeIF4AとATPの結合を阻害しないことから81–83),アロステリックな構造変化を誘導することによりeIF4AとRNAとの結合を減弱させていると理解されている(図8B).eIF4A上のHippuristanol結合領域はアミノ酸配列上,他のDEAD-box型RNA結合タンパク質と異なっており,これによりeIF4Aに対する特異性が生じていると考えられる82).