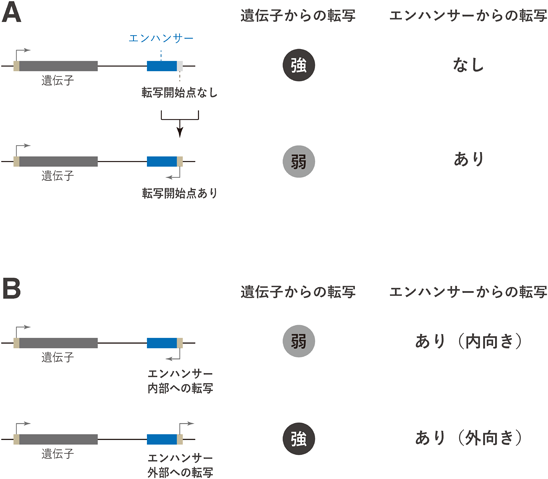
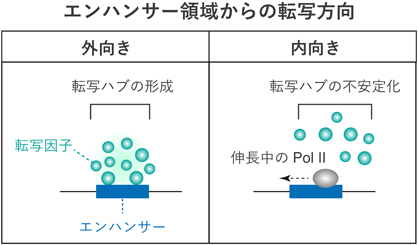
エンハンサー領域における非コードRNA転写の生物学的機能Function of non-coding enhancer transcription in gene regulation and genome evolution
東京大学定量生命科学研究所Institute for Quantitative Biosciences, The University of Tokyo ◇ 〒113–0032 東京都文京区弥生1–1–1 生命科学総合研究棟404 ◇ Life Sciences Research Building 404, 1–1–1 Yayoi, Bunkyo-ku, Tokyo 113–0032, Japan