2. スフィンゴ脂質産生細菌と細菌由来スフィンゴ脂質の化学構造
これまでに細胞膜の成分として含まれるスフィンゴ脂質の構造解析結果が報告された細菌は,筆者の知る範囲ではあるが,プロテオバクテリア門では,α-プロテオバクテリア綱のAcetobacter属,Caulobacter属,Sphingomonas属,Novosphingobium属とδ-プロテオバクテリア綱のMyxococcus属,Bdellovibrio属,そして,バクテロイデス門のBacteroides属,Parabacteroides属,Prevotella属,Porphyromonas属,Flectobacillus属,Sphingobacterium属,Alistipes属,Capnocytophaga属,Chryseobacterium属,Flavobacterium属,Odoribacter属とクロロビウム門のChlorobium属の細菌の一部があげられる5–16).脈絡なくさまざまな門や綱にスフィンゴ脂質含有細菌が分布している印象であるが,列挙した細菌の多くが真核生物などの宿主と共生することが興味深い共通点である.たとえば,ショウジョウバエや線虫におけるAcetobacter属細菌による共生現象はマイクロバイオーム研究における有用なモデル系となっている17–19).Bacteroides属やPrevotella属の細菌はヒトの腸内細菌叢の優勢種である20–22).Sphingomonas paucimobilisやSphingobacterium spiritivorumは,時にヒトの消化管感染症や日和見感染症を引き起こす院内病原菌として単離されるが,もともとは植物の根圏に由来する土壌細菌である23, 24).Tannerella forsythiaやPorphyromonas gingivalis, Capnocytophaga ochraceaはヒトにおける口腔内常在菌であると同時に,歯周病原菌として有名である25–27).Capnocytophaga属には,近年問題になっている人獣感染症の原因菌が含まれる28).Chryseobacterium gleumも日和見病原菌である16).Alistipes属には,大腸炎や肝硬変に対する保護効果やインスリン抵抗性の改善効果が報告された腸内細菌が含まれ29),Odoribacter属には,長寿者の顕著な便中代謝物として話題になった二次胆汁酸(イソアロリトコール酸)の産生菌が含まれる30).
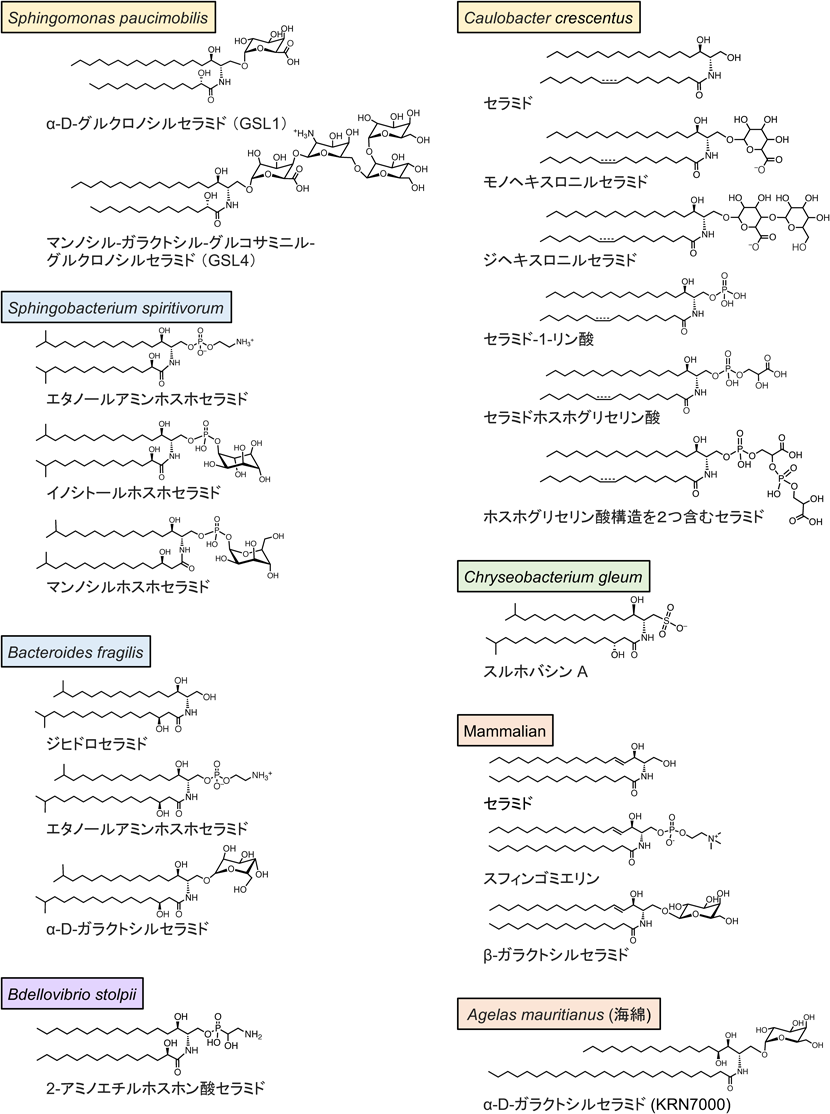
Sphingomonas paucimobilisは1990年に薮内らにより報告された好気性グラム陰性桿菌である31).グラム陰性細菌の特徴的成分であるリポ多糖を含まず,代わりに大量のスフィンゴ糖脂質を膜の主要成分として含んでおり,その化学構造はセラミドにグルクロン酸がα結合したα-D-グルクロノシルセラミドであることが判明している32)(図1).これまでに調べられたSphingomonas属およびその近縁属の細菌のいずれの株においても,D-グルクロン酸やD-ガラクツロン酸などの糖酸一つが付加されたGSL1シリーズのセラミドが主たるスフィンゴ糖脂質であると報告されているが33),四糖が付加されたGSL2, GSL3, GSL4シリーズやスフィンゴイド塩基がシクロプロパン構造を含む炭素数21のアシル基,あるいは不飽和結合を含む炭素数20のアシル基からなるスフィンゴ糖脂質も検出されている34, 35).また,アミド結合脂肪酸のアシル基のC2位にはヒドロキシ基を含み,これによって隣接する糖脂質の親水性頭部基のヒドロキシ基と水素結合ネットワークを形成して膜構造を安定化すると推測されている36).Sphingomonas属細菌に含まれるGSL4と呼ばれるスフィンゴ糖脂質の糖鎖にはグルコサミンのようなアミノ糖が含まれており親水頭部基の負電荷が中和される.Sphingomonas属細菌の中にはポリエチレングリコールや多糖類の一種であるアルギン酸などを高分子のまま細胞内に取り込んで分解してエネルギー源として利用する種が存在する37–39).その細胞表面,すなわち外膜には,さまざまなポリマーを取り込むための,くぼみの深さが76–147 nm程度の「体腔」の形成が観察されており,発見者により“mouth-like pit”と名づけられ,さらに内膜に存在するアルギン酸取り込み輸送・分解を担うABCトランスポーターや分解酵素も同定されている40, 41).細菌外膜中にリポ多糖の代わりにスフィンゴ糖脂質が存在することで,より柔軟でしなやかな構造が形成される可能性があり,“mouth-like pit”形成と関連するのかもしれない.
Sphingomonas属に先駆けて1983年に薮内らにより分類されたSphingobacterium属の細菌は,高濃度のスフィンゴリン脂質を含んでいることが知られており42),主成分の化学構造は,セラミドにホスホエタノールアミンが結合したエタノールアミンホスホセラミド,イノシトールホスホセラミド,マンノシルホスホセラミドである(図1).アミド結合脂肪酸はC2位にヒドロキシ基が付加されていることが多い43).スフィンゴリン脂質を含む細胞膜における脂質分子どうしの水素結合による横方向の相互作用は,スフィンゴ糖脂質からなるSphingomonas属の細菌の細胞膜におけるそれよりも弱いと予想されている.
Bdellovibrio属の細菌は,小型の曲がった桿菌で,海水や淡水,下水,土壌など多様な環境に生息し,他のさまざまな大型グラム陰性細菌に侵入し,被食者のペリプラズム空間内で寄生増殖し,栄養が尽きると被食者を溶菌させて出ていくという非常にユニークな細菌捕食作用を有する44).Bdellovibrio stolpiiに含まれるスフィンゴ脂質は,炭素−リン結合のある特異なスフィンゴホスホノ脂質である2-アミノエチルホスホン酸セラミドと報告されている45, 46)(図1).
バクテロイデス門に分類される細菌が合成するスフィンゴ脂質は,全脂肪酸プールの55–96%が分岐型のアシル基(イソ分岐脂質)であり47),この点が真核生物由来のスフィンゴ脂質との化学構造上の大きな違いである.偏性嫌気性細菌Bacteroides fragilisはヒトの口腔内から腸内までの細菌叢を構成する優勢菌であり,日和見病原菌でもあり,膜リン脂質の40–70%に及ぶスフィンゴ脂質を含む48).B. fragilisの産生するスフィンゴ脂質で最も多いのはエタノールアミンホスホセラミド,第二成分がホスホエタノールアミンが付加されていないジヒドロセラミド,第三成分はスフィンゴ糖脂質のα-D-ガラクトシルセラミドである4)(図1).
スルホノリピド(スルホバシンA)は,スフィンゴイド塩基部分にヒドロキシメチル基ではなく,スルホン酸基を持つ珍しいスフィンゴ脂質である10)(図1).今のところスルホノリピドを合成する細菌はバクテロイデス門に限られており,これらの細菌においてスルホノリピドと他のスフィンゴ脂質が同時に検出された報告はない16).
細菌由来のスフィンゴ脂質構造の多様性は,哺乳類に比べるとあまり解明されていない.しかし,哺乳類のスフィンゴ脂質が主に偶数鎖の直鎖スフィンゴイド骨格を持つのに対し,細菌スフィンゴ脂質のスフィンゴイド骨格やそれに結合した脂肪酸は,スルホノリピドも含めて,アシル基が奇数鎖長であることが多い.また,メチル化,水酸化,ホスホエタノールアミン,ホスホグリセリン酸またはホスホイノシトール,糖鎖などによって修飾されている傾向がある.
自然環境下におけるスフィンゴ脂質含有細菌の生存や,スフィンゴ脂質含有細菌と宿主の相互作用において,細菌側のスフィンゴ脂質の合成や代謝という営みがどの程度重要な要素なのかについては,現段階においてはまだほとんど解明されていないが,これまでに報告されている知見を紹介する.
Caulobacter crescentusは淡水の湖や小川に広く分布している貧栄養細菌(低栄養性細菌)である.特徴的な三日月型の形態やクラスター化,遊走細胞と固着性細胞への分化と非対称細胞分裂,ユニークな代謝酵素の活性制御など数々の興味深い特質を示すことから細胞周期研究のモデル生物として研究されている49–51).C. crescentusは,リン脂質の合成が制限されるようなリン酸飢餓状態下では,二糖結合型のセラミドGSL2を合成して膜組成を変えて環境的制約に適応する52).C. crescentusはグラム陰性菌でありながら,リピドA合成能を欠損しても生存可能で,セラミドにホスホグリセリン酸が一つあるいは二つ付加されたアニオン性スフィンゴ脂質を合成する53)(図1).C. crescentusのスフィンゴ脂質合成欠損株においては,カチオン性のポリペプチド系抗生物質であるポリミキシンBやコリスチンに対する耐性が増加したことから,これら抗生物質の標的が細胞膜に含まれるアニオン性スフィンゴ脂質であることが判明している52, 53).また,リン酸飢餓状態のC. crescentusはファージ感染への感受性が増加する.C. crescentusは,130 kDaのRsaAタンパク質からなるS層と呼ばれる準結晶表面を有しているが,スフィンゴ糖脂質の産生が増加するとS層の状態が変化し,その結果ファージの感染・増殖が容易になるのではないかと推察されている52).
Sollichらは,高熱の海底堆積物中にBacteroides属細菌に由来すると推測されるスフィンゴ糖脂質が豊富に存在することを発見し3),スフィンゴ糖脂質が細菌の環境生息域での熱ストレスに対する抵抗性を付与している可能性を示した.具体的には,細菌は,膜脂質としてスフィンゴ糖脂質を合成し,そのアミノ基に由来する水素結合形成により脂質分子の充填を密にすることで,過酷な高温高圧環境下での細胞膜の安定性と剛性の向上を達成しているのではないかという仮説を提唱している.
スルホノリピドは細菌の滑走運動に必要であることが示されている54–57).また,Cyclobacterium属細菌から単離されたスルホノリピドの一種のスルホバシン類については血小板凝集に必要なフォン・ビルブラント因子のアンタゴニスト作用やDNAポリメラーゼに対する阻害作用が報告されている58, 59).
襟鞭毛虫Salpingoeca rosettaは動物に最も近い単細胞真核生物であるが,餌となるAlgoriphagus machipongonensisとその近縁種の細菌が産生するロゼット誘導因子(RIF-1)と名づけられたスルホバシン類似のスルホノリピドによって,この襟鞭毛虫のロゼットコロニー形成が誘導される13).RIF-1は非常に高い活性を有し,フェムトモルオーダー(10−15 M)という極微量で効果を発揮することが確かめられている.細菌由来のスルホノリピドが真核生物の形態形成を誘導する典型例とされており,細菌が動物の進化に寄与した可能性を示唆する興味深い現象である.
一方,土壌細菌Chryseobacterium gleum由来のスルホバシンは複数種類のヒトがん由来培養細胞に対する細胞毒性や60),マクロファージに対する炎症性サイトカインの発現誘導作用16)など炎症促進活性を有する.メタゲノム解析によりマウスの盲腸においてAlistipes属とOdoribacter属の細菌がスルホバシンを含むスルホノリピドを合成しており,高脂肪食を与えたマウス盲腸では合成量が増加することが報告されている8).腸内細菌叢と宿主の相互作用におけるスルホノリピドの寄与が今後さらに明らかになると期待される.
現在までのところ,細菌由来のスフィンゴ糖脂質を介した細菌と哺乳類の相互作用で最もよく知られているのは,宿主の免疫系を調節することである.S. paucimobilisが産生するα-D-グルクロノシルセラミドやα-D-ガラクツロノシルセラミドのようなスフィンゴ糖脂質は,CD1d-拘束性ナチュラルキラーT(iNKT)細胞を活性化する61, 62).活性化iNKT細胞からは迅速にサイトカインが放出されて感染時の細菌排除が促進される.S. paucimobilisの近縁種であるNovosphingobium aromaticivoransの肝臓感染はS. paucimobilisと同様にCD1d-拘束性iNKT細胞を活性化し,肝臓と胆管に対する自己免疫反応を原因とする原発性胆汁性胆管炎を発症させることが報告されている63).このような自然免疫における細菌由来スフィンゴ糖脂質の作用は感染症の分野のみならず,がん免疫療法などの創薬分野でも注目されている.沖縄産海綿Agelas mauritianusの抽出液中から発見されたアゲラスフィン類を母化合物とする合成α-ガラクトシルセラミドKRN7000はNKT細胞を活性化する最強のリガンドとして研究試薬としても広く販売されている.アゲラスフィン類の真の生産者は海綿なのか,それとも共生微生物なのかは,現段階では不明である64, 65).
ヒトの歯周病原菌P. gingivalisについては,スフィンゴ脂質合成能を欠損した株と野生株W83株の比較研究の結果,スフィンゴ脂質合成はP. gingivalis菌における表面多糖形成に不可欠であることが報告されている66).スフィンゴ脂質の産生がP. gingivalis菌の定常状態期の生存継続に加え,酸化ストレス耐性や病原因子gingipain活性にも重要であり,スフィンゴ脂質欠損株は野生株と比較してより強力なマクロファージによる炎症反応を誘発する66).さらに,歯肉縁上プラーク,すなわちバイオフィルム内での状態を模した血液寒天培地での培養実験から,スフィンゴ脂質合成欠損株の個体表面での増殖能は野生株よりかなり劣ることが確認されている66).
一方で,スフィンゴ糖脂質を合成できない他の多くの病原体においては,宿主のスフィンゴ糖脂質に依存して感染を成立させ,病原性を発揮する場合がある67, 68).たとえば,ボツリヌス菌69),コレラ菌70)などは細菌毒素のスフィンゴ糖脂質への結合を介して感染を開始し,サルモネラ・チフス菌71),赤痢菌72),マイコバクテリウム属菌73)などはセラミドやスフィンゴミエリンに富んだ脂質ラフトを介したマクロファージ内腔へのエンドサイトーシスを利用して感染を成立させる.緑膿菌(Pseudomonas aeruginosa)は水回りなど生活環境中に広く常在する日和見感染症の起因菌である.緑膿菌は病原因子として溶血性ホスホリパーゼC(PlcH)を分泌する.このPlcHはスフィンゴミエリナーゼ活性を持ち,宿主細胞のスフィンゴミエリンを分解してセラミドを産生する74).沖野らは,緑膿菌のゲノム上には,plcHに隣接してセラミダーゼ遺伝子が配置されていること,緑膿菌感染時には,スフィンゴシン応答性転写制御因子SphRの制御下に宿主由来のスフィンゴ脂質に応答してセラミダーゼの発現が誘導されることを見いだし,菌体外へ分泌されたセラミダーゼはPlcHによる細胞毒性を増強することを報告している75–78).
腸内細菌叢を形成しているB. fragilisやBacteroides thetaiotaomicronなどのBacteroides属の細菌にとって,スフィンゴ脂質合成は細菌自身の腸内環境での生存維持のために必要であり79),対する宿主側にとっては,大腸内のCD1d-拘束性iNKT細胞の恒常性維持,すなわち,環境曝露に対する大腸内のiNKT細胞の耐性を確立するために重要な役割を果たしている80, 81).Olszakらは,無菌(germ-free)マウスにおいてはSPF(specific-pathogen-free)マウスに比べて大腸粘膜固有層のiNKT細胞数が増加しており,このことがかえって炎症反応を亢進させてしまい,薬剤誘発性の炎症性腸疾患やアレルギー性喘息のモデルにおいては,無菌マウスの方がSPFマウスより重症化することを見いだしている80).生後間もない無菌マウスをSPF環境に曝露すると,肝臓や脾臓でのiNKT細胞レベルには影響することなく大腸内のiNKT細胞数がSPFマウスと同程度にまで正常化し,その効果は長期間持続した.薬剤誘発性大腸炎の重症度もSPFマウスと同程度であった.しかし成長後の無菌マウスに同様の処理をしても状態は改善されない80).この観察はヒトの幼児期に微生物に曝露されると,アレルギーや喘息などの免疫介在性疾患の発症が抑制されるという通念に合致する.新生児発達期の宿主の腸内環境が共生細菌由来の抑制性スフィンゴ糖脂質であるα-D-ガラクトシルセラミドにさらされると,大腸におけるiNKT細胞の増殖が阻害され恒常性が変化する.その結果,自己免疫疾患やアレルギー疾患における破壊的なiNKT細胞の活性化が抑制され炎症性免疫疾患から保護されると考えられる81).B. fragilisの産生するスフィンゴ脂質の中でα-D-ガラクトシルセラミドだけで確認される現象であり,スフィンゴ糖脂質の些細な構造の違いが,特異的に正反対の効果をもたらす点でも興味深い.
B. thetaiotaomicronの野生株あるいはスフィンゴ脂質合成能欠損株をそれぞれ無菌マウスへ移植して定着前後の変化を比較するmono-colonization実験において,移植菌体と移植された宿主を対象とする脂質プロファイリング解析の結果,腸内細菌のスフィンゴ脂質合成能欠損を補う代償反応として宿主の内因性スフィンゴ脂質合成が亢進し,その結果として上皮層に炎症の病理組織学的徴候が認められ,宿主のサイトカイン・プロファイル解析からも腸内で炎症が起きていることが示された82).
ゲノム遺伝学とクリックケミストリーアプローチ(BioOrthogonal-Sort-Seq-Spec:BOSSS)83, 84)の応用により,食餌由来スフィンゴ脂質が腸内細菌によってより代謝されてより複雑なスフィンゴ脂質に変換されること84),腸内で産生された細菌由来のスフィンゴ脂質が宿主の上皮組織や肝門脈に移行することが確かめられている85).さらに,B. thetaiotaomicronがマウスの腸内でL-セリンではなくL-ホモセリンをアミノ酸起源とするホモセリン型スフィンゴ脂質を合成し,この脂質が結腸や肝臓組織に移行することが示されている86).このホモセリン型スフィンゴ脂質をHepG2培養細胞に添加すると,解糖系,酸化的リン酸化経路,脂肪酸代謝経路の遺伝子発現が亢進する86).さらに,脂肪除去食を2週間摂取させて肝脂肪症を誘発した無菌マウスに,野生型B. thetaiotaomicronを7日間毎日投与すると過剰な脂肪蓄積の症状が改善した一方で,スフィンゴ脂質合成能欠損株を投与した場合には,このような改善はみられない86).腸内細菌由来スフィンゴ脂質が,直接的あるいは間接的に,宿主の代謝経路を脂肪合成からβ酸化による脂肪分解へと転換させることによって,肝臓での脂肪の蓄積を減少させる可能性が示唆される.
腸内細菌によるL-アラニンやグリシンからのデオキシ型のスフィンゴ脂質産生も報告されている87).真核生物においてデオキシ型スフィンゴ脂質は神経障害や代謝障害に関与する有毒な代謝産物とされることから88),腸内細菌叢によるこれらスフィンゴ脂質の産生もまた宿主に影響を及ぼす可能性がある82, 89).細菌由来スフィンゴ脂質が宿主に送達される仕組みは細胞外小胞が関与している可能性が考えられるが,現段階では解明されていない85).
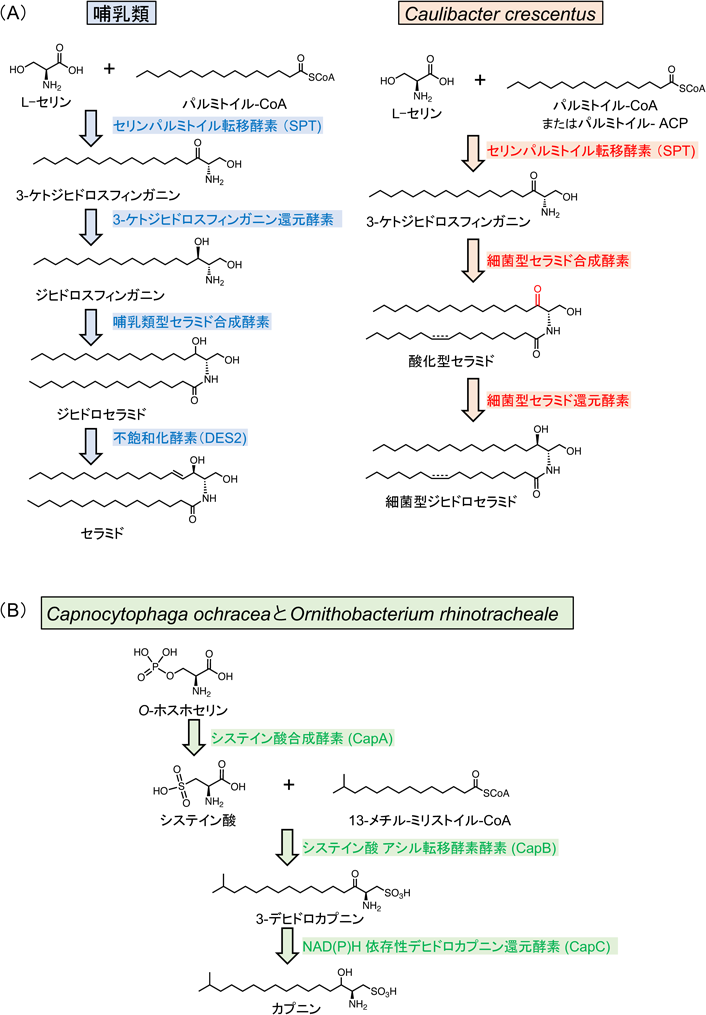
細菌においても,真核生物と同様に,スフィンゴ脂質の生合成は3-ケトジヒドロスフィンゴシンの合成から始まる90, 91).長鎖塩基の化学構造はすべてのスフィンゴ脂質の基本骨格であり,この反応は経路全体の律速段階である.ピリドキサール5′-リン酸(pyridoxal 5′-phosphate:PLP)を補酵素とするセリンパルミトイルトランスフェラーゼ(serine palmitoyltransferase:SPT)によって触媒される90–92).筆者は2001年にSphingomonas paucimobilis SPTを精製し,ペプチドマッピングとゲノムDNAライブラリーからSPT遺伝子をクローニングした90).さらに,Sphingobacterium multivorumやBdellovibrio stolpiiなどの複数種類の細菌由来SPTを対象として,酵素タンパク質の結晶構造解析や反応機構解析研究に取り組んでいる91–102).S. multivorum SPTをクエリー配列としてBLAST検索を行うと,多くの細菌ゲノム中でSPTに特徴的な補酵素結合モチーフ配列「GTFSKS」を含むPLP-依存性酵素,SPT様タンパク質をコードしている遺伝子がヒットする.該当遺伝子はいずれの細菌においてもビオチン生合成経路の初発酵素の8-アミノ-7-オキソノナン酸合成酵素としてアサインされていたが,筆者が確認した範囲ではいずれもがSPT活性を有していた.さらにその前後の配列を調べると,SPT-like geneがヒットした非常に多くの細菌種で,図2に示す四つのORFがクラスターを形成するSPTオペロンとして保存されていた.
上記のSPTオペロンを指標として,近年,C. crescentusとB. thetaiotaomicronを主な標的とした質量分析によるメタボローム解析を組み合わせた遺伝学的スクリーニングによって,いくつかの細菌におけるスフィンゴ脂質生合成経路の大筋が明らかとなり103–105),代謝経路を構成する酵素タンパク質をコードする遺伝子の同定が進められている4, 82)(図3).
細菌SPTの通常のアミノ酸基質はL-セリンであり,グリシンやL-アラニン,L-ホモセリン,L-トレオニオンからも対応する長鎖塩基を合成できる82, 86, 87, 89, 99, 100).筆者が検討した範囲では,いずれの細菌由来のSPTもアシルCoAを基質として代謝でき,細菌の種類によってはアシル基の鎖長や不飽和結合の有無に関して幅広い基質特異性を示す酵素も見いだしている.その一方で,ロドバクテリア(α-,β-,γ-プロテオバクテリア)のSPTは,アシルキャリアタンパク質に結合したチオエステル基質を優先的に用いることが報告されている105).ロドバクテリアは,遊離脂肪酸をスフィンゴ脂質の生合成に向かわせるために,このような複雑な3-ケトジヒドロスフィンゴシンの形成方法を用いているのではないかと推察されている.
真核生物におけるスフィンゴ脂質生合成の第二段階は,3-ケトジヒドロスフィンゴシン還元酵素による3-ケトジヒドロスフィンゴシンからジヒドロスフィンゴシンへの変換である106).ジヒドロスフィンゴシンはBacteroides属の細菌においても検出されており,B. thetaiotaomicronにおいて3-ケトジヒドロスフィンゴシン還元酵素活性を持つタンパク質をコードする遺伝子(BT_0972)が同定された85, 107).
食餌性ジヒドロスフィンゴシンはB. thetaiotaomicronによって選択的に同化され,ジヒドロセラミドに変換されることから84),真核生物と同様のセラミド合成過程が存在することは間違いないと思われる.しかし,細菌においては,哺乳類のセラミドでみられる長鎖塩基C4位とC5位の間の二重結合が導入されず,スフィンゴシン型のセラミドは産生されない.さらに,C. crescentusを対象にした研究から,細菌には二つ目のスフィンゴ脂質生合成経路が存在する可能性が示唆された103).この経路では,まず3-ケトジヒドロスフィンゴシンが細菌型セラミド合成酵素(bacterial ceramide synthase:bCerS)の作用によって「酸化型ジヒドロセラミド」に変換され,次いで細菌型セラミド還元酵素(bacterial ceramide reductase:bCerR)によって長鎖塩基C3位のケト基が還元されてジヒドロセラミドが産生する103).B. thetaiotaomicronに関してもbCerRのオルソログが見つかっており,細菌のジヒドロセラミド生合成経路には少なくとも2系統が存在する可能性が示唆されている103).C. crescentusは真菌類や植物と同様にヒドロキシ基を付加してフィトセラミドを形成できるが,長鎖塩基のC3位ケト基の還元がなくてもヒドロキシ基導入が起きることから,セラミド水酸化酵素が基質としてジヒドロセラミドまたは酸化型ジヒドロセラミドのいずれかを用いることができること,あるいはアシル鎖の水酸化が酸化型ジヒドロセラミドの還元の上流で起こることが示唆されている.
細菌でもジヒドロセラミドがさまざまな,かつ種特異的な親水性頭部官能基の修飾付加反応を受けて複合スフィンゴ脂質が完成する.Bacteroides属の複数種の細菌を対象とした比較研究において,α-ガラクトシルジヒドロセラミド,ジヒドロセラミドエタノールアミン,ジヒドロセラミドホスホイノシトールの三つの主要なスフィンゴ脂質が同定された89).ジヒドロセラミドエタノールアミンはすべての菌種で産生されていたが,α-ガラクトシルジヒドロセラミドとジヒドロセラミドホスホイノシトールには細菌ごとの産生量の違いがみられた.Heaverらは,SPT発現を誘導制御できるB. thetaiotaomicronの菌株を用いてジヒドロセラミドホスホイノシトール生合成におけるイノシトール脂質合成遺伝子クラスターの関与を調べ,B. thetaiotaomicronにおけるジヒドロセラミドホスホイノシトールの生合成において,myo-イノシトールリン酸の合成からジヒドロセラミドホスホイノシトール合成までの4段階の反応を担う四つの遺伝子を同定した104).一方で,Bacteroides vulgatusではジヒドロセラミドホスホイノシトールリン酸を中間体としない別の生合成経路でジヒドロセラミドホスホイノシトールを合成することを報告している104).このホスホイノシトール経路の解析研究に先行して,B. fragilisのα-ガラクトシルジヒドロセラミド産生にはセラミドUDP-α-ガラクトシルセラミド合成酵素が関与することが,沖野らによるZymomonas mobillisのグルクロノシルセラミド合成酵素欠損株を用いた相補実験と生化学解析によって確認されている108).
SPT, bCerS, bCerRがC. crescentusの中心的なセラミド合成酵素として同定されたことから,他の生物種におけるオルソログを探索し,さらに潜在的なセラミド産生能をバイオインフォマティクス的にスクリーニングすることが可能となった.SPT, bCerS, CerRについて米国生物工学情報センター(NCBI)のProkaryotic Representative Genomes Database(5700以上の代表的な細菌)をBLAST解析した結果,17分類群に属する272種において,SPT, bCerS, CerRの3遺伝子のオルソログを含むことが同定された.リピドミクス解析によりStreptomyces aurantiacusにジヒドロセラミドが存在することが確認され,グラム陽性菌にセラミド脂質が存在する証拠が得られた.
スルホノリピドの生合成は,O-ホスホ-L-セリンのスルホン化によって生じたL-システイン酸と13-メチルミリストイルCoAの縮合反応による3-ケトカプニンの合成が初発反応である.3-ケトカプニンの3位のケト基が還元されるとカプニンを生じ,その後,カプニンが別のiso-脂肪酸でN-アシル化されると,スルホバシン類などのスルホノリピドが生成する8).Capnocytophaga属の細菌を対象とした研究によって,スルホバシンの前駆体であるカプニンの生合成に関与する三つの酵素,Capnocytophaga ochracea由来システイン酸合成酵素(CapA),システイン酸-C-アシル転移酵素(CapB)とOrnithobacterium rhinotracheale由来デヒドロカプシン還元酵素(CapC)の遺伝子が同定されている28).CapAとCapBはともにPLP依存性酵素である.ヒト腸内細菌Alistipes finegoldiiのCapBホモログとして,SulAと呼ばれるタンパク質も同定された.スフィンゴ脂質生合成経路の初発酵素であるSPTと同じα-オキサミン合成酵素ファミリーのメンバーであり,X線結晶解析によって酵素活性部位の触媒残基の配向も両者間でよく保存されていることが判明している109).
「真核生物の細胞小器官は,異種生物が細胞内に取り込まれ細胞内で共生することにより生じた」とする「内部共生説」は1970年にMargulisが提唱し110),ミトコンドリアについては,16–18億年前にアーキア(古細菌)に共生したα-プロテオバクテリア(プロトミトコンドリアと呼ばれる)を祖先とする考え方が広く受け入れられている111).ミトコンドリアの起源の解明はヒトを含む真核生物がどのようにして生まれたのかの理解につながり,おそらく万人が興味を持つ課題と思われる.しかしながら,プロトミトコンドリア(ミトコンドリアの祖先の細菌)に,現代のα-プロテオバクテリアの中のどの細菌が最も近縁なのかについては,偏性細胞内寄生性のRickettsia目の細菌であるとする説もあり112–114),それ以外のα-プロテオバクテリア網の細菌であるという説もあり115),現在利用可能な膨大なゲノム情報があるにもかかわらず,論争が続いている.
2023年,細菌のSPTオペロンの発見をきっかけに,原始ミトコンドリアの祖先細菌の最も可能性の高い近縁種としてα-プロテオバクテリア網の海洋性細菌Iodidimonas spp.とその近縁種が新たに提案された116).Geigerらは,データベースに登録されていたα-プロテオバクテリア網の全細菌のゲノム情報を対象に,従来から用いられる細菌とミトコンドリアに共通する呼吸鎖電子伝達系のタンパク質複合体の遺伝子クラスターに加えて,マイトファジーなどいくつかのミトコンドリアの重要な生理機能を担うためには必須と想定される代謝経路を担う酵素タンパク質をコードする遺伝子の保存性に着目した.ミトコンドリアの機能発現にはミトコンドリア特異的に含まれる膜脂質の生合成は必須であるという仮定に基づき,カルジオリピンの生合成関連遺伝子とSPTオペロンを含むセラミド生合成関連遺伝子の保存性という二つの新たな基準を追加して系統解析を行った.設定した条件項目を最も多く満たした細菌は,日本の天然ガス鹹水(化石水)から分離されたヨウ素酸化菌Iodidimonas muriae117)であり,プロトミトコンドリアに最も関連する特性を持つと結論づけられた.I. muriaeが単離された環境には共生宿主であるアーキアも存在する可能性が高く,そのような環境は海底熱水泉など世界中にあることから,さらなる新発見が続くことが期待される.
引用文献References
1) Fredrickson, J.K., Balkwill, D.L., Drake, G.R., Romine, M.F., Ringelberg, D.B., & White, D.C. (1995) Aromatic-degrading Sphingomonas isolates from the deep subsurface. Appl. Environ. Microbiol., 61, 1917–1922.
2) Schubotz, F., Wakeham, S.G., Lipp, J.S., Fredricks, H.F., & Hinrichs, K.U. (2009) Detection of microbial biomass by intact polar membrane lipid analysis in the water column and surface sediments of the Black Sea. Environ. Microbiol., 11, 2720–2734.
3) Sollich, M., Yoshinaga, M.Y., Hausler, S., Price, R.E., Hinrichs, K.U., & Buhring, S.I. (2017) Heat stress dictates microbial lipid composition along a thermal gradient in marine sediments. Front. Microbiol., 8, 1550.
4) Wieland Brown, L.C., Penaranda, C., Kashyap, P.C., Williams, B.B., Clardy, J., Kronenberg, M., Sonnenburg, J.L., Comstock, L.E., Bluestone, J.A., & Fischbach, M.A. (2013) Production of alpha-galactosylceramide by a prominent member of the human gut microbiota. PLoS Biol., 11, e1001610.
5) Kato, M., Muto, Y., Tanaka-Bandoh, K., Watanabe, K., & Ueno, K. (1995) Sphingolipid composition in Bacteroides species. Anaerobe, 1, 135–139.
6) Olsen, I. & Jantzen, E. (2001) Sphingolipids in bacteria and fungi. Anaerobe, 7, 103–112.
7) Sohlenkamp, C. & Geiger, O. (2016) Bacterial membrane lipids: Diversity in structures and pathways. FEMS Microbiol. Rev., 40, 133–159.
8) Walker, A., Pfitzner, B., Harir, M., Schaubeck, M., Calasan, J., Heinzmann, S.S., Turaev, D., Rattei, T., Endesfelder, D., Castell, W.Z., et al. (2017) Sulfonolipids as novel metabolite markers of Alistipes and Odoribacter affected by high-fat diets. Sci. Rep., 7, 11047.
9) Radka, C.D., Frank, M.W., Rock, C.O., & Yao, J. (2020) Fatty acid activation and utilization by Alistipes finegoldii, a representative Bacteroidetes resident of the human gut microbiome. Mol. Microbiol., 113, 807–825.
10) Godchaux, W. 3rd & Leadbetter, E.R. (1983) Unusual sulfonolipids are characteristic of the Cytophaga-Flexibacter group. J. Bacteriol., 153, 1238–1246.
11) Godchaux, W. 3rd & Leadbetter, E.R. (1984) Sulfonolipids of gliding bacteria. Structure of the N-acylaminosulfonates. J. Biol. Chem., 259, 2982–2990.
12) Godchaux, W. III & Leadbetter, E.R. (1988) Sulfonolipids are localized in the outer membrane of the gliding bacterium Cytophaga johnsonae. Arch. Microbiol., 150, 42–47.
13) Alegado, R.A., Brown, L.W., Cao, S., Dermenjian, R.K., Zuzow, R., Fairclough, S.R., Clardy, J., & King, N. (2012) A bacterial sulfonolipid triggers multicellular development in the closest living relatives of animals. eLife, 1, e00013.
14) Batrakov, S.G., Nikitin, D.I., Sheichenko, V.I., & Ruzhitsky, A.O. (1998) A novel sulfonic-acid analogue of ceramide is the major extractable lipid of the gram-negative marine bacterium Cyclobacterium marinus WH. Biochim. Biophys. Acta Lipids Lipid Metab., 1391, 79–91.
15) Batrakov, S.G., Mosezhnyi, A.E., Ruzhitsky, A.O., Sheichenko, V.I., & Nikitin, D.I. (2000) The polar-lipid composition of the sphingolipid-producing bacterium Flectobacillus major. Biochim. Biophys. Acta Mol. Cell Biol. Lipids, 1484, 225–240.
16) Hou, L., Tian, H.Y., Wang, L., Ferris, Z.E., Wang, J., Cai, M., Older, E.A., Raja, M.R.K., Xue, D., Sun, W., et al. (2022) Identification and biosynthesis of pro-inflammatory sulfonolipids from an opportunistic pathogen Chryseobacterium gleum. ACS Chem. Biol., 17, 1197–1206.
17) Erkosar, B. & Leulier, F. (2014) Transient adult microbiota, gut homeostasis and longevity: Novel insights from the Drosophila model. FEBS Lett., 588, 4250–4257.
18) Ogawa, S., Tachimoto, H., & Kaga, T. (2010) Elevation of ceramide in Acetobacter malorum S24 by low pH stress and high temperature stress. J. Biosci. Bioeng., 109, 32–36.
19) Zhang, F., Berg, M., Dierking, K., Felix, M.A., Shapira, M., Samuel, B.S., & Schulenburg, H. (2017) Caenorhabditis elegans as a Model for Microbiome Research. Front. Microbiol., 8, 485.
20) Goodrich, J.K., Davenport, E.R., Beaumont, M., Jackson, M.A., Knight, R., Ober, C., Spector, T.D., Bell, J.T., Clark, A.G., & Ley, R.E. (2016) Genetic determinants of the gut microbiome in UK twins. Cell Host Microbe, 19, 731–743.
21) Smits, S.A., Leach, J., Sonnenburg, E.D., Gonzalez, C.G., Lichtman, J.S., Reid, G., Knight, R., Manjurano, A., Changalucha, J., Elias, J.E., et al. (2017) Seasonal cycling in the gut microbiome of the Hadza hunter-gatherers of Tanzania. Science, 357, 802–806.
22) Human Microbiome Project, C. (2012) Structure, function and diversity of the healthy human microbiome. Nature, 486, 207–214.
23) Berg, G., Eberl, L., & Hartmann, A. (2005) The rhizosphere as a reservoir for opportunistic human pathogenic bacteria. Environ. Microbiol., 7, 1673–1685.
24) Hibi, A. & Kumano, Y. (2017) Sphingobacterium spiritivorum bacteremia due to cellulitis in an elderly man with chronic obstructive pulmonary disease and congestive heart failure: A case report. J. Med. Case Rep., 11, 277.
25) Megson, Z.A., Pittenauer, E., Duda, K.A., Engel, R., Ortmayr, K., Koellensperger, G., Mach, L., Allmaier, G., Holst, O., Messner, P., et al. (2015) Inositol-phosphodihydroceramides in the periodontal pathogen tannerella forsythia: Structural analysis and incorporation of exogenous myo-inositol. Biochim. Biophys. Acta Mol. Cell Biol. Lipids, 1851, 1417–1427.
26) Rocha, F.G., Moye, Z.D., Ottenberg, G., Tang, P., Campopiano, D.J., Gibson, F.C. 3rd, & Davey, M.E. (2020) Porphyromonas gingivalis sphingolipid synthesis limits the host inflammatory response. J. Dent. Res., 99, 568–576.
27) Grundner, M., Munjakovic, H., Tori, T., Sepcic, K., Gaspersic, R., Oblak, C., Seme, K., Guella, G., Trenti, F., & Skocaj, M. (2022) Ceramide phosphoethanolamine as a possible marker of periodontal disease. Membranes (Basel), 12, 655.
28) Liu, Y., Wei, Y., Teh, T.M., Liu, D., Zhou, Y., Zhao, S., Ang, E.L., Zhao, H., & Zhang, Y. (2022) Identification and characterization of the biosynthetic pathway of the sulfonolipid capnine. Biochemistry, 61, 2861–2869.
29) Takeuchi, T., Kubota, T., Nakanishi, Y., Tsugawa, H., Suda, W., Kwon, A.T., Yazaki, J., Ikeda, K., Nemoto, S., Mochizuki, Y., et al. (2023) Gut microbial carbohydrate metabolism contributes to insulin resistance. Nature, 621, 389–395.
30) Sato, Y., Atarashi, K., Plichta, D.R., Arai, Y., Sasajima, S., Kearney, S.M., Suda, W., Takeshita, K., Sasaki, T., Okamoto, S., et al. (2021) Novel bile acid biosynthetic pathways are enriched in the microbiome of centenarians. Nature, 599, 458–464.
31) Yabuuchi, E., Yano, I., Oyaizu, H., Hashimoto, Y., Ezaki, T., & Yamamoto, H. (1990) Proposals of Sphingomonas paucimobilis gen. nov. and comb. nov., Sphingomonas parapaucimobilis sp. nov., Sphingomonas yanoikuyae sp. nov., Sphingomonas adhaesiva sp. nov., Sphingomonas capsulata comb. nov., and two genospecies of the genus Sphingomonas. Microbiol. Immunol., 34, 99–119.
32) Kawasaki, S., Moriguchi, R., Sekiya, K., Nakai, T., Ono, E., Kume, K., & Kawahara, K. (1994) The cell envelope structure of the lipopolysaccharide-lacking gram-negative bacterium Sphingomonas paucimobilis. J. Bacteriol., 176, 284–290.
33) Takeuchi, M., Hamana, K., & Hiraishi, A. (2001) Proposal of the genus Sphingomonas sensu stricto and three new genera, Sphingobium, Novosphingobium and Sphingopyxis, on the basis of phylogenetic and chemotaxonomic analyses. Int. J. Syst. Evol. Microbiol., 51, 1405–1417.
34) Kawahara, K., Seydel, U., Matsuura, M., Danbara, H., Rietschel, E.T., & Zahringer, U. (1991) Chemical structure of glycosphingolipids isolated from Sphingomonas paucimobilis. FEBS Lett., 292, 107–110.
35) Kawahara, K., Moll, H., Knirel, Y.A., Seydel, U., & Zähringer, U. (2000) Structural analysis of two glycosphingolipids from the lipopolysaccharide-lacking bacterium Sphingomonas capsulata. Eur. J. Biochem., 267, 1837–1846.
36) Nikaido, H. (2003) Molecular basis of bacterial outer membrane permeability revisited. Microbiol. Mol. Biol. Rev., 67, 593–656.
37) Kawai, F. (1999) Sphingomonads involved in the biodegradation of xenobiotic polymers. J. Ind. Microbiol. Biotechnol., 23, 400–407.
38) Momma, K., Okamoto, M., Mishima, Y., Mori, S., Hashimoto, W., & Murata, K. (2000) A novel bacterial ATP-binding cassette transporter system that allows uptake of macromolecules. J. Bacteriol., 182, 3998–4004.
39) Murata, K., Kawai, S., & Hashimoto, W. (2022) Bacteria with a mouth: Discovery and new insights into cell surface structure and macromolecule transport. Proc. Jpn. Acad., Ser. B, Phys. Biol. Sci., 98, 529–552.
40) 村田幸作,丸山如江(2015)細菌「超チャネル」と新規細胞創成—巨大分子輸送とその環境/エネルギーへの応用—.摂南大学 融合科学研究所論文集,1, 3–20.
41) 橋本渉,丸山如江,伊藤貴文,高瀬隆一,村田幸作(2016)ヘテロ多糖の輸送にかかわる細菌由来超分子の構造基盤.化学と生物,54, 885–891.
42) Yabuuchi, E., Kaneko, T., Yano, I., Moss, C.W., & Miyoshi, N. (1983) Sphingobacterium gen. nov., Sphingobacterium spiritivorum comb. nov., Sphingobacterium multivorum comb. nov., Sphingobacterium mizutae sp. nov., and Flavobacterium indologenes sp. nov.: Glucose-Nonfermenting Gram-Negative Rods in CDC Groups IIK-2 and IIb. Int. J. Syst. Evol. Microbiol., 33, 580–598.
43) Naka, T., Fujiwara, N., Yano, I., Maeda, S., Doe, M., Minamino, M., Ikeda, N., Kato, Y., Watabe, K., Kumazawa, Y., et al. (2003) Structural analysis of sphingophospholipids derived from Sphingobacterium spiritivorum, the type species of genus Sphingobacterium. Biochim. Biophys. Acta Mol. Cell Biol. Lipids, 1635, 83–92.
44) Stolp, H. & Starr, M.P. (1963) Bdellovibrio bacteriovorus gen. et sp. n., a predatory, ectoparasitic, and bacteriolytic microorganism. Antonie van Leeuwenhoek, 29, 217–248.
45) Watanabe, Y., Nakajima, M., Hoshino, T., Jayasimhulu, K., Brooks, E.E., & Kaneshiro, E.S. (2001) A novel sphingophosphonolipid head group 1-hydroxy-2-aminoethyl phosphonate in Bdellovibrio stolpii. Lipids, 36, 513–519.
46) Jayasimhulu, K., Hunt, S.M., Kaneshiro, E.S., Watanabe, Y., & Giner, J.L. (2007) Detection and identification of Bacteriovorax stolpii UKi2 sphingophosphonolipid molecular species. J. Am. Soc. Mass Spectrom., 18, 394–403.
47) Kaneda, T. (1991) Iso- and anteiso-fatty acids in bacteria: Biosynthesis, function, and taxonomic significance. Microbiol. Rev., 55, 288–302.
48) Kunsman, J.E. & Caldwell, D.R. (1974) Comparison of the sphingolipid content of rumen Bacteroides species. Appl. Microbiol., 28, 1088–1089.
49) Nierman, W.C., Feldblyum, T.V., Laub, M.T., Paulsen, I.T., Nelson, K.E., Eisen, J.A., Heidelberg, J.F., Alley, M.R., Ohta, N., Maddock, J.R., et al. (2001) Complete genome sequence of Caulobacter crescentus. Proc. Natl. Acad. Sci. USA, 98, 4136–4141.
50) Ikushiro, H., Nagami, A., Takai, T., Sawai, T., Shimeno, Y., Hori, H., Miyahara, I., Kamiya, N., & Yano, T. (2018) Heme-dependent inactivation of 5-aminolevulinate synthase from Caulobacter crescentus. Sci. Rep., 8, 14228.
51) Lasker, K., Mann, T.H., & Shapiro, L. (2016) An intracellular compass spatially coordinates cell cycle modules in Caulobacter crescentus. Curr. Opin. Microbiol., 33, 131–139.
52) Stankeviciute, G., Guan, Z., Goldfine, H., & Klein, E.A. (2019) Caulobacter crescentus adapts to phosphate starvation by synthesizing anionic glycoglycerolipids and a novel glycosphingolipid. MBio, 10, e00107-19.
53) Zik, J.J., Yoon, S.H., Guan, Z., Stankeviciute Skidmore, G., Gudoor, R.R., Davies, K.M., Deutschbauer, A.M., Goodlett, D.R., Klein, E.A., & Ryan, K.R. (2022) Caulobacter lipid A is conditionally dispensable in the absence of fur and in the presence of anionic sphingolipids. Cell Rep., 39, 110888.
54) Abbanat, D.R., Godchaux, W. 3rd, Polychroniou, G., & Leadbetter, E.R. (1985) Biosynthesis of a sulfonolipid in gliding bacteria. Biochem. Biophys. Res. Commun., 130, 873–878.
55) Abbanat, D.R., Leadbetter, E.R., Godchaux, W. III, & Escher, A. (1986) Sulphonolipids are molecular determinants of gliding motility. Nature, 324, 367–369.
56) White, R.H. (1984) Biosynthesis of the sulfonolipid 2-amino-3-hydroxy-15-methylhexadecane-1-sulfonic acid in the gliding bacterium Cytophaga johnsonae. J. Bacteriol., 159, 42–46.
57) Vences-Guzman, M.A., Pena-Miller, R., Hidalgo-Aguilar, N.A., Vences-Guzman, M.L., Guan, Z., & Sohlenkamp, C. (2021) Identification of the Flavobacterium johnsoniae cysteate-fatty acyl transferase required for capnine synthesis and for efficient gliding motility. Environ. Microbiol., 23, 2448–2460.
58) Kamiyama, T., Umino, T., Itezono, Y., Nakamura, Y., Satoh, T., & Yokose, K. (1995) Sulfobacins A and B, novel von Willebrand factor receptor antagonists. II. Structural elucidation. J. Antibiot. (Tokyo), 48, 929–936.
59) Maeda, J., Nishida, M., Takikawa, H., Yoshida, H., Azuma, T., Yoshida, M., & Mizushina, Y. (2010) Inhibitory effects of sulfobacin B on DNA polymerase and inflammation. Int. J. Mol. Med., 26, 751–758.
60) Chaudhari, P.N., Wani, K.S., Chaudhari, B.L., & Chincholkar, S.B. (2009) Characteristics of sulfobacin A from a soil isolate Chryseobacterium gleum. Appl. Biochem. Biotechnol., 158, 231–241.
61) Kinjo, Y., Wu, D., Kim, G., Xing, G.W., Poles, M.A., Ho, D.D., Tsuji, M., Kawahara, K., Wong, C.H., & Kronenberg, M. (2005) Recognition of bacterial glycosphingolipids by natural killer T cells. Nature, 434, 520–525.
62) Mattner, J., Debord, K.L., Ismail, N., Goff, R.D., Cantu, C. 3rd, Zhou, D., Saint-Mezard, P., Wang, V., Gao, Y., Yin, N., et al. (2005) Exogenous and endogenous glycolipid antigens activate NKT cells during microbial infections. Nature, 434, 525–529.
63) Mattner, J., Savage, P.B., Leung, P., Oertelt, S.S., Wang, V., Trivedi, O., Scanlon, S.T., Pendem, K., Teyton, L., Hart, J., et al. (2008) Liver autoimmunity triggered by microbial activation of natural killer T cells. Cell Host Microbe, 3, 304–315.
64) Natori, T., Koezuka, Y., & Higa, T. (1993) Agelasphins, novel α-galactosylceramides from the marine sponge Agelas mauritianus. Tetrahedron Lett., 34, 5591–5592.
65) 名取威徳(2019)海洋天然物と免疫作動性物質~α-GalCerの基礎について~.エンドトキシン・自然免疫研究,22, 21–24.
66) Moye, Z.D., Valiuskyte, K., Dewhirst, F.E., Nichols, F.C., & Davey, M.E. (2016) Synthesis of sphingolipids impacts survival of Porphyromonas gingivalis and the presentation of surface polysaccharides. Front. Microbiol., 7, 1919.
67) 岩渕和久(2018)ヒト好中球に特異的なラクトシルセラミドを介した感染免疫応答機構.Medical Mycology Journal,59, J51–J61.
68) Manes, S., del Real, G., & Martinez, A.C. (2003) Pathogens: raft hijackers. Nat. Rev. Immunol., 3, 557–568.
69) Petro, K.A., Dyer, M.A., Yowler, B.C., & Schengrund, C.L. (2006) Disruption of lipid rafts enhances activity of botulinum neurotoxin serotype A. Toxicon, 48, 1035–1045.
70) Kenworthy, A.K., Schmieder, S.S., Raghunathan, K., Tiwari, A., Wang, T., Kelly, C.V., & Lencer, W.I. (2021) Cholera toxin as a probe for membrane biology. Toxins (Basel), 13, 543.
71) Brandstaetter, H., Kendrick-Jones, J., & Buss, F. (2012) Myo1c regulates lipid raft recycling to control cell spreading, migration and Salmonella invasion. J. Cell Sci., 125, 1991–2003.
72) Lafont, F., Van Nhieu, G.T., Hanada, K., Sansonetti, P.J., & van der Goot, F.G. (2002) Initial steps of Shigella infection depend on the cholesterol/sphingolipid raft-mediated CD44–IpaB interaction. EMBO J., 21, 4449–4457.
73) Fine-Coulson, K., Reaves, B.J., Karls, R.K., & Quinn, F.D. (2012) The role of lipid raft aggregation in the infection of type II pneumocytes by Mycobacterium tuberculosis. PLoS One, 7, e45028.
74) LaBauve, A.E. & Wargo, M.J. (2014) Detection of host-derived sphingosine by Pseudomonas aeruginosa is important for survival in the murine lung. PLoS Pathog., 10, e1003889.
75) 沖野望,伊東信(2009)細菌セラミダーゼの生理機能と高次構造の解明.生化学,81, 911–916.
76) Okino, N. & Ito, M. (2007) Ceramidase enhances phospholipase C-induced hemolysis by Pseudomonas aeruginosa. J. Biol. Chem., 282, 6021–6030.
77) 沖野望,伊東信(2018)スフィンゴシンに特異的な転写制御因子を介した緑膿菌セラミダーゼの発現誘導機構とその生理的意義.生化学,90, 178–182.
78) Okino, N. & Ito, M. (2016) Molecular mechanism for sphingosine-induced Pseudomonas ceramidase expression through the transcriptional regulator SphR. Sci. Rep., 6, 38797.
79) An, D., Na, C., Bielawski, J., Hannun, Y.A., & Kasper, D.L. (2011) Membrane sphingolipids as essential molecular signals for Bacteroides survival in the intestine. Proc. Natl. Acad. Sci. USA, 108(Suppl 1), 4666–4671.
80) Olszak, T., An, D., Zeissig, S., Vera, M.P., Richter, J., Franke, A., Glickman, J.N., Siebert, R., Baron, R.M., Kasper, D.L., et al. (2012) Microbial exposure during early life has persistent effects on natural killer T cell function. Science, 336, 489–493.
81) An, D., Oh, S.F., Olszak, T., Neves, J.F., Avci, F.Y., Erturk-Hasdemir, D., Lu, X., Zeissig, S., Blumberg, R.S., & Kasper, D.L. (2014) Sphingolipids from a symbiotic microbe regulate homeostasis of host intestinal natural killer T cells. Cell, 156, 123–133.
82) Brown, E.M., Ke, X., Hitchcock, D., Jeanfavre, S., Avila-Pacheco, J., Nakata, T., Arthur, T.D., Fornelos, N., Heim, C., Franzosa, E.A., et al. (2019) Bacteroides-derived sphingolipids are critical for maintaining intestinal homeostasis and symbiosis. Cell Host Microbe, 25, 668–680.e7.
83) Ford, D.A. (2021) A BOSSS platform: Using functionalized lipids and click chemistry for new discoveries in lipid research. J. Lipid Res., 62, 100025.
84) Lee, M.T., Le, H.H., & Johnson, E.L. (2021) Dietary sphinganine is selectively assimilated by members of the mammalian gut microbiome. J. Lipid Res., 62, 100034.
85) Johnson, E.L., Heaver, S.L., Waters, J.L., Kim, B.I., Bretin, A., Goodman, A.L., Gewirtz, A.T., Worgall, T.S., & Ley, R.E. (2020) Sphingolipids produced by gut bacteria enter host metabolic pathways impacting ceramide levels. Nat. Commun., 11, 2471.
86) Le, H.H., Lee, M.T., Besler, K.R., & Johnson, E.L. (2022) Host hepatic metabolism is modulated by gut microbiota-derived sphingolipids. Cell Host Microbe, 30, 798–808.e7.
87) Duan, J. & Merrill, A.H. Jr. (2015) 1-Deoxysphingolipids encountered exogenously and made de novo: Dangerous mysteries inside an enigma. J. Biol. Chem., 290, 15380–15389.
88) Penno, A., Reilly, M.M., Houlden, H., Laura, M., Rentsch, K., Niederkofler, V., Stoeckli, E.T., Nicholson, G., Eichler, F., Brown, R.H. Jr., et al. (2010) Hereditary sensory neuropathy type 1 is caused by the accumulation of two neurotoxic sphingolipids. J. Biol. Chem., 285, 11178–11187.
89) Ryan, E., Gonzalez Pastor, B., Gethings, L.A., Clarke, D.J., & Joyce, S.A. (2023) Lipidomic analysis reveals differences in Bacteroides species driven largely by plasmalogens, glycerophosphoinositols and certain sphingolipids. Metabolites, 13, 360.
90) Ikushiro, H., Hayashi, H., & Kagamiyama, H. (2001) A water-soluble homodimeric serine palmitoyltransferase from Sphingomonas paucimobilis EY2395T strain. Purification, characterization, cloning, and overproduction. J. Biol. Chem., 276, 18249–18256.
91) Ikushiro, H., Islam, M.M., Tojo, H., & Hayashi, H. (2007) Molecular characterization of membrane-associated soluble serine palmitoyltransferases from Sphingobacterium multivorum and Bdellovibrio stolpii. J. Bacteriol., 189, 5749–5761.
92) Ikushiro, H. & Hayashi, H. (2011) Mechanistic enzymology of serine palmitoyltransferase. Biochim. Biophys. Acta. Proteins Proteom., 1814, 1474–1480.
93) Ikushiro, H., Hayashi, H., & Kagamiyama, H. (2004) Reactions of serine palmitoyltransferase with serine and molecular mechanisms of the actions of serine derivatives as inhibitors. Biochemistry, 43, 1082–1092.
94) Ikushiro, H., Fujii, S., Shiraiwa, Y., & Hayashi, H. (2008) Acceleration of the substrate Calpha deprotonation by an analogue of the second substrate palmitoyl-CoA in Serine Palmitoyltransferase. J. Biol. Chem., 283, 7542–7553.
95) Ikushiro, H., Islam, M.M., Okamoto, A., Hoseki, J., Murakawa, T., Fujii, S., Miyahara, I., & Hayashi, H. (2009) Structural insights into the enzymatic mechanism of serine palmitoyltransferase from Sphingobacterium multivorum. J. Biochem., 146, 549–562.
96) Shiraiwa, Y., Ikushiro, H., & Hayashi, H. (2009) Multifunctional role of His159in the catalytic reaction of serine palmitoyltransferase. J. Biol. Chem., 284, 15487–15495.
97) Lowther, J., Charmier, G., Raman, M.C., Ikushiro, H., Hayashi, H., & Campopiano, D.J. (2011) Role of a conserved arginine residue during catalysis in serine palmitoyltransferase. FEBS Lett., 585, 1729–1734.
98) Ikushiro, H., Takahashi, A., Murakami, T., Katayama, A., Sawai, T., Goto, H., Miyahara, I., Kamiya, N., & Yano, T. (2022) Crystal structure of Sphingobacterium multivorum serine palmitoyltransferase complexed with tris(hydroxymethyl)aminomethane. Acta Crystallogr. F Struct. Biol. Commun., 78, 408–415.
99) Ikushiro, H., Murakami, T., Takahashi, A., Katayama, A., Sawai, T., Goto, H., Koolath, S., Murai, Y., Monde, K., Miyahara, I., et al. (2023) Structural insights into the substrate recognition of serine palmitoyltransferase from Sphingobacterium multivorum. J. Biol. Chem., 299, 104684.
100) Ikushiro, H., Honda, T., Murai, Y., Murakami, T., Takahashi, A., Sawai, T., Goto, H., Ikushiro, S.I., Miyahara, I., Hirabayashi, Y., et al. (2024) Racemization of the substrate and product by serine palmitoyltransferase from Sphingobacterium multivorum yields two enantiomers of the product from D-serine. J. Biol. Chem., 300, 105728.
101) 生城浩子,林秀行(2011)細菌由来セリンパルミトイル転移酵素の立体構造と触媒機構の解析.生化学,83, 105–110.
102) 生城浩子(2015)セリンパルミトイル転移酵素の反応制御機構—変異酵素の副反応から明らかになった立体化学的反応制御.生化学,87, 298–307.
103) Stankeviciute, G., Tang, P., Ashley, B., Chamberlain, J.D., Hansen, M.E.B., Coleman, A., D’Emilia, R., Fu, L., Mohan, E.C., Nguyen, H., et al. (2022) Convergent evolution of bacterial ceramide synthesis. Nat. Chem. Biol., 18, 305–312.
104) Heaver, S.L., Le, H.H., Tang, P., Baslé, A., Mirretta Barone, C., Vu, D.L., Waters, J.L., Marles-Wright, J., Johnson, E.L., Campopiano, D.J., et al. (2022) Characterization of inositol lipid metabolism in gut-associated Bacteroidetes. Nat. Microbiol., 7, 986–1000.
105) Padilla-Gomez, J., Olea-Ozuna, R.J., Contreras-Martinez, S., Morales-Tarre, O., Garcia-Soriano, D.A., Sahonero-Canavesi, D.X., Poggio, S., Encarnacion-Guevara, S., Lopez-Lara, I.M., & Geiger, O. (2022) Specialized acyl carrier protein used by serine palmitoyltransferase to synthesize sphingolipids in Rhodobacteria. Front. Microbiol., 13, 961041.
106) Gault, C.R., Obeid, L.M., & Hannun, Y.A. (2010) An overview of sphingolipid metabolism: From synthesis to breakdown. Adv. Exp. Med. Biol., 688, 1–23.
107) Lee, M.T., Le, H.H., Besler, K.R., & Johnson, E.L. (2022) Identification and characterization of 3-ketosphinganine reductase activity encoded at the BT_0972 locus in Bacteroides thetaiotaomicron. J. Lipid Res., 63, 100236.
108) Okino, N., Li, M., Qu, Q., Nakagawa, T., Hayashi, Y., Matsumoto, M., Ishibashi, Y., & Ito, M. (2020) Two bacterial glycosphingolipid synthases responsible for the synthesis of glucuronosylceramide and alpha-galactosylceramide. J. Biol. Chem., 295, 10709–10725.
109) Radka, C.D., Miller, D.J., Frank, M.W., & Rock, C.O. (2022) Biochemical characterization of the first step in sulfonolipid biosynthesis in Alistipes finegoldii. J. Biol. Chem., 298, 102195.
110) Sagan, L. (1967) On the origin of mitosing cells. J. Theor. Biol., 14, 255–274.
111) Imachi, H., Nobu, M.K., Nakahara, N., Morono, Y., Ogawara, M., Takaki, Y., Takano, Y., Uematsu, K., Ikuta, T., Ito, M., et al. (2020) Isolation of an archaeon at the prokaryote-eukaryote interface. Nature, 577, 519–525.
112) Wang, Z. & Wu, M. (2014) Phylogenomic reconstruction indicates mitochondrial ancestor was an energy parasite. PLoS One, 9, e110685.
113) Fan, L., Wu, D., Goremykin, V., Xiao, J., Xu, Y., Garg, S., Zhang, C., Martin, W.F., & Zhu, R. (2020) Phylogenetic analyses with systematic taxon sampling show that mitochondria branch within Alphaproteobacteria. Nat. Ecol. Evol., 4, 1213–1219.
114) Ferla, M.P., Thrash, J.C., Giovannoni, S.J., & Patrick, W.M. (2013) New rRNA gene-based phylogenies of the Alphaproteobacteria provide perspective on major groups, mitochondrial ancestry and phylogenetic instability. PLoS One, 8, e83383.
115) Martijn, J., Vosseberg, J., Guy, L., Offre, P., & Ettema, T.J.G. (2018) Deep mitochondrial origin outside the sampled alphaproteobacteria. Nature, 557, 101–105.
116) Geiger, O., Sanchez-Flores, A., Padilla-Gomez, J., & Degli Esposti, M. (2023) Multiple approaches of cellular metabolism define the bacterial ancestry of mitochondria. Sci. Adv., 9, eadh0066.
117) Iino, T., Ohkuma, M., Kamagata, Y., & Amachi, S. (2016) Iodidimonas muriae gen. nov., sp. nov., an aerobic iodide-oxidizing bacterium isolated from brine of a natural gas and iodine recovery facility, and proposals of Iodidimonadaceae fam. nov., Iodidimonadales ord. nov., Emcibacteraceae fam. nov. and Emcibacterales ord. nov. Int. J. Syst. Evol. Microbiol., 66, 5016–5022.
118) von Gerichten, J., Schlosser, K., Lamprecht, D., Morace, I., Eckhardt, M., Wachten, D., Jennemann, R., Grone, H.J., Mack, M., & Sandhoff, R. (2017) Diastereomer-specific quantification of bioactive hexosylceramides from bacteria and mammals. J. Lipid Res., 58, 1247–1258.