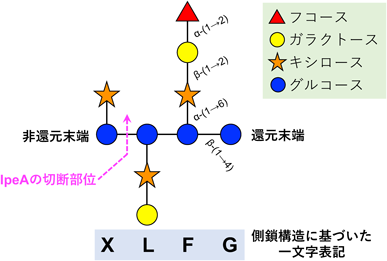
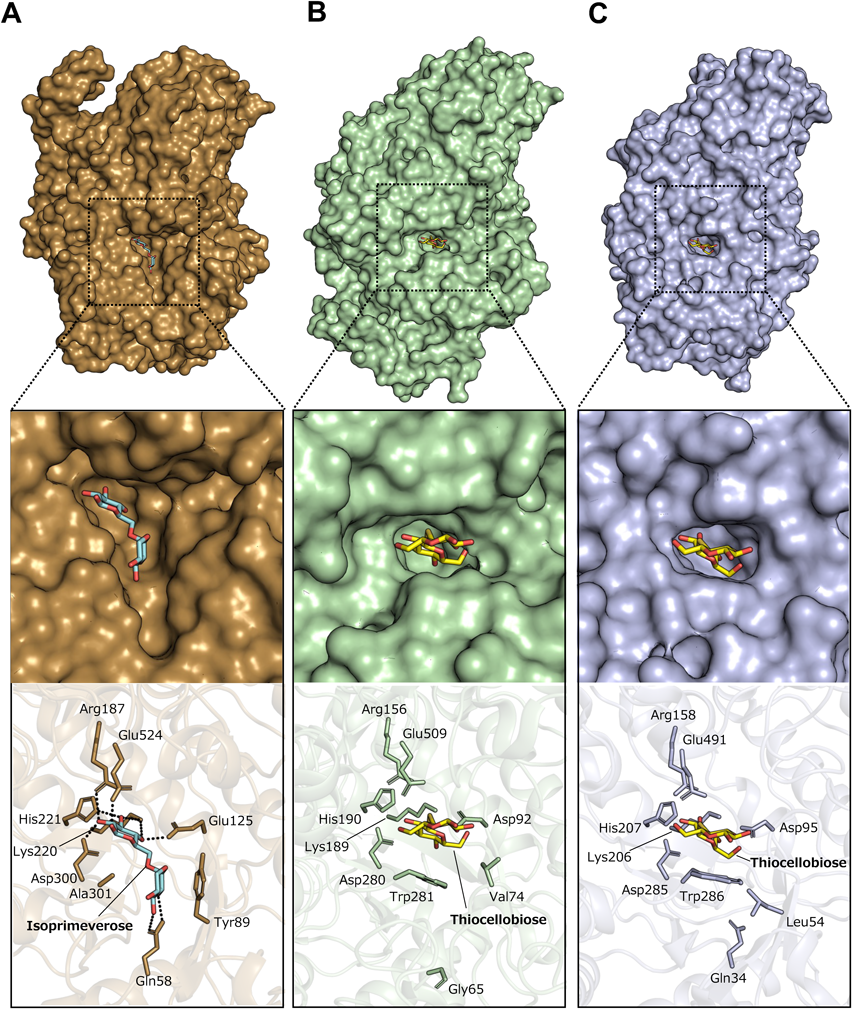
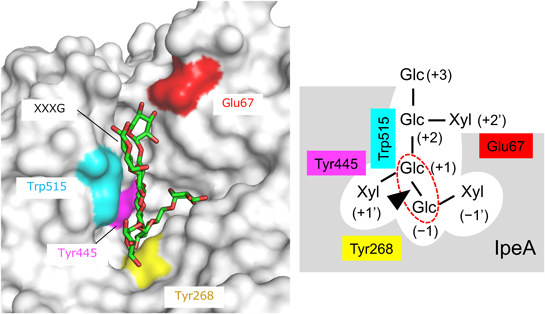
黄麹菌由来キシログルカン分解酵素の機能と構造Function and structure of xyloglucan degrading enzyme
1 香川大学農学部応用生物科学科Department of Applied Biological Science, Faculty of Agriculture, Kagawa University ◇ 〒761–0795 香川県木田郡三木町池戸2393 ◇ 2393 Ikenobe, Miki, Kagawa 761–0795, Japan
2 産業技術総合研究所機能化学研究部門Research Institute for Sustainable Chemistry, National Institute of Advanced Industrial Science and Technology (AIST) ◇ 〒739–0046 広島県東広島市鏡山3–11–32 ◇ 3–11–32 Kagamiyama, Higashi-Hiroshima, Hiroshima 739–0046, Japan