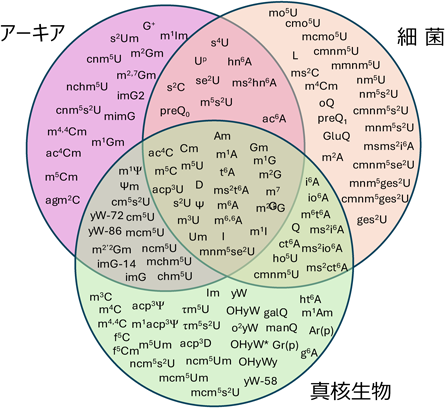
RNAには多様な転写後修飾が含まれており,これらがさまざまな生命現象と密接に関わっていることが知られている.mRNAワクチンの登場により,脚光を浴びているように,RNA修飾の担う役割としては,自然免疫からの回避,細胞内局在の決定,立体構造の安定化,RNA結合タンパク質との相互作用,遺伝情報の修飾と解読などが知られているが,その機能と生合成過程には未解明な部分が多く残されている.近年,真核生物のmRNAやnon-coding RNAに大量のイノシン(I)やN6-メチルアデノシン(m6A)をはじめとするいくつかのRNA修飾が見いだされ,遺伝子発現が転写後修飾によって制御されるエピトランスクリプトミクスという概念が生まれている1).同じ核酸であるDNAにはわずか数種類の修飾塩基が見つかっているにすぎないが,RNAには現在までに約150種類もの修飾が確認されている.これらのRNA修飾は,塩基やリボースのメチル化,アセチル化,水酸化,脱水環化,硫化,セレノ化,還元,異性化,アミノ酸や糖の付加など,多様な化学的バリエーションに富んでいる(図1)2).RNA修飾の多様性は,RNAが新たな機能を担うために進化の過程で獲得されたと考えられる.実際,種を超えて高度に保存されたRNA修飾よりも,各生物界で独自に獲得されたRNA修飾の種類が多いことがわかる(図1).近年も筆者らの研究室を含む複数の研究グループから,新規のRNA修飾が続々と報告されており3, 4),RNA修飾のケミカルスペースはさらに拡大すると予想される5).筆者らは,ミトコンドリア脳筋症の代表病型であるMELASやMERRFが,tRNAの修飾欠損で生じることを明らかにし,RNA修飾の欠損や異常が疾患の原因になる“RNA修飾病”という概念を提唱している5–7).実際,RNA修飾酵素の変異が,糖尿病や精神神経疾患などさまざまな疾患の原因であることが明らかになり,RNA修飾と疾患との関連が注目されている8, 9).
最近,米国科学・工学・医学アカデミー(NASEM)が,RNA修飾が生命現象に与える影響の重要性を認識し,医療や農業を含む生命科学のさまざまな分野に貢献するため,RNA修飾をシーケンスするためのロードマップを作成し,大規模な研究報告書を発表した10).この報告書は,エピトランスクリプトームシーケンス技術の発展を目指し,国家プロジェクト,産業界,第三セクター,国際的な協力体制を構築することで,ヒトゲノムプロジェクトに匹敵するような巨大な研究ムーブメントを創出することを目指している.エピトランスクリプトミクス研究は新しい創薬や治療の標的としても注目されており,この分野に特化したプロジェクト研究やベンチャー企業も設立され,産業界からも注目されている.
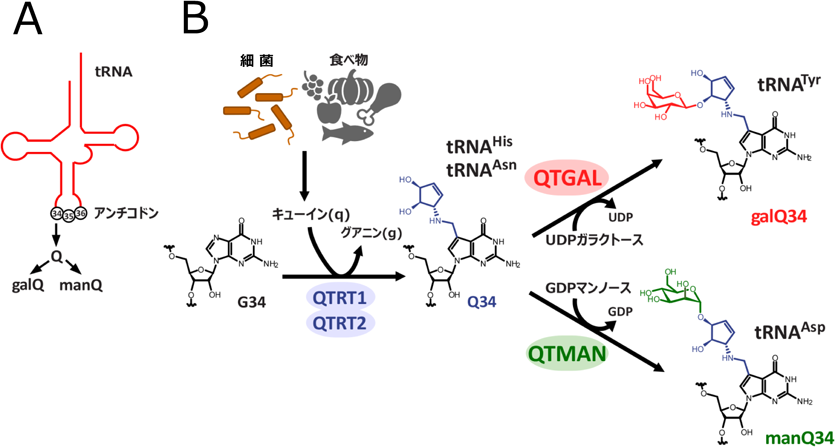
RNAの中でも,特にtRNAは最も複雑に修飾されたRNA分子であり,タンパク質合成において重要な役割を担っている.これまでに見つかっているRNA修飾の約8割はtRNAから見つかっている.tRNAのアンチコドン領域(32~38位)(図2A)にはさまざまな修飾が存在し,これらはtRNAがmRNA上のコドンを正確に読み取る能力を付与し,正確かつ効率的なタンパク質合成を可能にする5, 11).キューオシン(Q)は7-デアザグアノシン骨格とシクロペンテン環を持つグアノシンの誘導体で,細菌からヒトに至る多くの生物にみられるユニークなtRNA修飾である(図2B).細菌はQの塩基部分であるキューイン(q)を生合成するが,ヒトや脊椎動物はqを自前で生合成することができず,腸内細菌や食物から摂取したqを栄養素として腸管から体内へと取り込み,tRNAのQ修飾に利用する12)(図2B).ヒトでは,QTRT1とQTRT2の複合体がトランスグリコシラーゼとして機能し,tRNAのアンチコドン1字目のグアノシンの塩基部分をqにつけ替えることでQ修飾を形成する.ヒトや脊椎動物では,4種類の細胞質tRNAがQ修飾を持つが,チロシン(Tyr)に対応するtRNATyrはQにガラクトースが付加されたガラクトシルキューオシン(galQ)に変換され,アスパラギン酸(Asp)に対応するtRNAAspではQにマンノースが付加されたマンノシルキューオシン(manQ)に変換される(図2B).galQやmanQは1976年に西村暹博士らによって発見され13),tRNA修飾の中で糖が付与された稀有な修飾体として注目されているが,その生合成や機能は約半世紀もの間謎に包まれていた.
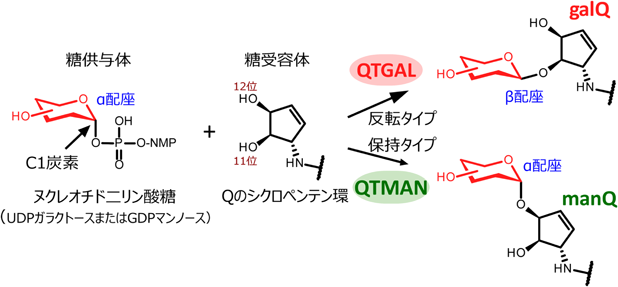
筆者らの研究グループは,ラット肝臓の抽出液から複数のカラムクロマトグラフィーを用いた生化学的手法により,tRNATyrのQ修飾にガラクトースを転移する酵素(B3GNTL1)を同定し,キューオシンtRNAガラクトース転移酵素(QTGAL)(図2B)と命名した14).さらに,ブタ肝臓の抽出液を分画し,tRNAAspのQ修飾にマンノースを転移する酵素(GTDC1)を同定し,キューオシンtRNAマンノース転移酵素(QTMAN)(図2B)と命名した14).QTGALとQTMANはそれぞれUDPガラクトースおよびGDPマンノースを基質として,galQとmanQを生合成する.糖転移反応では,糖のC1炭素の立体配座がα配座からβ配座へと反転する場合と,α配座のまま保持される場合がある(図3).QTGALとQTMANは糖転移酵素のスーパーファミリーに属し,もともと機能未知な糖転移酵素として登録されていた.QTGALはGT-Aフォールドを持つGT2ファミリーに属し,Qのシクロペンテン環の11位の水酸基にUDPガラクトースのC1炭素をSN2型の求核置換攻撃させることで,C1炭素の立体配座がβ配座へと反転し,galQを生成する(図3).一方で,QTMANはGT-Bフォールドを持つGT4ファミリーに属し,保持型の糖転移反応を触媒することで,Qのシクロペンテン環の12位の水酸基にα配座のままマンノースが転移し,manQを生成する(図3).QTMANによる酵素反応の速度論的解析から,GDPマンノースに対するミカエリス–メンテン定数(Km値)が69 µMと比較的高く,細胞内のGDPマンノース濃度(0.9~11 µM)を考慮すると,QTMANによるmanQ生成反応は細胞内のGDPマンノース濃度で調節されている可能性が示唆された.
さらに,筆者らはQTGALおよびQTMANの遺伝子を破壊したヒトの培養細胞,QTGAL KOおよびQTMAN KOを作製し,これらのKO細胞から精製したtRNAにはそれぞれgalQおよびmanQが完全に消失していることを確認した.
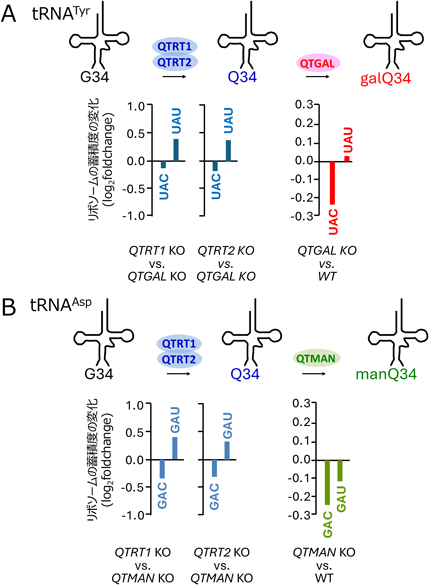
筆者らは糖付加Q修飾の機能を調べるため,理化学研究所の岩崎信太郎研究室と共同で,リボソームプロファイリングを行った.QTGAL KO細胞では,tRNATyrが読むコドンのうちUACコドンの翻訳速度が速くなり(図4A),QTMAN KO細胞ではtRNAAspが読むGACとGAUコドンの翻訳速度が速くなることが判明した(図4B).Q修飾は3字目がUのコドンの翻訳速度を速める働きがある(図4A, B)ことから,Qの糖付加修飾は速すぎる翻訳速度を制御する役割があることが判明した.一般的にtRNA修飾は翻訳速度を促進する働きがあるが,Qの糖付加は翻訳速度を抑制することが判明した初めてのtRNA修飾であるといえるであろう.また,tRNATyrは潜在的に終止コドンであるUAGとUAAを読み飛ばすことが知られているが,galQのガラクトース付加修飾には終止コドンの読み飛ばしを防止する役割があることも判明した.
一般に,tRNA修飾は適切な翻訳速度の維持に重要な役割を担い,tRNA修飾が欠損すると,新生タンパク質のフォールディングに異常が生じることが知られている.各KO細胞にレポーター遺伝子を導入し,タンパク質の凝集度を計測したところ,いずれのKO細胞においても凝集したタンパク質が蓄積したことから,糖付加Q修飾は適切な翻訳速度を調節することで,プロテオスタシスの維持に重要な役割を担っていることが判明した.
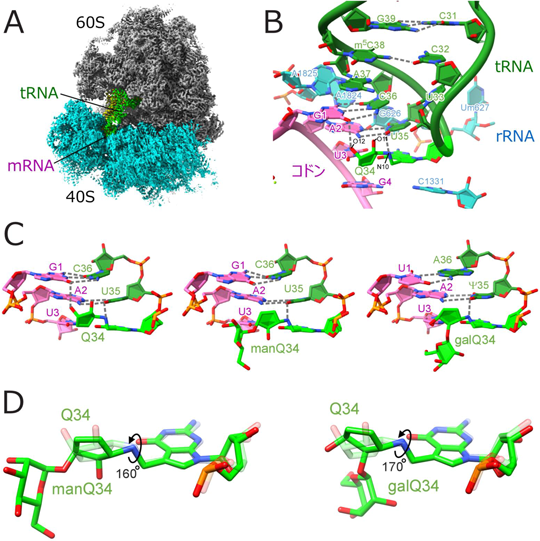
筆者らは,理化学研究所の白水美香子研究室の支援の下,リボソームとtRNA複合体の構造解析を行った.ヒト細胞から精製したリボソームに修飾状態の異なるtRNAを結合させて,クライオ電子顕微鏡を用いた構造解析を行った(図5A, B).その結果,Q修飾のシクロペンテン環は水素結合を介してコドン–アンチコドン対合が形成する二重らせんの主溝に結合し,主溝結合基(major groove binder)として機能することで,コドン認識の効率を上げていることが明らかとなった(図5C).特にこの効果はコドン3字目がUを持つ,UAUやGAUコドンの際に効果が高いことも判明した.一方で,Qの糖付加は,シクロペンテン環を回転させることでこの効果を弱める働きがある(図5D)ことがわかり,糖付加Q修飾による翻訳調節の分子基盤が明らかとなった.
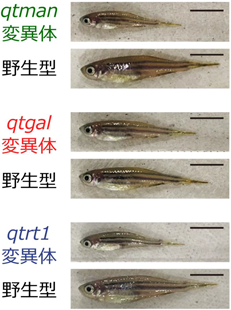
糖付加Q修飾による生理機能を探求するために,京都産業大学の三嶋雄一郎研究室との共同研究により,ゼブラフィッシュを用いたqtgalおよびqtmanのノックアウト(KO)系統の解析を行った.これらの変異体は正常に胚発生したが,野生型と比較して生後の成長が顕著に遅く,体長が短いという表現型を示した(図6).さらにqtgal KO系統では,eIF2αのリン酸化が顕著に亢進していることが判明した.これは,適切な翻訳速度が乱れたことにより,タンパク質のフォールディング異常を検知し,統合的ストレス応答が作動していることを示唆している.以上の結果から,糖付加Q修飾はゼブラフィッシュの健全な生育に必要であることが判明した.
Q修飾とがんとの関係について多くの研究報告がある12, 15).悪性腫瘍やがん細胞においてQ修飾が低下することが報告されている16–18).またQ修飾の低下は各がんのステージと強い相関がある19).さらに,QTRT1の欠損は,がん細胞の増殖を抑制することが知られている20).TCGAデータベース21)によると多くの腫瘍においてQTRT1, QTRT2, QTMAN, QTGALの発現量の増加がみられる.QTRT1が高発現すると腎臓がんの予後が悪く,子宮頚がんでは逆の傾向がある.QTRT2については,発現量が多いほど脳腫瘍の予後が悪い.QTGALが高発現すると腎臓がんの予後が悪くなる.QTMANは発現量が低いと脳腫瘍の予後が悪いが,発現量が高いと肝臓がんの予後が悪い.がん以外の疾患に関しても,QTMANの機能障害がグリシン代謝異常を介してNMDA受容体の過剰活性化を引き起こし,神経発達障害の原因になる可能性が示唆されている22).また,神経前駆細胞(NPC)とニューロンでGTDC1(QTMAN)を欠損させると,神経変性疾患の患者から作製したiPS細胞と同様の表現型を示した23).
引用文献References
1) Frye, M., Jaffrey, S.R., Pan, T., Rechavi, G., & Suzuki, T. (2016) RNA modifications: What have we learned and where are we headed? Nat. Rev. Genet., 17, 365–372.
2) Cappannini, A., Ray, A., Purta, E., Mukherjee, S., Boccaletto, P., Moafinejad, S.N., Lechner, A., Barchet, C., Klaholz, B.P., Stefaniak, F., et al. (2024) MODOMICS: A database of RNA modifications and related information. 2023 update. Nucleic Acids Res., 52(D1), D239–D244.
3) Miyauchi, K., Kimura, S., Akiyama, N., Inoue, K., Ishiguro, K., Vu, T., Srisuknimit, V., Koyama, K., Hayashi, G., Soma, A., et al. (2024) A tRNA modification with aminovaleramide facilitates AUA decoding in protein synthesis. Nat. Chem. Biol. (in press).
4) Ohira, T., Minowa, K., Sugiyama, K., Yamashita, S., Sakaguchi, Y., Miyauchi, K., Noguchi, R., Kaneko, A., Orita, I., Fukui, T., et al. (2022) Reversible RNA phosphorylation stabilizes tRNA for cellular thermotolerance. Nature, 605, 372–379.
5) Suzuki, T. (2021) The expanding world of tRNA modifications and their disease relevance. Nat. Rev. Mol. Cell Biol., 22, 375–392.
6) Asano, K., Suzuki, T., Saito, A., Wei, F.Y., Ikeuchi, Y., Numata, T., Tanaka, R., Yamane, Y., Yamamoto, T., Goto, T., et al. (2018) Metabolic and chemical regulation of tRNA modification associated with taurine deficiency and human disease. Nucleic Acids Res., 46, 1565–1583.
7) Chujo, T. & Tomizawa, K. (2021) Human transfer RNA modopathies: Diseases caused by aberrations in transfer RNA modifications. FEBS J., 288, 7096–7122.
8) Delaunay, S., Helm, M., & Frye, M. (2024) RNA modifications in physiology and disease: Towards clinical applications. Nat. Rev. Genet., 25, 104–122.
9) Orellana, E.A., Siegal, E., & Gregory, R.I. (2022) tRNA dysregulation and disease. Nat. Rev. Genet., 23, 651–664.
10) National Academies of Sciences E, Medicine (2024) Charting a Future for Sequencing RNA and Its Modifications: A New Era for Biology and Medicine. The National Academies Press, Washington, DC.
11) Suzuki, T. (2005) Biosynthesis and function of tRNA wobble modifications. In Fine-Tuning of RNA Functions by Modification and Editing (Grosjean, H. ed.), pp. 23–69, Springer-Verlag Berlin and Heidelberg.
12) Suzuki, T., Ogizawa, A., Ishiguro, K., & Nagao, A. (2024) Physiological and pathological relevance of tRNA queuosine modification and its glycosylated derivatives. Cell Chem. Biol. (in press).
13) Kasai, H., Nakanishi, K., Macfarlane, R.D., Torgerson, D.F., Ohashi, Z., McCloskey, J.A., Gross, H.J., & Nishimura, S. (1976) Letter: The structure of Q* nucleoside isolated from rabbit liver transfer ribonucleic acid. J. Am. Chem. Soc., 98, 5044–5046.
14) Zhao, X., Ma, D., Ishiguro, K., Saito, H., Akichika, S., Matsuzawa, I., Mito, M., Irie, T., Ishibashi, K., Wakabayashi, K., et al. (2023) Glycosylated queuosines in tRNAs optimize translational rate and post-embryonic growth. Cell, 186, 5517–5535 e5524.
15) Fergus, C., Barnes, D., Alqasem, M.A., & Kelly, V.P. (2015) The queuine micronutrient: Charting a course from microbe to man. Nutrients, 7, 2897–2929.
16) Baranowski, W., Dirheimer, G., Jakowicki, J.A., & Keith, G. (1994) Deficiency of queuine, a highly modified purine base, in transfer RNAs from primary and metastatic ovarian malignant tumors in women. Cancer Res., 54, 4468–4471.
17) Gündüz, U., Elliott, M.S., Seubert, P.H., Houghton, J.A., Houghton, P.J., Trewyn, R.W., & Katze, J.R. (1992) Absence of tRNA-guanine transglycosylase in a human colon adenocarcinoma cell line. Biochim. Biophys. Acta Mol. Basis Dis., 1139, 229–238.
18) Huang, B.S., Wu, R.T., & Chien, K.Y. (1992) Relationship of the queuine content of transfer ribonucleic acids to histopathological grading and survival in human lung cancer. Cancer Res., 52, 4696–4700.
19) Emmerich, B., Zubrod, E., Weber, H., Maubach, P.A., Kersten, H., & Kersten, W. (1985) Relationship of queuine-lacking transfer RNA to the grade of malignancy in human leukemias and lymphomas. Cancer Res., 45, 4308–4314.
20) Zhang, J., Lu, R., Zhang, Y., Matuszek, Z., Zhang, W., Xia, Y., Pan, T., & Sun, J. (2020) tRNA queuosine modification enzyme modulates the growth and microbiome recruitment to breast tumors. Cancers (Basel), 12, 628.
21) Blum, A., Wang, P., & Zenklusen, J.C. (2018) SnapShot: TCGA-analyzed tumors. Cell, 173, 530.
22) Errichiello, E., Lecca, M., Vantaggiato, C., Motta, Z., Zanotta, N., Zucca, C., Bertuzzo, S., Piubelli, L., Pollegioni, L., & Bonaglia, M.C. (2024) Further evidence supporting the role of GTDC1 in glycine metabolism and neurodevelopmental disorders. Eur. J. Hum. Genet., 32, 920–927.
23) Aksoy, I., Utami, K.H., Winata, C.L., Hillmer, A.M., Rouam, S.L., Briault, S., Davila, S., Stanton, L.W., & Cacheux, V. (2017) Personalized genome sequencing coupled with iPSC technology identifies GTDC1 as a gene involved in neurodevelopmental disorders. Hum. Mol. Genet., 26, 367–382.