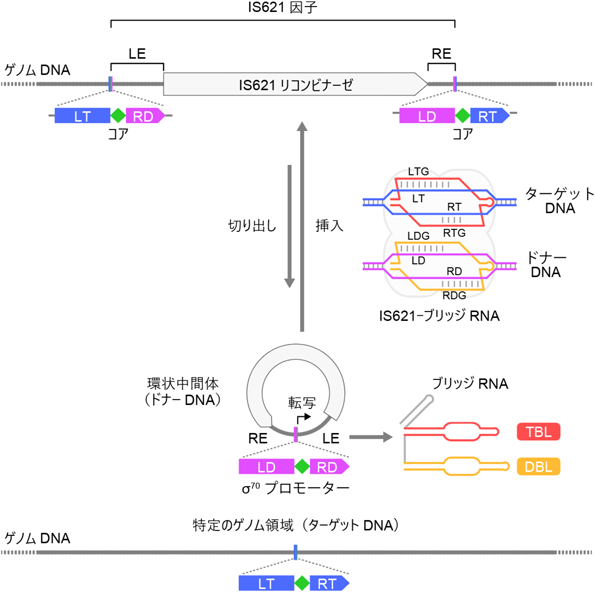
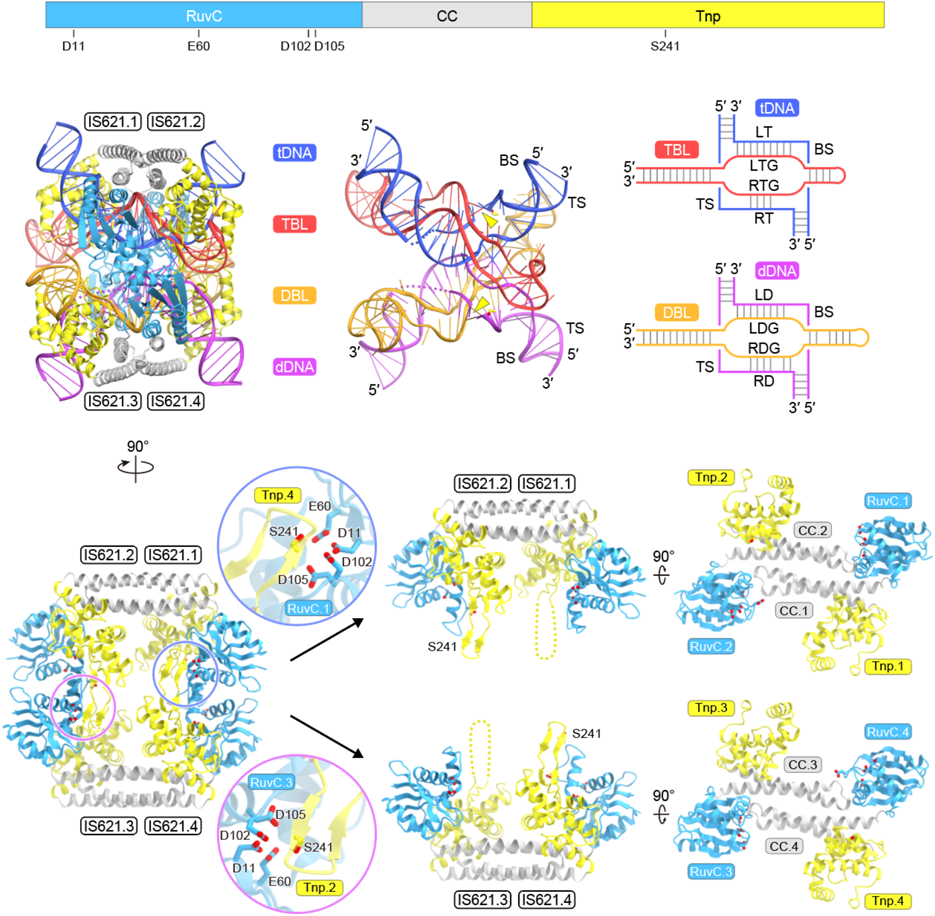
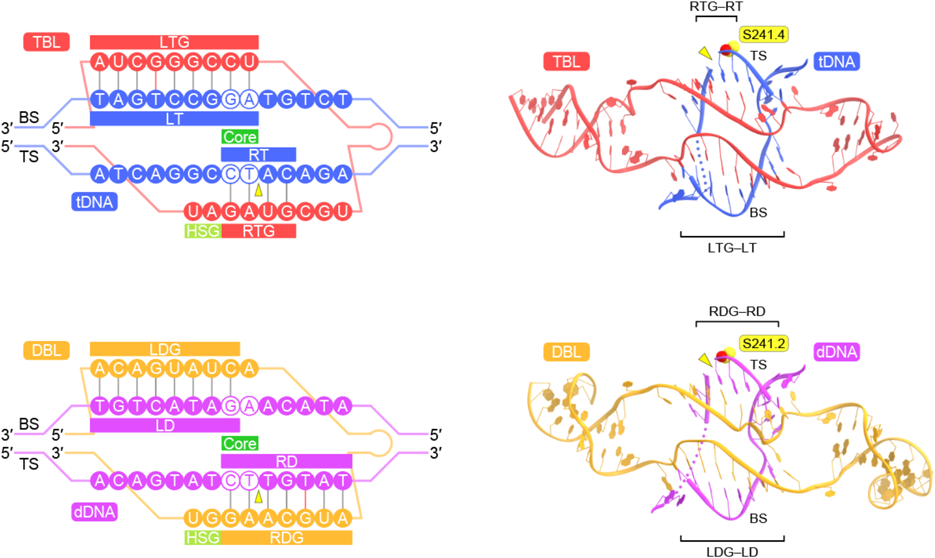
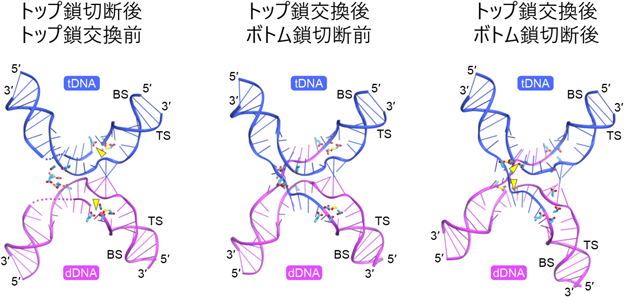
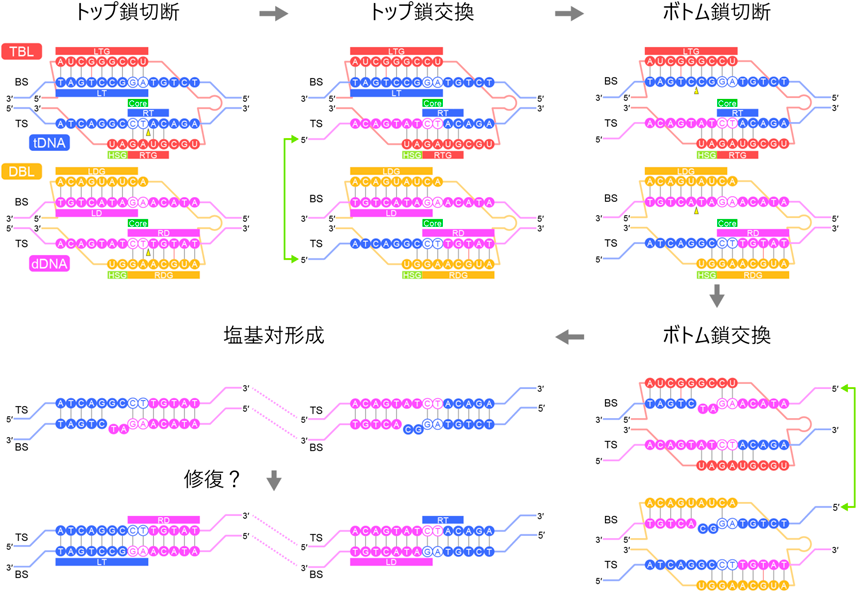
IS110ファミリーリコンビナーゼはブリッジRNA依存性DNA組換え反応を触媒するIS110 family recombinases catalyze bridge RNA-guided DNA recombination
1 東京大学先端科学技術研究センターStructural Biology Division, Research Center for Advanced Science and Technology, The University of Tokyo ◇ 〒153–8904 東京都目黒区駒場4丁目6番1号 ◇ 4–6–1 Komaba, Meguro-ku, Tokyo 153–8904, Japan
2 東京大学大学院工学系研究科化学生命工学専攻Department of Chemistry and Biotechnology, Graduate School of Engineering, The University of Tokyo ◇ 〒153–8904 東京都目黒区駒場4丁目6番1号 ◇ 4–6–1 Komaba, Meguro-ku, Tokyo 153–8904, Japan