1)停滞したリボソーム由来の新生ペプチド鎖を分解する品質管理機構
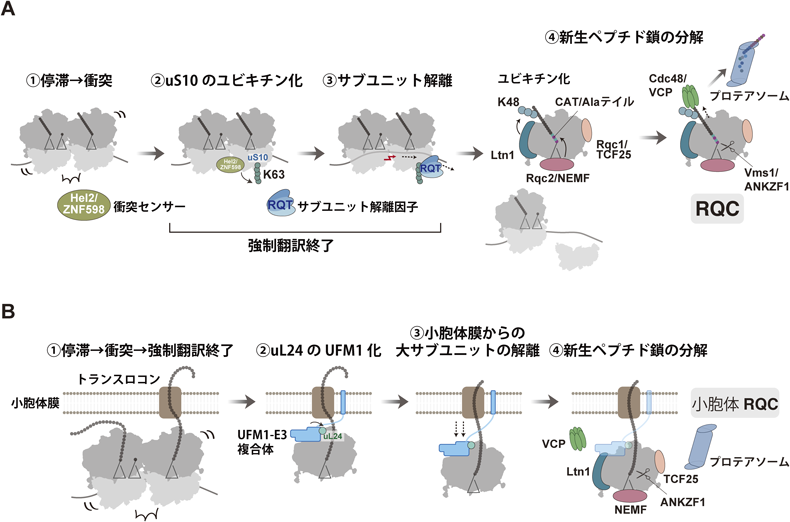
翻訳伸長中のリボソーム停滞はタンパク質合成効率の低下に加え,毒性を示しうる不完全なタンパク質の蓄積を引き起こし,タンパク質恒常性が破綻する原因となる.このような異常翻訳産物を排除する品質管理機構はribosome-associated quality control(RQC)と呼ばれている1).RQCは出芽酵母からヒトまで保存された普遍的な品質管理機構である.その初期段階ではE3ユビキチンリガーゼHel2(ヒトではZNF598)が停滞したリボソームを認識し,uS10を含む複数のリボソーム小サブユニットタンパク質をユビキチン化する(図1A①,②)2–8).uS10に付加されたユビキチン鎖に,Slh1, Cue3, Rqt4からなるribosome-associated quality control trigger(RQT;ヒトではASCC3, ASCC2, TRIP4からなるASC-1)複合体がリクルートされ,Slh1/ASCC3のATPアーゼ活性依存的にリボソームが大・小サブユニットへ解離する(図1A③)9–12).解離後の大サブユニットにLtn1, Rqc1, Rqc2, Cdc48(ヒトではLtn1, TCF25, NEMF, VCP)からなるRQC複合体が結合する13–17).E3ユビキチンリガーゼであるLtn1が新生ペプチド鎖上のリシン残基をユビキチン化し,またRqc2/NEMFは新生ペプチド鎖のC末端にアラニンとトレオニンからなるC-terminal alanine threonine(CAT)テイルもしくはアラニンのみからなるアラニン(Ala)テイルを付加する15, 18–22).最後に翻訳終結因子eRF1のホモログであるVms1/ANKZF1がペプチジルtRNAのCCA末端を切断し23–27),ATPアーゼであるCdc48/VCPとその補因子Ufd1/UFD1およびNpl4/NPLOC4からなる複合体が新生ペプチド鎖をリボソームトンネルから引き抜き,プロテアソームへ輸送する(図1A④)14, 28, 29).
2)Hel2による衝突リボソームの認識とユビキチン化
Hel2はuS10, uS3, eS7など,リボソーム小サブユニット上の複数のタンパク質をユビキチン化するが,RQCの引き金となるのはuS10のユビキチン化である7).停滞したリボソームのuS10に付加されたユビキチン鎖の連結様式はK63結合型であり,RQCにおけるペプチド鎖分解の前段階であるリボソームのサブユニット解離,すなわち翻訳の強制終了の目印となる.サブユニット解離は,リボソーム停滞を解消するとともに翻訳産物の分解を可能とし,異常翻訳の影響を最小限に抑えるための合理的な戦略である.
ではリボソーム停滞はどのように感知されるのか? 近年の生化学的および構造学的解析より,リボソームどうしの衝突が停滞の目印の一つとされることが明らかになってきた.クライオ電子顕微鏡を用いた解析では,翻訳効率が低いmRNA配列の導入や翻訳因子の活性阻害によって停滞したリボソームが後続のリボソームと衝突して形成する特徴的なdi-ribosome(disome)またはtri-ribosome(trisome)構造が観察されている8, 11, 12, 30, 31).出芽酵母の代表的なdisome構造では,二つのリボソームの小サブユニットタンパク質Asc1が衝突して直接相互作用しており,この相互作用がdisomeの構造を安定化していると考えられる8).重要なことに,RQCの初期段階でHel2のユビキチン化標的となるuS10は衝突部位の近傍に位置している.また,RQCを誘導する内在性停滞配列を持つSDD1 mRNAを試験管内で翻訳させ,リボソーム衝突を再現した実験系では,Hel2が単独に存在するリボソームよりもdisome, trisomeに優先的に結合してユビキチン化する12).これらの結果から,Hel2が衝突リボソームを認識する「衝突センサー」であることが強く示唆される(図1A①,②).興味深いことに,SDD1 mRNA上で二つのリボソームが衝突して形成するdisomeと比較して,三つの衝突リボソームからなるtrisomeの方が,より効率的にユビキチン化される.Hel2はリボソームの衝突部位に結合し,衝突するリボソームの数に応じてリボソームとの親和性が変化する可能性が考えられる.しかし,Hel2自身の構造は決定されておらず,リボソームとの実際の結合様式およびユビキチン化活性の制御機構の詳細を今後明らかにしていく必要がある.
3)RQT複合体による衝突リボソームのサブユニット解離
RQT複合体は,RQCにおいて衝突したリボソームのサブユニット解離を触媒する因子である.出芽酵母のRQT複合体はATPアーゼ型のRNAヘリカーゼ活性を有するSlh1,ユビキチン結合ドメインCUEを持つCue3,機能不明のC2HC5型ジンクフィンガードメインを持つRqt4から構成される.SDD1 mRNAで衝突したtrisomeがRQT複合体およびATPの添加によってサブユニットに解離することが試験管で再現されており,特にSlh1のATP結合活性中心に位置する316番目のリシン残基がサブユニット解離に必須である12).最近,連続レアコドン配列で衝突したdisomeを用いた研究でも同様の結果が得られ,さらに先頭で停滞したリボソームにRQT複合体が結合した構造が決定された32).リボソーム小サブユニットのヘッドがボディに対して反時計回りに20°回転した,サブユニット解離の中間状態とみなされる特殊な構造クラスが観察されている.ヘッドの回転はmRNAを3′側へ引っぱることによって起こると予想され,この引っぱる力はmRNAトンネルの入口付近に結合するSlh1のヘリカーゼ活性により加えられると推定される.結果として,停滞したリボソームのサブユニット会合の不安定化に加え,衝突してきた隣接のリボソームの構造変化を引き起こす可能性がある.最終的に後続のリボソームがくさび(楔)のように先頭のリボソームのサブユニット間に突き刺さって解離させるというモデルが提唱されている(図1A③).
Slh1がサブユニット解離に中心的な役割を果たすことが示された一方,Cue3, Rqt4の機能は十分に理解されていなかった.最近,RQT複合体とユビキチン鎖との結合アッセイにより,Cue3はCUEドメインを介して,Rqt4はN末端領域を介してK63結合型ポリユビキチン鎖(K63鎖)に特異的に結合することが明らかになった33).RQT複合体を高速原子間力顕微鏡で観察すると,Cue3はSlh1から約10 nmの範囲内に位置している一方,Rqt4の長い天然変性領域が触手のようにSlh1から約30 nmの範囲内で激しく動く様子がみられる.このことから,RQT複合体は広い可動範囲を持つRqt4を用いてユビキチン鎖を走査し,Rqt4がリボソームの遠位側からK63鎖を識別し結合した後,Cue3が近位側からK63鎖に結合すると同時にSlh1をユビキチン化部位の近傍に固定する可能性が考えられる33).
また近年,ヒトにおける内在性の停滞誘導配列を持つXBP1u mRNA34)を用いて,リボソームの衝突からZNF598によるユビキチン化,ヒトRQT複合体(ASC-1)によるK63鎖の認識およびサブユニット解離までの多段階反応が試験管内で再構成された11).特筆すべきことに,uS10だけでなく,出芽酵母にみられないeS10のユビキチン化も検出された.さらに,哺乳類と出芽酵母における衝突リボソーム構造,特に衝突部位の構造の違いも発見された.RQCの一般的な分子メカニズムは高度に保存されているが,哺乳類のリボソーム衝突とその解消は,進化上で出芽酵母に存在しない制御機構を獲得している可能性が示唆されている.
4)新生ペプチド鎖への分解目印の付加
停滞したリボソームがサブユニットに解離後,大サブユニットに結合するLtn1によって,合成途上の新生ペプチド鎖にK48結合型ポリユビキチン鎖が付加され,これがプロテアソーム依存的分解の目印となる(図1A④)1, 14, 19, 35).Ltn1が欠損した場合,出芽酵母のRqc2が小サブユニット,mRNAおよびGTPアーゼ非依存的に,新生ペプチド鎖のC末端にアラニンおよびトレオニンを付加する20).この非典型的な翻訳反応はCATテイル付加と呼ばれており,リボソームトンネル内に埋もれていた新生ペプチド鎖上のリシン残基をリボソーム外に露出させることで,Ltn1によるユビキチン化を促進する機能を持つ36).最近,大サブユニットに結合したRqc2の高解像度の構造が得られ,CATテイル付加反応は,①Rqc2によるtRNAのリクルート,②ペプチド転移反応,③転座様反応,④脱アシル化tRNAの解離,という四つの過程の繰り返しによって行われることが示唆された37).なお,このときのペプチド転移反応には翻訳伸長因子eIF5Aが促進因子として働き,eIF5Aの結合がCATテイル付加に必要なリボソーム大サブユニットの構造変化を引き起こすことが示された.
哺乳類においても,異常翻訳産物はLtn1によってユビキチン化されプロテアソーム依存的な分解へと導かれる15).一方で,出芽酵母と異なり,Rqc2のホモログであるNEMFは新生ペプチド鎖にアラニンのみで構成されるAlaテイルを付加する21, 22).Alaテイルはデグロンとしての役割を担い,E3ユビキチンリガーゼであるKLHDC10およびPirh2が直接結合することによって新生ペプチド鎖の分解を誘導する38, 39).原核生物にもRqc2/NEMFのホモログが存在しており,たとえば枯草菌ではRqcHが新生ペプチド鎖にAlaテイルを付加し,ClpXPプロテアーゼによる分解を惹起する40, 41).このように,ユビキチン-プロテアソーム系を持たない細菌で利用されている異常翻訳産物分解の戦略がヒトにおいて一部保存されている点は非常に興味深い.
近年,ubiquitin-fold modifier 1(UFM1)化42)と呼ばれるユビキチン様修飾と哺乳類におけるタンパク質恒常性維持との関連が大きな注目を集めている.UFM1化を触媒するE3複合体であるUFL1-UFBP1-CDK5RAP3は小胞体膜に局在し,リボソーム大サブユニットタンパク質uL24のUFM1修飾を行う43, 44).ごく最近報告された構造解析から,大サブユニットに結合したE3複合体が,uL24のUFM1化依存的に粗面小胞体膜上のトランスロコンと大サブユニットとの間の相互作用を弱めることで,正常な翻訳終結後の大サブユニットを小胞体膜から解放させる機能を持つことが示唆されている45, 46).一方で,XBP1uなどの小胞体膜上で翻訳されるmRNAでリボソームが停滞すると,翻訳を強制終了させ新生ペプチド鎖をLtn1依存的に分解する「小胞体RQC」が誘導される47).uL24のUFM1修飾が小胞体RQCに必須であることが最近判明し48, 49),強制翻訳終了時の大サブユニットの再利用および異常翻訳産物の排除にもUFM1化が重要な役割を担う可能性が十分考えられる(図1B).UFM1化E3複合体の機能を詳細に解明することで,高等真核生物における多面的なRQC制御機構の理解が深まると期待される.
1)リボソーム衝突によって引き起こされるmRNAの切断・分解機構
リボソーム停滞・衝突の要因としてmRNA上の停滞誘導配列が多数同定されている.連続した塩基性アミノ酸配列50),使用頻度の低いレアコドンの連続配列や特定のコドン対51–54),強固な二次構造を形成する配列55, 56)を翻訳した場合,リボソームが停滞・衝突する.また酸化ストレス等によって損傷・修飾を受けたmRNA57, 58)上でもリボソームが停滞するため,細胞は,リボソーム停滞を目印とし,異常mRNAを積極的に除去する品質管理機構を保持している.
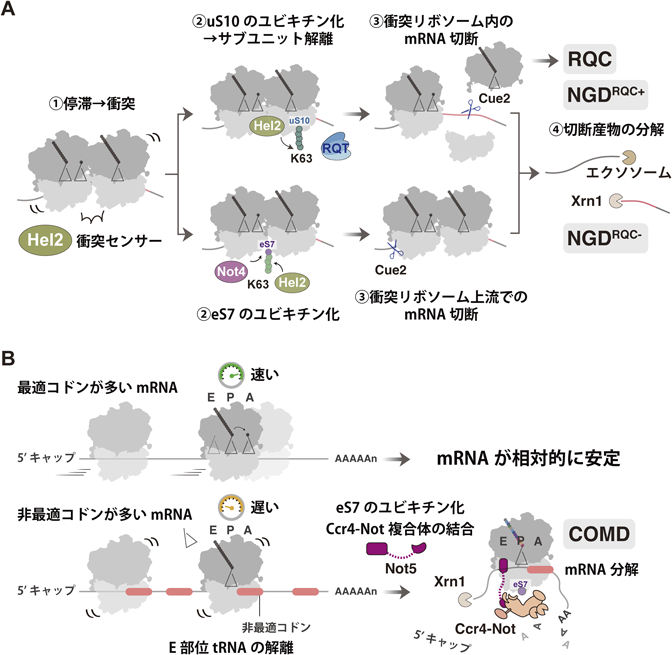
出芽酵母では,衝突センサーHel2によるリボソームのユビキチン化は翻訳産物の分解系RQCだけでなく,mRNAの切断・分解系No-go Decay(NGD)も惹起する8, 59).NGDにおいて,衝突したリボソームの近傍でエンドヌクレアーゼCue2がmRNAを切断した後60),5′,3′切断産物がそれぞれ細胞質内のエキソヌクレアーゼであるエクソソームとXrn1によって分解される8).NGDは,RQCと共役するか否かによって,NGDRQC+とNGDRQC-という二つの経路に分岐する8, 61)(図2A).NGDRQC+はHel2によるuS10のユビキチン化を介したリボソームのサブユニット解離を必要とし,mRNAの切断部位が衝突したリボソーム内に存在する.uS10のユビキチン化もしくはサブユニット解離が阻害されると,代わりにeS7のユビキチン化に依存したNGDRQC-が誘導され,このときmRNAはNGDRQC+の場合よりも上流側で切断される.eS7のユビキチン化は,Ccr4-Not複合体(次項参照)の構成因子であるNot4による一つ目のユビキチンの付加(モノユビキチン化)と,Hel2によるK63鎖の伸長(ポリユビキチン化)からなる二段階反応である8).
mRNA切断を担うCue2の立体構造はまだ解明されていないが,近年の遺伝学的解析により,Cue2のN末端領域が切断部位の選択性に重要であることが見いだされた61).停滞誘導配列を持つmRNAの切断中間産物の末端をプライマー伸長法で決定し,異なるCue2変異体を発現した酵母株間で切断末端の位置を比較した結果,Cue2の122番目のトリプトファン残基が,衝突したリボソーム内でのmRNA切断(NGDRQC+)に必要であることが示された.一方で,Cue2のN末端領域に存在するユビキチン結合ドメインは,衝突したリボソームの上流でのmRNA切断(NGDRQC−)に必須であるが,リボソーム内でのmRNA切断(NGDRQC+)には必須でないことが明らかとなった.このようにCue2が切断部位を変えることで,リボソームのユビキチン化部位やサブユニット解離の状況に柔軟に対応できることは非常に興味深い.線虫や細菌においても,Cue2にも存在するmRNA切断活性中心であるSMRドメインを有する因子が,リボソーム停滞に起因するmRNA分解に寄与することが報告されている62–65).哺乳類にも類似した機構が存在するかについては今後検証していく必要がある.
2)非最適コドンにおけるリボソームの速度低下に起因するmRNAの不安定化機構
コドンの最適度はコドンの使用頻度と,各々のコドンに対応するtRNA量との関係を表す.個々のmRNAが持つ固有の安定性がコドンの最適度によって調節され,最適度が高いコドン(最適コドン)を持つmRNAほど安定であり,最適度が低いコドン(非最適コドン)が多く存在するmRNAは不安定である66).出芽酵母を用いた研究より,コドンの最適度依存的なmRNAの安定性決定機構において,Ccr4–Not複合体が主要な制御因子として働くことが近年明らかになった67).
Ccr4–Not複合体はmRNAの転写,分解,翻訳抑制など,遺伝子発現を多方面から制御する複合体であり,リボソームと直接結合する.Ccr4–Not複合体と共精製されたリボソームによって翻訳されるmRNAの配列特徴を調べる選択的リボソームプロファイリング解析では,Ccr4–Not複合体とリボソームの親和性がA部位に位置するコドンの最適度と非常に強い逆相関を示す67).また,Ccr4–Not複合体の構成因子の一つであるNot5が,A部位にtRNAを含まないリボソームのE部位に結合することが構造解析で判明している.非最適コドンを翻訳する際に,対応するtRNAの量が少ないため,A部位にtRNAが存在しない状態が長く継続し,結果としてE部位に結合していたtRNAが解離する.このようなリボソームにCcr4–Not複合体がNot5を介して結合することで,mRNAの3′末端のポリA鎖の短縮化および5′末端のキャップ構造の除去を促進し68, 69),mRNA分解を誘導すると考えられる(図2B).Not5とリボソームの結合には,Not4によるリボソームタンパク質eS7のユビキチン化が必要である.Not5やNot4の欠損株,eS7のユビキチン部位の変異株では,コドンの最適度とmRNAの安定性との相関が失われ,mRNAの動的恒常性が破綻してしまう67).こうしたコドンの最適度により制御されるmRNA分解系はcodon optimality-mediated mRNA decay(COMD)と名づけられた.興味深いことに,前述したmRNAの内部切断分解系であるNGDRQC−もNot4によるeS7のユビキチン化を必要とする8).機序の異なる二つのmRNA分解系の関係を解析することは,翻訳と共役したmRNA不安定化機構の全体像を理解する上で重要である.また,試験管内再構成系では,Not5のヒトホモログであるCNOT3が停滞した哺乳類リボソームの空のE部位に結合し,Not4のホモログであるCNOT4がeS7をユビキチン化することが最近報告され70),COMDがヒトでも保存されている可能性が示唆されている.
1)機能欠損リボソームサブユニットを分解する品質管理機構
mRNA上の停滞誘導配列のほか,翻訳装置であるリボソーム自身の機能欠損も異常翻訳の原因となりうる.出芽酵母では,活性中心を形成するリボソームRNA(rRNA)に塩基置換変異を導入すると,変異型rRNAを保持するリボソームサブユニットはnonfunctional rRNA decay(NRD)と呼ばれる機構によって分解・排除される71).NRDには,大サブユニットのペプチド転移活性中心(peptidyl-transferase center:PTC)を形成する25S rRNAを分解標的とする25S NRDと,小サブユニットの暗号解読活性中心(decoding center:DC)を形成する18S rRNAを分解標的とする18S NRDが存在する.ここでは,分子機構がより詳細に解析されている18S NRDについて説明する.
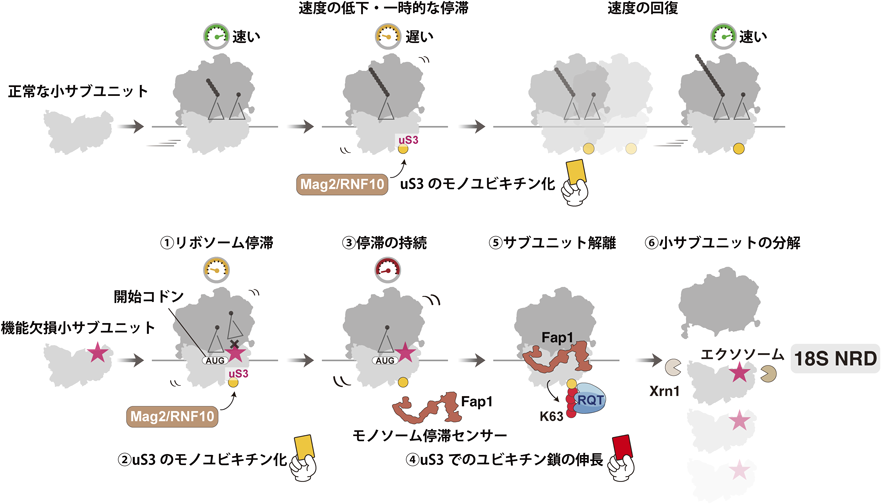
18S NRDには翻訳が必要であり,暗号解読活性の欠失に起因する翻訳異常が引き金となる可能性が以前から示唆されていた72).近年,遺伝学的手法を用いた解析より18S NRDに必須な因子が新たに同定され,リボソームプロファイリング法によって18S NRDを惹起する翻訳異常の実体が解明されてきた73–75).18S NRDのモデル基質である18S:A1755C(DC変異型)rRNAを保持する機能欠損リボソームは開始コドンで停滞し,E3ユビキチンリガーゼMag2とFap1によって認識される.Mag2とFap1はリボソーム小サブユニットタンパク質uS3を順次ユビキチン化し,RQT複合体依存的なサブユニット解離を誘導する.ユビキチン化された小サブユニットは最終的に分解へと導かれ,この過程にはmRNA分解を担うXrn1およびエクソソームが関与することが示唆されているが72),分解機構の詳細は明らかになっていない(図3).
2)Mag2とFap1による停滞認識とユビキチン化
DC変異型18S rRNAを保持するリボソームを特異的に精製し,翻訳されているmRNAの領域を解析する選択的リボソームプロファイリングの結果,DCの活性が失われたリボソームのほとんどはA部位にtRNAを含んでおらず,P部位に開始コドンを位置させた状態で停滞していることが判明した75).機能欠損リボソームは2番目のコドンの解読ができず,翻訳伸長反応に移転できないことが示された.一般的に,開始コドンより上流には翻訳伸長中のリボソームが存在しないため,開始コドンでのリボソーム停滞は後続のリボソームとの衝突を伴わない「単独」の停滞となる.これは,前述したRQC, NGDの引き金であるリボソーム衝突とは異なる停滞認識機構の存在を示唆し,衝突センサーHel2が欠損していてもDC変異型18S rRNAが分解される結果と一致している.一方,18S NRDに必須な因子を探索する遺伝学的スクリーニングによりMag2とFap1が同定され,これらのE3ユビキチンリガーゼが機能欠損リボソームのuS3をユビキチン化することが見いだされた.興味深いことに,試験管内のユビキチン化反応系では,Mag2がuS3にユビキチンを1分子のみ付加する74, 75).さらにFap1を添加するとユビキチン鎖が伸長しK63鎖が形成されるが,Fap1によるK63鎖の形成にはMag2による最初のユビキチン付加が前提となる75).
Mag2とFap1はいずれもリボソームに結合しており,停滞センサー因子の候補とされるが,二つの因子は異なる基質特異性を持つことが示唆されている75).Mag2, Fap1それぞれと共精製される内在性リボソームの特徴を選択的リボソームプロファイリングによって解析した結果,Mag2とリボソームとの親和性がA部位のコドンの最適度と逆相関することが示された.Mag2は非最適コドンを翻訳中のリボソームなど,速度が低下したリボソームを全般的に認識していると考えられる.そのメカニズムの解明には今後の詳細な解析が必要であるが,先述したNot5による失速リボソームの認識と類似した機構である可能性がある.
Mag2と異なり,Fap1とリボソームの結合は,開始コドン近傍や主にモノソームによって翻訳されるmRNA上など,隣接するリボソームの出現頻度が低い場所に集中する傾向があることが示された.mRNAが複数のリボソームで翻訳されるポリソームと,単独のリボソームで翻訳されるモノソームを比較した場合,Fap1はモノソームに対してより高い親和性を有する.クライオ電子顕微鏡を用いた解析の結果,Fap1が小サブユニット上のmRNAの入口と出口を連結する細長い構造をとり,両端のドメインがそれぞれmRNAと相互作用している様子が観察された75).翻訳伸長反応中のリボソームの構造変化とmRNAの相対的な動きがFap1との相互作用を破壊するため,リボソームが静止状態にあるときのみFap1の安定な結合が可能になると考えられる.また,RQCやNGDを誘導する衝突リボソームの構造と比較すると,リボソームどうしの衝突が停滞したリボソームへのFap1の結合を立体的に妨害することが示唆される.したがって,Fap1は単独のリボソーム上でmRNAの動きを感知し,衝突を伴わないリボソーム停滞に特化した「モノソーム停滞センサー」であると提唱された.
単独で停滞したリボソームは,衝突で形成される明確な構造的特徴を持たないため,正常な翻訳反応を行っているリボソームや,一時的に速度が低下したリボソームとの区別が難しい.また,大量のエネルギーを消費して合成された翻訳装置の分解は,細胞にとって高いコストとリスクを伴う.以上の知見を合わせると,停滞した機能欠損リボソームはMag2とFap1の両方に認識されて初めて分解の標的となる,という二重確認モデルが考えられる(図3).Mag2は衝突の有無にかかわらず,翻訳速度の遅いリボソームをモノユビキチン化して異常翻訳装置の候補とする.その後,単独のリボソーム停滞が持続する場合にFap1が結合してユビキチン鎖を伸長させ,18S NRDを惹起する.このようにユビキチン化が多段階で行われることは,二つの停滞センサー因子によって異常と判定された翻訳装置のみを排除する慎重な戦略といえる.
3)機能欠損リボソームのサブユニット解離
18S NRDでは,uS3のユビキチン化がリボソームサブユニットの解離を促し,機能欠損小サブユニットの分解を誘導するとともに正常な大サブユニットの再利用を可能とする74, 75).解離因子であるRQT複合体の構成タンパク質Slh1の欠損株ではDC変異型18S rRNAが顕著に安定化し,uS3でのユビキチン鎖がRQT複合体をリクルートすることを示唆する結果が得られている74).これは,RQCとNGDを惹起するuS10でのユビキチン鎖と類似した役割であり,RQT複合体によるリボソームサブユニットの解離は,異常翻訳産物,基質,装置の分解系に共通して,翻訳品質管理機構にきわめて重要なステップであることを示唆している.RQT複合体が多様な停滞状態に適応できることが示されるが,その詳細な機序は不明である.衝突リボソームで提唱されているモデルとして,Slh1の活性依存的にmRNAが引っぱられると,後続のリボソームがくさびとして働いて先頭のリボソームのサブユニット解離を引き起こす.一方で,後続のリボソームが存在しない単独のリボソーム上では,RQT複合体が衝突リボソーム上とは異なる機序で働くと想定される.また,RQT複合体以外のサブユニット解離因子として,翻訳終結因子eRF1–eRF3と相同性を持つDom34-Hbs1が18S NRDに部分的に寄与することが報告されている72, 74, 76).Dom34-Hbs1は空のA部位を持つリボソーム77–80)や翻訳に参加していないリボソーム81)を解離させる機能を果たすが,18S NRDにおける役割やRQT複合体との関連はいまだ不明である.機能欠損リボソームを基質として用いたサブユニット解離の試験管内再構成や構造解析で,新たなサブユニット解離機構が解明されることが期待される.
4)停滞に起因するリボソーム分解系の一般性と保存性
リボソーム自身の機能欠損に限らず,さまざまなストレスによって18S NRDが誘導されることが示唆されている.出芽酵母では栄養枯渇,翻訳阻害,核酸損傷誘導といったリボソーム停滞の誘導条件下ではもちろん,mTOR阻害や小胞体ストレスなど,リボソーム停滞との直接的な関連が知られていないストレスもuS3のユビキチン化を促進する57, 82, 83).興味深いことに,一部のストレス時にみられるuS3のユビキチン化は衝突センサーHel2に依存しており,リボソーム衝突も小サブユニットの分解を引き起こしうる可能性が高い.また,試験管内では,Mag2がuS3をモノユビキチンした後,Hel2はFap1と同様にユビキチン鎖を伸長させる活性を有することが明らかとされている74).
ストレスによるuS3のユビキチン化の亢進は哺乳類細胞でも観察される82, 84–86).近年,Mag2のヒトホモログであるRNF10がuS3およびuS5のユビキチン化に必須であることが報告された85, 86).RNF10を過剰発現させると,ユビキチン化の上昇とともに,細胞内のリボソーム大サブユニットに対する小サブユニットの割合が低下する85).このことから,哺乳類においてもリボソームのユビキチン化に依存した小サブユニット特異的な分解系が存在することが強く示唆される.この分解系は,異常なリボソームを排除するだけでなく,ストレス時にリボソームサブユニットの量を調節し,ストレス応答の一環として機能する可能性がある.ストレス時の翻訳の状態や小サブユニットの寿命制御について解析していくことは,停滞に起因するリボソーム分解系の一般性の検証および生理的機能の理解に不可欠である.
本稿では,異常翻訳であるリボソーム停滞を認識・解消するとともにその産物・基質・装置を分解する翻訳品質管理機構を紹介してきた.細胞はさまざまな停滞に効率よく対応するために,基質特異性の異なる停滞センサー因子を用いた複数の停滞感知機構を備えている.リボソーム停滞の様式が異常翻訳の原因を反映し,細胞は停滞の特徴に基づいて排除すべき「部品」を見極める能力を有する可能性がある.たとえば,翻訳伸長の最初段階に生じる異常である単独のリボソーム停滞は翻訳装置の品質不良を示している可能性が高いのに対して,mRNAの途中までの翻訳を前提とするリボソーム衝突は,翻訳基質の配列異常に起因することが多いと想定される.これに対応して,単独のリボソーム停滞はリボソーム自身を分解に導き,リボソーム衝突はmRNA・新生ペプチド鎖の分解を引き起こす.一方で,品質管理経路は互いに独立して実行されるとは限らない点に注意する必要がある.停滞センサー因子とリボソーム上のユビキチン化標的は多対多の関係性を持っており,ユビキチン化部位と品質管理経路の対応関係も単純ではない.リボソームのユビキチン化を目印とする複数の品質管理経路は高い共通性と柔軟性を持ち,細胞内の状況に応じて経路間のバランスが巧妙に制御されている可能性が考えられる.しかし,これまでの研究では主に,標的の異なる品質管理経路それぞれ独自のモデル基質に着目した解析が行われてきたため,品質管理経路間のクロストークについての知見が不足している.リボソーム停滞における翻訳産物・基質・装置を同時に追跡する手法が開発されれば,翻訳品質管理機構の全容の理解に大きく貢献できると期待される.
出芽酵母で発見された翻訳品質管理経路の多くは,その分子メカニズムがヒトまで保存されている.RQC関連遺伝子内の変異と神経変性疾患および自閉症との関連が示唆されていおり87, 88),マウスではRQCの機能喪失は神経細胞の発達障害や細胞死,行動異常を引き起こす89, 90).また,翻訳装置であるリボソームの異常は,造血不全や,皮膚の異常,腫瘍など,総じてリボソーム病と呼ばれる疾患の原因となる91).異常翻訳とその産物や装置を排除する機構の生理的機能の解明は,疾患発症機構を理解するための新たな切り口となる可能性が考えられる.さらに,翻訳基質であるmRNAの安定性制御機構の理解は,ワクチンに代表されるmRNA医薬品の開発研究の発展につながると期待される.