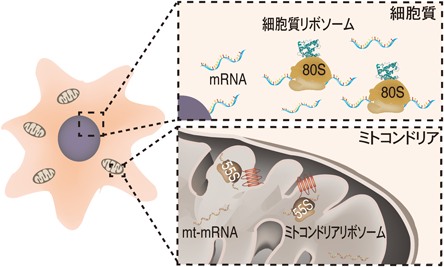
ミトコンドリアは細胞内の主要なエネルギー供給源であり,酸化的リン酸化反応を介してATPを産生し,細胞の恒常性を維持している.ミトコンドリアの起源であるプロテオバクテリアが細胞内共生に至り1),約20億年を経て,ミトコンドリアを形作る遺伝子の多くは核ゲノムへと移行した.しかしながら,ミトコンドリアは依然として自身のゲノムDNAを保持し,わずかなミトコンドリアタンパク質をコードしている.ヒトのミトコンドリアゲノムは,ポリシストロニックmRNA 2種を含む11種のmRNA(mt-mRNA)に加え(タンパク質としては合計13種),2種のrRNA(mt-rRNA),22種のtRNA(mt-tRNA)を保持している.オルガネラ内で専用のリボソームを組み立てることで,酸化的リン酸化複合体に必須の13種のタンパク質を合成している2).その結果,真核生物の細胞内には細胞質とミトコンドリア内部で二つの異なるタンパク質合成系が共在することになる.これらは,その駆動される場所やメカニズムが大きく異なる(図1).
ミトコンドリア翻訳活性はATP産生能をつかさどるため,その異常は脳卒中様エピソード(MELAS),ミオクロニーてんかん(MERRF),リー症候群などの疾患を引き起こすことが知られている3–6).これらの病態はミトコンドリア機能異常に基づくことからミトコンドリア病と総称され,わが国では難病指定されている多臓器疾患である.このように,ミトコンドリア翻訳は細胞において重要な反応であるにもかかわらず,そのメカニズムや制御機構の理解は,解析の困難さから立後れている.これは細胞質に比べ,ミトコンドリア翻訳は細胞全体から見ればごく一部であり,細胞質翻訳と夾雑する中で,ミトコンドリア翻訳のみを取り出して解析することが困難であることによる7).本稿では,ミトコンドリア翻訳を研究するために開発された生化学的解析手法,次世代シークエンス技術を紹介しつつ,ミトコンドリアリボソームプロファイリングの発展に焦点を当て,その最新の活用例を紹介する.
1)新規合成タンパク質の放射性標識
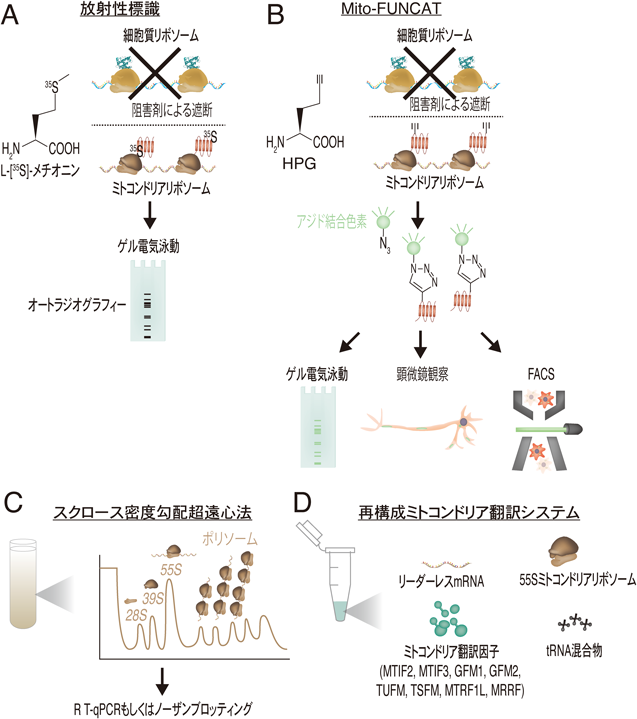
古くから,細胞内のタンパク質合成量を測定するために,新規合成タンパク質の代謝標識法が用いられてきた.放射性標識アミノ酸(たとえば,L-[35S]-メチオニン単独やL-[35S]-メチオニンとL-[35S]-システインの混合物)で一定時間細胞を処理することにより,その処理時間に合成されたタンパク質を標識することができる.細胞内では,細胞質リボソームによって翻訳されるタンパク質が大部分を占めるため,そのままではミトコンドリア翻訳の検出は困難であるが,cycloheximide, anisomycin, emetineなどの細胞質リボソーム特異的な阻害剤下8, 9)で実験を行うことにより,ミトコンドリアリボソームで合成されるタンパク質のみを標識することができる(図2A).定量は通常,ドデシル硫酸ナトリウム-ポリアクリルアミドゲル電気泳動(SDS-PAGE)とオートラジオグラフィーによって行われ,ヒトでは13個のタンパク質産物をすべて同定することができる10–13).
2)蛍光非標準アミノ酸タギング(FUNCAT)
放射性アミノ酸を用いる伝統的な方法は現在でも汎用されているが,X線撮影に基づく検出は,より広範な技術応用を妨げてきた.この課題を解決するために,クリックケミストリー(一対の官能基を迅速,効率的,特異的,共有結合的に連結する有機反応)に適合するメチオニン誘導体が開発された.L-ホモプロパルギルグリシン(HPG)とL-アジドホモアラニン(AHA)は,それぞれアルキン基とアジド基を持つ非放射性メチオニン誘導体であり,新たに合成されたタンパク質に組み込まれる性質を有する.それゆえ,その後の細胞溶解とクリック反応により,アナログが組み込まれたタンパク質を蛍光標識(たとえば,HPGにはアジド結合色素,AHAにはアルキン結合色素)することができる.この方法はもともと細胞質翻訳の測定のために開発されたが14–21),放射性アミノ酸を用いた方法と同様,細胞質翻訳の阻害剤を処理することで,ミトコンドリア翻訳のみを蛍光標識できる(mito-FUNCAT)(図2B)22–25).実際,標識タンパク質をゲル上で分析することで,ミトコンドリアゲノムにコードされる13個のタンパク質の合成率を定量することができる(on-gel mito-FUNCAT).さらに,mito-FUNCATをin situアプローチ23–25)や蛍光活性化セルソーティング(FACS)26)に応用することで,単一細胞におけるミトコンドリア翻訳の空間的ダイナミクスや細胞集団でのミトコンドリア翻訳の不均一性を解析することができるようになった(図2B).
3)スクロース密度勾配超遠心法
タンパク質合成を担うリボソーム複合体の形成を測定することで翻訳状態を解析することができる.ショ糖密度勾配超遠心法では,質量に沿ってリボソーム複合体を分離し,小サブユニット(28S),大サブユニット(39S),モノソーム(55S),ポリソームを分画することができる(図2C).mRNA上のリボソーム数が多いほどタンパク質合成速度が速いことを示すため,ポリソーム画分のノーザンブロッティングや定量的逆転写PCR(RT-qPCR)を行うことで,個々のmt-mRNAの翻訳効率を評価することができる(図2C)22, 27–29).
4)再構成ミトコンドリア翻訳システム
翻訳の素過程である開始,伸長,終結,リボソームリサイクルといった,ミトコンドリア翻訳の各ステップに関する詳細な分子メカニズムを理解するには,無細胞in vitroアプローチが有用な手段となる.精製した組換えミトコンドリア翻訳因子,55Sミトコンドリアリボソーム,酵母または大腸菌由来のtRNA混合物を用いて,哺乳類ミトコンドリア翻訳の再構成系が開発された(図2D)30, 31).このpure systemは,mt-mRNAの特徴であるリーダー配列を持たないmRNAから,モデルタンパク質(細菌のジヒドロ葉酸レダクターゼやナノルシフェラーゼ)とミトコンドリアゲノムにコードされたタンパク質を合成することができる(図2D).
3. 次世代シークエンサーによるミトコンドリア翻訳の網羅的解析
上述の古典的生化学によるアプローチや近年の構造生物学の進歩によって,ミトコンドリア翻訳のメカニズムに関する知見は徐々に増加し,他の翻訳系(細胞質翻訳系や細菌翻訳系)と異なるユニークなメカニズムによって駆動されることが明らかになってきた2).一方で,実際の細胞や組織におけるミトコンドリア翻訳の摂動や,疾患が及ぼすミトコンドリア翻訳への影響などを,包括的かつ高解像度に理解することは難しいのが現状である.ここでは,その課題を補完する技術として,リボソームプロファイリング(またはRibo-Seq)を紹介し,その最新のアップデートについて論ずる.
1)リボソームプロファイリングとその技術的制約
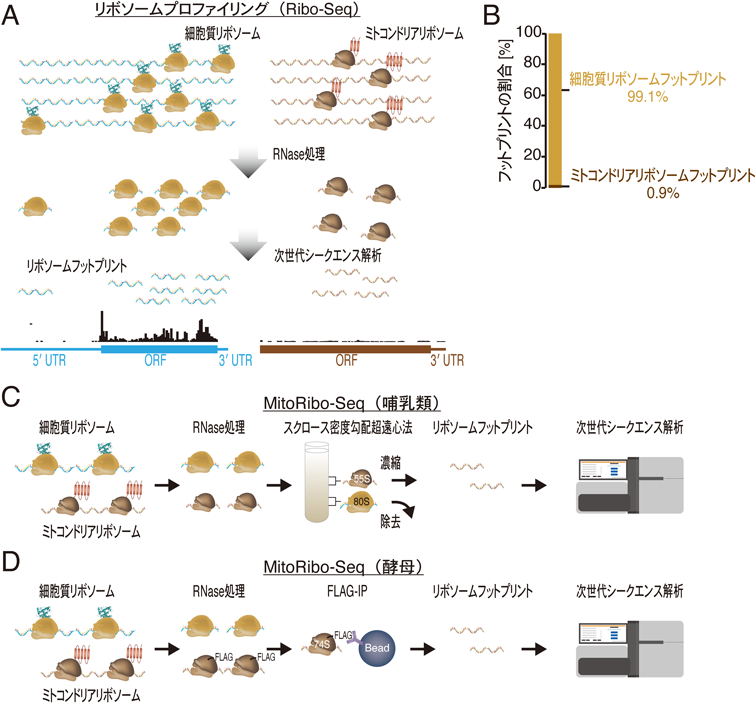
リボソームプロファイリングは,次世代シークエンサーを応用し,細胞内の翻訳状態を網羅的かつ定量的に解析する手法である32).リボソームは,mRNA上で塩基配列を1コドンずつ読み進めることでタンパク質を合成する.このmRNA–リボソーム複合体にリボヌクレアーゼ消化を施すと,途端にmRNAは分解するが,リボソームで保護されたmRNA部分は分解を免れる.それゆえ,残存したmRNA断片(リボソームフットプリント)を精製し,次世代シークエンサーで読み解くことで,リボソームの数や位置情報をゲノムワイドに調べることができる(図3A).実際,各フットプリントの位置は,リボソームによって物理的に保護されたmRNA領域を示すため,詳細なデータ解析によって,mRNA上のリボソーム数や各コドンにおけるリボソームの翻訳速度,リボソームがmRNA上で翻訳を開始する位置と終了する位置などについての情報を得ることができる33).また,リボソームプロファイリングと同時にRNA-Seqを行い,リボソームフットプリントの数をRNA量で補正することで,翻訳とRNA量の変化を明確に切り分けて解析することが可能である.
リボソームプロファイリングでは,細胞質リボソームとミトコンドリアリボソーム由来のフットプリントを同時に解析することができる34–36).しかしながら,従来の方法では,ミトコンドリアリボソーム由来のフットプリントは全体の1%程度しか得られず(図3B),ミトコンドリア翻訳を1コドン分解能で理解することは難しいという課題があった.
2)ミトコンドリアリボソームプロファイリング
上述のリボソームプロファイリングが抱える問題を解決するために,ミトコンドリアリボソームフットプリントを濃縮する改良型のリボソームプロファイリングが開発されてきた.たとえば,ヒトの細胞質リボソーム(80S)とミトコンドリアリボソーム(55S)はサイズが異なるため,スクロース密度勾配超遠心とそれに続く分画によって分離することができる(図3C)29, 37–44).一方でこの方法は,細胞質リボソーム(80S)とミトコンドリアリボソーム(74S)のサイズが類似している酵母には適さない.それゆえ,遺伝子組換えによってミトコンドリアリボソームタンパク質(MrpS1/uS17m)にFLAGタグをつけ,免疫精製によって分離するアプローチがとられている(図3D)45, 46).これらのミトコンドリアリボソームプロファイリング(MitoRibo-Seq)法により,ミトコンドリア翻訳の詳細な解析が可能になった.実際にmt-tRNAの修飾29, 39, 40),mt-tRNAの点変異37),細胞質翻訳との共役関係42, 45),TACO1によるPro-Proコドンの伸長反応促進43)などが明らかにされている.
MitoRibo-Seq法は,ミトコンドリアリボソームフットプリントのカバレッジを改善し,コドン分解能での解析を可能とする.しかしながら,この手法が開発され,約10年が経過したにもかかわらず,ミトコンドリア翻訳のコドン分解能での理解はいまだ乏しいのが現状である.これは,従来のMitoRibo-Seq法の技術的煩雑さに起因する.たとえば,スクロース密度勾配超遠心法は,ローターによって1度に扱えるサンプル数が最大6サンプルまでに限られ,精製に時間がかかる(~1日)というスループット性の欠点がある.また,酵母に用いられている免疫沈降法によるアプローチは,貴重な疾患細胞やマウス組織などに応用することは困難である.こういった技術的制約から,MitoRibo-Seq法の使用は制限され,大規模な解析は行われてこなかった.
3)MitoIP-Thor-Ribo-Seq
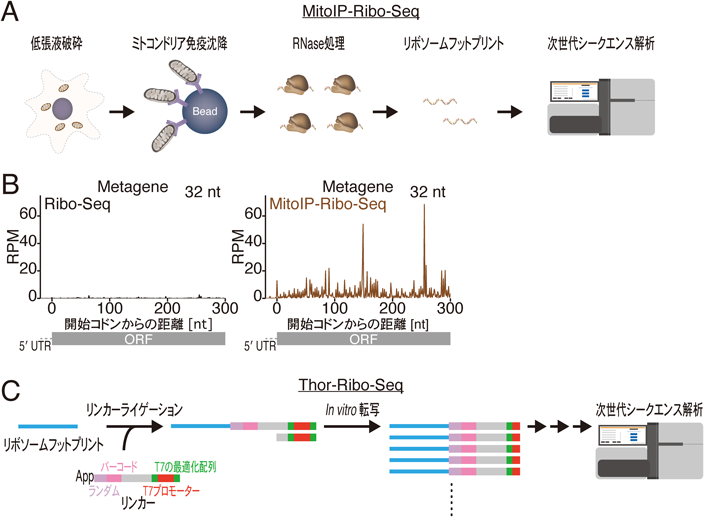
簡便かつ高解像度にミトコンドリア翻訳を解析するために,最近新たなミトコンドリアリボソームプロファイリング法が開発された47).この手法では,ミトコンドリア外膜に局在するTOM22の抗体を用いて,ミトコンドリアを免疫沈降し,リボソームプロファイリングを行う(図4A).ミトコンドリア免疫沈降法(MitoIP)によるミトコンドリアリボソーム精製は短時間(~2時間)かつ簡便に行えるため,並行して複数のサンプルを処理すること(10サンプル以上)が可能である.非常にシンプルな手法ではあるが,ミトコンドリア転写産物上のリードカバレッジを格段に増加させることができる(図4B).また,これらのミトコンドリアリボソームフットプリントは3塩基周期性を示し(リボソームはmRNA上を1コドンずつ3′方向へ移動するため,高品質なフットプリントは3塩基周期性を持つ)(図4B),コドン分解能での解析が可能である.
MitoIP法の欠点の一つとして,最終的に回収できるサンプル量が限られる(15 cmディッシュから約1~2 µgのトータルRNAに該当するサンプルを回収)という点があげられる.リボソームプロファイリングのライブラリー調製には,一般的に10 µgのトータルRNAに該当するサンプルが必要であり,サンプル量の制限は,ライブラリー調製を妨げうる.この課題を克服するために,リボソームプロファイリングに関わる新たな手法が適応された(図4C).T7 high-resolution original RNA(Thor)は,RNA依存性RNA増幅に基づく技術である48).ライブラリー調製の初期段階に,T7ポリメラーゼによってリボソームフットプリントの逆鎖RNAを増幅することで,少量サンプルからの堅牢なライブラリー調製を可能とした(図4C).MitoIP法とThor技術の組合わせは(MitoIP-Thor-Ribo-Seq),従来のMitoRibo-Seqの制約を補填する新たな手法となりうる.
4. MitoIP-Thor-Ribo-Seqの最新の適応例
MitoIP-Thor-Ribo-Seqの開発によって,ミトコンドリア翻訳のハイスループットかつハイコンテントな解析が可能となった.ここでは,その活用例を紹介しつつ,ミトコンドリア翻訳の複雑性とダイナミクスに関する最新の知見について議論する.
1)ミトコンドリアtRNA修飾の網羅的解析
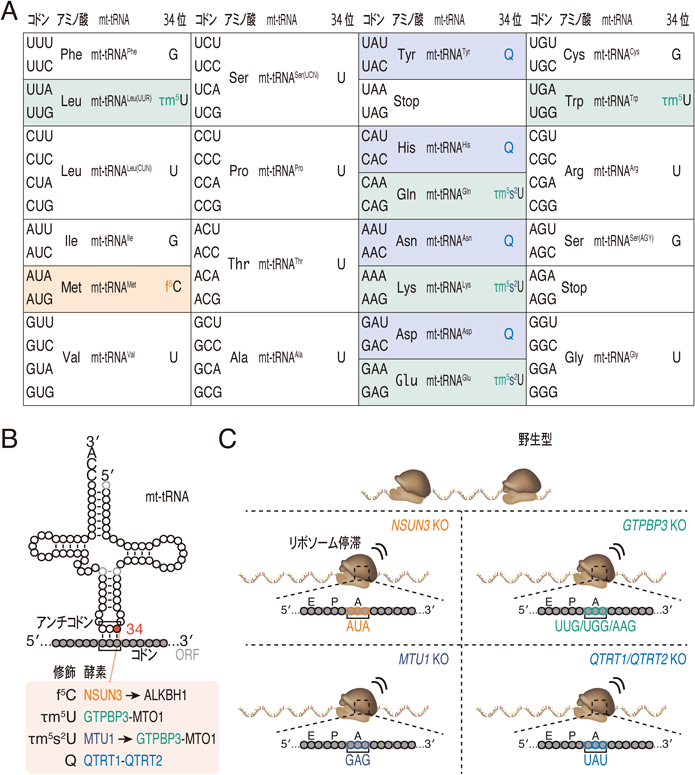
哺乳類ミトコンドリアでは,正規の遺伝暗号表と比較し変則的なものに進化している.AUAはMet, UGAはTrpによってコードされ,AGAおよびAGGは停止コドンとなっている(図5A).60のセンスコドンは,22種のmt-tRNAによって解読され,これはあらゆる生物およびオルガネラの翻訳システムの中で最小セットである49).少ない種類のtRNAから遺伝暗号を正確に解読するために,mt-tRNAは核コード修飾酵素によって修飾を受ける.特に読み取りに重要なウォブル位置(34位)には,5-ホルミルシチジン(f5C),5-タウリノメチルウリジン(τm5U),5-タウリノメチル-2-チオウリジン(τm5s2U),キューオシン(Q)が修飾される(図5B)35).これらの責任酵素とその役割は生化学的手法を軸に明らかにされてきているものの35, 39, 50–61),それぞれのtRNA修飾がミトコンドリア翻訳ヘ与える影響を包括的に理解するには至っていない.この課題に答えるために,それぞれの責任酵素を欠失させた細胞において,MitoIP-Thor-Ribo-Seqが適用された47).
網羅的な解析の結果,tRNA修飾の欠損によって,特定コドン上でミトコンドリアリボソームが停滞していることが明らかにされた(図5C).この失速の多くは,修飾されるtRNAに対応するコドンで発生し,修飾がデコーディング(A site)やトランスロケーション(P site)に重要な役割を持つことが示唆された47).たとえば,Metはf5C修飾を受けるが,この修飾酵素NSUN3の欠損によって,A site(およびP site)のAUAコドン上でミトコンドリアリボソームが停滞する(図5C).同様に,τm5U修飾はUUG/UGG/AAGコドン,τm5s2U修飾はGAGコドン,Q修飾はUAUコドンの解読に必要であることが明らかにされた(図5C)47).
2)ミトコンドリア病患者細胞への適応
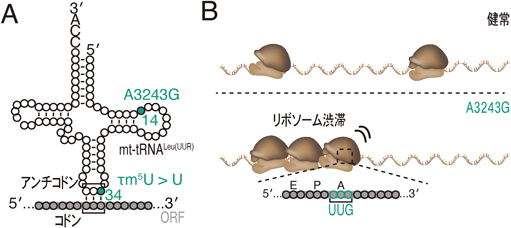
ミトコンドリア翻訳障害は,呼吸障害を伴うミトコンドリア機能障害を招き,多臓器での重篤な臨床症状を引き起こしうる3, 4).この原因としてmt-tRNAの病原性点突然変異が知られており,mt-tRNALeu(UUR)の14番目の位置に相当するA3243G変異は,MELASで最も頻発する変異である62–64).これまでの研究によって,A3243G変異によるtRNA立体構造の不安定は,MTO1-GTPBP3複合体の基質認識を妨げ,τm5U修飾率を低下させると考えられている(図6A)51, 65–67).
疾患細胞における翻訳ダイナミクスの変化を精査するために,A3243Gを持つMELAS患者筋芽細胞67, 68)にMitoIP-Thor-Ribo-Seqが適用された47).コドン分解能の解析の結果,UUGコドン上でミトコンドリアリボソームが停滞していることが明らかになった(図6B下).これは,前述のGTPBP3(m5U修飾酵素)欠損細胞の結果と同様の傾向を示しており,変異によるm5U修飾の欠損によって引き起こされていると推測される.さらに,UUGコドン前後のフットプリント変化を詳細に解析することによって,UUGコドンでの顕著なリボソーム停滞は,後続のリボソームの衝突,すなわちリボソーム渋滞を引き起こすことが明らかにされた(図6B下)47).
3)ミトコンドリアリボソーム衝突(ダイソーム)の解析
リボソームはmRNA上を一定の速度で伸長し新生鎖を合成するわけではなく,tRNA修飾による伸長の反応促進69)やタンパク質の局在化を図るための一時停止70)など,加速や減速を伴って伸長を進め,タンパク質合成の忠実性を保っている.ミトコンドリア翻訳においては,新生鎖を共翻訳的に膜挿入し71–76),アセンブリ因子との相互作用を介して酸化的リン酸化複合体が組み立てられていくため,伸長速度は厳密に制御される必要がある.実際,MT-CO1の翻訳は伸長途中で一時停止し,新生鎖とアセンブリ因子が相互作用することで翻訳が再開することが報告されている77).
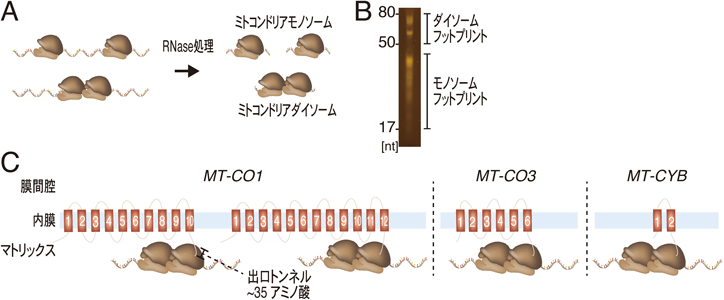
こういったプログラムされた翻訳休止点や疾患に基づくリボソーム渋滞をゲノムワイドに探索するために,衝突したリボソーム(ダイソーム)の解析が細胞質翻訳系や細菌翻訳系を中心に行われてきた78–83).一般的にRNase処理によって生成されるリボソームフットプリントは約20~30塩基長であるが,ダイソームではリボソーム二つ分の長さに相当するフットプリントが生成されるため,50~80塩基長のRNA断片を精製することで,網羅的かつ定量的にリボソーム衝突を調べることができる(Disome-Seq).一方で,従来のMitoRibo-Seqではスクロース密度勾配超遠心によって55S(ミトコンドリアモノソーム)を単離するため,ミトコンドリアダイソームの解析はこれまで行うことができなかった(図7A).MitoIPでは,スクロース密度勾配超遠心のようにミトコンドリアダイソームの除去は行わないため,RNase後に生じた50~80塩基長のRNA断片を精製・増幅し(図7B),次世代シークエンス解析が可能である(MitoIP-Thor-Disome-Seq)47).
MitoIP-Thor-Disome-Seqによって,ミトコンドリア転写産物上でリボソーム衝突が普遍的に発生していることが明らかにされた(図7C).実際,mt-mRNAの特定の位置において,ミトコンドリアダイソームが生じやすいことが示された47).これらの位置は,特定の膜貫通ドメイン(TMD)の合成完了部位に割り当てられる(図7C).それゆえ,TMDの膜貫通前に合成が完了してしまうことを防ぐため,ミトコンドリアリボソームが一時休止することを示唆している.さらに,このような翻訳休止点は,細胞種間での保存性が高いことから47),共翻訳膜挿入の忠実性を保つためにプログラムされたチェックポイントである可能性がある.
引用文献References
1) Martijn, J., Vosseberg, J., Guy, L., Offre, P., & Ettema, T.J.G. (2018) Deep mitochondrial origin outside the sampled alphaproteobacteria. Nature, 557, 101–105.
2) Kummer, E. & Ban, N. (2021) Mechanisms and regulation of protein synthesis in mitochondria. Nat. Rev. Mol. Cell Biol., 22, 307–325.
3) Schon, K.R., Ratnaike, T., van den Ameele, J., Horvath, R., & Chinnery, P.F. (2020) Mitochondrial diseases: A diagnostic revolution. Trends Genet., 36, 702–717.
4) Suomalainen, A. & Battersby, B.J. (2018) Mitochondrial diseases: The contribution of organelle stress responses to pathology. Nat. Rev. Mol. Cell Biol., 19, 77–92.
5) Pearce, S., Nezich, C.L., & Spinazzola, A. (2013) Mitochondrial diseases: Translation matters. Mol. Cell. Neurosci., 55, 1–12.
6) Rubalcava-Gracia, D., Bubb, K., Levander, F., Burr, S.P., August, A.V., Chinnery, P.F., Koolmeister, C., & Larsson, N.-G. (2024) LRPPRC and SLIRP synergize to maintain sufficient and orderly mammalian mitochondrial translation. Nucleic Acids Res., 52, 11266–11282.
7) Apostolopoulos, A. & Iwasaki, S. (2022) Into the matrix: Current methods for mitochondrial translation studies. J. Biochem., 171, 379–387.
8) Garreau de Loubresse, N., Prokhorova, I., Holtkamp, W., Rodnina, M.V., Yusupova, G., & Yusupov, M. (2014) Structural basis for the inhibition of the eukaryotic ribosome. Nature, 513, 517–522.
9) Wong, W., Bai, X.-C., Brown, A., Fernandez, I.S., Hanssen, E., Condron, M., Tan, Y.H., Baum, J., & Scheres, S.H.W. (2014) Cryo-EM structure of the Plasmodium falciparum 80S ribosome bound to the anti-protozoan drug emetine. eLife, 3, e03080.
10) Chomyn, A. (1996) In vivo labeling and analysis of human mitochondrial translation products. Methods Enzymol., 264, 197–211.
11) Sasarman, F. & Shoubridge, E.A. (2012) Radioactive labeling of mitochondrial translation products in cultured cells. Methods Mol. Biol., 837, 207–217.
12) Barrientos, A., Korr, D., & Tzagoloff, A. (2002) Shy1p is necessary for full expression of mitochondrial COX1 in the yeast model of Leigh’s syndrome. EMBO J., 21, 43–52.
13) Gouget, K., Verde, F., & Barrientos, A. (2008) In vivo labeling and analysis of mitochondrial translation products in budding and in fission yeasts. Methods Mol. Biol., 457, 113–124.
14) Beatty, K.E., Liu, J.C., Xie, F., Dieterich, D.C., Schuman, E.M., Wang, Q., & Tirrell, D.A. (2006) Fluorescence visualization of newly synthesized proteins in mammalian cells. Angew. Chem. Int. Ed., 45, 7364–7367.
15) Beatty, K.E. & Tirrell, D.A. (2008) Two-color labeling of temporally defined protein populations in mammalian cells. Bioorg. Med. Chem. Lett., 18, 5995–5999.
16) Roche, F.K., Marsick, B.M., & Letourneau, P.C. (2009) Protein synthesis in distal axons is not required for growth cone responses to guidance cues. J. Neurosci., 29, 638–652.
17) Dieterich, D.C., Hodas, J.J.L., Gouzer, G., Shadrin, I.Y., Ngo, J.T., Triller, A., Tirrell, D.A., & Schuman, E.M. (2010) In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat. Neurosci., 13, 897–905.
18) Tcherkezian, J., Brittis, P.A., Thomas, F., Roux, P.P., & Flanagan, J.G. (2021) Transmembrane receptor DCC associates with protein synthesis machinery and regulates translation. Cell, 184, 2520.
19) Hinz, F.I., Dieterich, D.C., Tirrell, D.A., & Schuman, E.M. (2012) Non-canonical amino acid labeling in vivo to visualize and affinity purify newly synthesized proteins in larval zebrafish. ACS Chem. Neurosci., 3, 40–49.
20) Yoon, B.C., Jung, H., Dwivedy, A., O’Hare, C.M., Zivraj, K.H., & Holt, C.E. (2012) Local translation of extranuclear lamin B promotes axon maintenance. Cell, 148, 752–764.
21) Signer, R.A.J., Magee, J.A., Salic, A., & Morrison, S.J. (2014) Haematopoietic stem cells require a highly regulated protein synthesis rate. Nature, 509, 49–54.
22) Zhang, X., Zuo, X., Yang, B., Li, Z., Xue, Y., Zhou, Y., Huang, J., Zhao, X., Zhou, J., Yan, Y., et al. (2014) MicroRNA directly enhances mitochondrial translation during muscle differentiation. Cell, 158, 607–619.
23) Estell, C., Stamatidou, E., El-Messeiry, S., & Hamilton, A. (2017) In situ imaging of mitochondrial translation shows weak correlation with nucleoid DNA intensity and no suppression during mitosis. J. Cell Sci., 130, 4193–4199.
24) Yousefi, R., Fornasiero, E.F., Cyganek, L., Montoya, J., Jakobs, S., Rizzoli, S.O., Rehling, P., & Pacheu-Grau, D. (2021) Monitoring mitochondrial translation in living cells. EMBO Rep., 22, e51635.
25) Zorkau, M., Albus, C.A., Berlinguer-Palmini, R., Chrzanowska-Lightowlers, Z.M.A., & Lightowlers, R.N. (2021) High-resolution imaging reveals compartmentalization of mitochondrial protein synthesis in cultured human cells. Proc. Natl. Acad. Sci. USA, 118, e2008778118.
26) Kimura, Y., Saito, H., Osaki, T., Ikegami, Y., Wakigawa, T., Ikeuchi, Y., & Iwasaki, S. (2022) Mito-FUNCAT-FACS reveals cellular heterogeneity in mitochondrial translation. RNA, 28, 895–904.
27) Fung, S., Nishimura, T., Sasarman, F., & Shoubridge, E.A. (2013) The conserved interaction of C7orf30 with MRPL14 promotes biogenesis of the mitochondrial large ribosomal subunit and mitochondrial translation. Mol. Biol. Cell, 24, 184–193.
28) Antonicka, H., Sasarman, F., Nishimura, T., Paupe, V., & Shoubridge, E.A. (2013) The mitochondrial RNA-binding protein GRSF1 localizes to RNA granules and is required for posttranscriptional mitochondrial gene expression. Cell Metab., 17, 386–398.
29) Pearce, S.F., Rorbach, J., Van Haute, L., D’Souza, A.R., Rebelo-Guiomar, P., Powell, C.A., Brierley, I., Firth, A.E., & Minczuk, M. (2017) Maturation of selected human mitochondrial tRNAs requires deadenylation. eLife, 6, e27596.
30) Lee, M., Matsunaga, N., Akabane, S., Yasuda, I., Ueda, T., & Takeuchi-Tomita, N. (2021) Reconstitution of mammalian mitochondrial translation system capable of correct initiation and long polypeptide synthesis from leaderless mRNA. Nucleic Acids Res., 49, 371–382.
31) Lee, M. & Takeuchi-Tomita, N. (2023) Reconstitution of Mammalian Mitochondrial Translation System Capable of Long Polypeptide Synthesis. Methods Mol. Biol., 2661, 233–255.
32) Ingolia, N.T., Ghaemmaghami, S., Newman, J.R.S., & Weissman, J.S. (2009) Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science, 324, 218–223.
33) Ingolia, N.T. (2014) Ribosome profiling: New views of translation, from single codons to genome scale. Nat. Rev. Genet., 15, 205–213.
34) Iwasaki, S., Floor, S.N., & Ingolia, N.T. (2016) Rocaglates convert DEAD-box protein eIF4A into a sequence-selective translational repressor. Nature, 534, 558–561.
35) Suzuki, T., Yashiro, Y., Kikuchi, I., Ishigami, Y., Saito, H., Matsuzawa, I., Okada, S., Mito, M., Iwasaki, S., Ma, D., et al. (2020) Complete chemical structures of human mitochondrial tRNAs. Nat. Commun., 11, 4269.
36) Kashiwagi, K., Shichino, Y., Osaki, T., Sakamoto, A., Nishimoto, M., Takahashi, M., Mito, M., Weber, F., Ikeuchi, Y., Iwasaki, S., et al. (2021) eIF2B-capturing viral protein NSs suppresses the integrated stress response. Nat. Commun., 12, 7102.
37) Rooijers, K., Loayza-Puch, F., Nijtmans, L.G., & Agami, R. (2013) Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nat. Commun., 4, 2886.
38) Gao, F., Wesolowska, M., Agami, R., Rooijers, K., Loayza-Puch, F., Lawless, C., Lightowlers, R.N., & Chrzanowska-Lightowlers, Z.M.A. (2017) Using mitoribosomal profiling to investigate human mitochondrial translation. Wellcome Open Res., 2, 116.
39) Morscher, R.J., Ducker, G.S., Li, S.H.-J., Mayer, J.A., Gitai, Z., Sperl, W., & Rabinowitz, J.D. (2018) Mitochondrial translation requires folate-dependent tRNA methylation. Nature, 554, 128–132.
40) Schöller, E., Marks, J., Marchand, V., Bruckmann, A., Powell, C.A., Reichold, M., Mutti, C.D., Dettmer, K., Feederle, R., Hüttelmaier, S., et al. (2021) Balancing of mitochondrial translation through METTL8-mediated m3C modification of mitochondrial tRNAs. Mol. Cell, 81, 4810–4825.e12.
41) Li, S.H.-J., Nofal, M., Parsons, L.R., Rabinowitz, J.D., & Gitai, Z. (2021) Monitoring mammalian mitochondrial translation with MitoRiboSeq. Nat. Protoc., 16, 2802–2825.
42) Soto, I., Couvillion, M., Hansen, K.G., McShane, E., Moran, J.C., Barrientos, A., & Churchman, L.S. (2022) Balanced mitochondrial and cytosolic translatomes underlie the biogenesis of human respiratory complexes. Genome Biol., 23, 170.
43) Brischigliaro, M., Krüger, A., Moran, J.C., Antonicka, H., Ahn, A., Shoubridge, E.A., Rorbach, J., & Barrientos, A. (2024) The human mitochondrial translation factor TACO1 alleviates mitoribosome stalling at polyproline stretches. Nucleic Acids Res., 52, 9710–9726.
44) Singh, V., Moran, J.C., Itoh, Y., Soto, I.C., Fontanesi, F., Couvillion, M., Huynen, M.A., Churchman, L.S., Barrientos, A., & Amunts, A. (2024) Structural basis of LRPPRC–SLIRP-dependent translation by the mitoribosome. Nat. Struct. Mol. Biol., Online ahead of print.
45) Couvillion, M.T., Soto, I.C., Shipkovenska, G., & Churchman, L.S. (2016) Synchronized mitochondrial and cytosolic translation programs. Nature, 533, 499–503.
46) Couvillion, M.T. & Churchman, L.S. (2017) Mitochondrial ribosome (mitoribosome) profiling for monitoring mitochondrial translation in vivo. Curr. Protoc. Mol. Biol., 119, 4.28.1–4.28.25.
47) Wakigawa, T., Mito, M., Yamashiro, H., Tomuro, K., Tani, H., Tomizawa, K., Chujo, T., Nagao, A., Suzuki, T., Wei, F.-Y., et al. (2024) Complexity and dynamics of in organello translation landscape assessed by high-resolution mitochondrial ribosome profiling. bioRxiv 2023, 2023.07.19.549812.
48) Mito, M., Shichino, Y., & Iwasaki, S. (2023) Thor-Ribo-Seq: Ribosome profiling tailored for low input with RNA-dependent RNA amplification. bioRxiv 2023, 2023.01.15.524129.
49) Suzuki, T., Nagao, A., & Suzuki, T. (2011) Human mitochondrial tRNAs: Biogenesis, function, structural aspects, and diseases. Annu. Rev. Genet., 45, 299–329.
50) Nakano, S., Suzuki, T., Kawarada, L., Iwata, H., Asano, K., & Suzuki, T. (2016) NSUN3 methylase initiates 5-formylcytidine biogenesis in human mitochondrial tRNA(Met). Nat. Chem. Biol., 12, 546–551.
51) Asano, K., Suzuki, T., Saito, A., Wei, F.-Y., Ikeuchi, Y., Numata, T., Tanaka, R., Yamane, Y., Yamamoto, T., Goto, T., et al. (2018) Metabolic and chemical regulation of tRNA modification associated with taurine deficiency and human disease. Nucleic Acids Res., 46, 1565–1583.
52) Haag, S., Sloan, K.E., Ranjan, N., Warda, A.S., Kretschmer, J., Blessing, C., Hübner, B., Seikowski, J., Dennerlein, S., Rehling, P., et al. (2016) NSUN3 and ABH1 modify the wobble position of mt-tRNAMet to expand codon recognition in mitochondrial translation. EMBO J., 35, 2104–2119.
53) Van Haute, L., Dietmann, S., Kremer, L., Hussain, S., Pearce, S.F., Powell, C.A., Rorbach, J., Lantaff, R., Blanco, S., Sauer, S., et al. (2016) Deficient methylation and formylation of mt-tRNAMet wobble cytosine in a patient carrying mutations in NSUN3. Nat. Commun., 7, 1–10.
54) Kawarada, L., Suzuki, T., Ohira, T., Hirata, S., Miyauchi, K., & Suzuki, T. (2017) ALKBH1 is an RNA dioxygenase responsible for cytoplasmic and mitochondrial tRNA modifications. Nucleic Acids Res., 45, 7401–7415.
55) Takemoto, C., Spremulli, L.L., Benkowski, L.A., Ueda, T., Yokogawa, T., & Watanabe, K. (2009) Unconventional decoding of the AUA codon as methionine by mitochondrial tRNAMet with the anticodon f5CAU as revealed with a mitochondrial in vitro translation system. Nucleic Acids Res., 37, 1616–1627.
56) Suzuki, T., Suzuki, T., Wada, T., Saigo, K., & Watanabe, K. (2002) Taurine as a constituent of mitochondrial tRNAs: New insights into the functions of taurine and human mitochondrial diseases. EMBO J., 21, 6581–6589.
57) Umeda, N., Suzuki, T., Yukawa, M., Ohya, Y., Shindo, H., Watanabe, K., & Suzuki, T. (2005) Mitochondria-specific RNA-modifying enzymes responsible for the biosynthesis of the wobble base in mitochondrial tRNAs. Implications for the molecular pathogenesis of human mitochondrial diseases. J. Biol. Chem., 280, 1613–1624.
58) Fakruddin, M., Wei, F.-Y., Suzuki, T., Asano, K., Kaieda, T., Omori, A., Izumi, R., Fujimura, A., Kaitsuka, T., Miyata, K., et al. (2018) Defective mitochondrial tRNA taurine modification activates global proteostress and leads to mitochondrial disease. Cell Rep., 22, 482–496.
59) Peng, G.-X., Zhang, Y., Wang, Q.-Q., Li, Q.-R., Xu, H., Wang, E.-D., & Zhou, X.-L. (2021) The human tRNA taurine modification enzyme GTPBP3 is an active GTPase linked to mitochondrial diseases. Nucleic Acids Res., 49, 2816–2834.
60) Tuorto, F., Legrand, C., Cirzi, C., Federico, G., Liebers, R., Müller, M., Ehrenhofer-Murray, A.E., Dittmar, G., Gröne, H.-J., & Lyko, F. (2018) Queuosine-modified tRNAs confer nutritional control of protein translation. EMBO J., 37, e99777.
61) Chen, Y.-C., Kelly, V.P., Stachura, S.V., & Garcia, G.A. (2010) Characterization of the human tRNA-guanine transglycosylase: Confirmation of the heterodimeric subunit structure. RNA, 16, 958–968.
62) Goto, Y.-I., Nonaka, I., & Horai, S. (1990) A mutation in the tRNA(Leu)(UUR) gene associated with the MELAS subgroup of mitochondrial encephalomyopathies. Nature, 348, 651–653.
63) Kobayashi, Y., Momoi, M.Y., Tominaga, K., Momoi, T., Nihei, K., Yanagisawa, M., Kagawa, Y., & Ohta, S. (1990) A point mutation in the mitochondrial tRNA(Leu)(UUR) gene in MELAS (mitochondrial myopathy, encephalopathy, lactic acidosis and stroke-like episodes). Biochem. Biophys. Res. Commun., 173, 816–822.
64) Goto, Y., Nonaka, I., & Horai, S. (1991) A new mtDNA mutation associated with mitochondrial myopathy, encephalopathy, lactic acidosis and stroke-like episodes (MELAS). Biochim. Biophys. Acta Mol. Basis Dis., 1097, 238–240.
65) Yasukawa, T., Suzuki, T., Ueda, T., Ohta, S., & Watanabe, K. (2000) Modification defect at anticodon wobble nucleotide of mitochondrial tRNAs(Leu)(UUR) with pathogenic mutations of mitochondrial myopathy, encephalopathy, lactic acidosis, and stroke-like episodes. J. Biol. Chem., 275, 4251–4257.
66) Kirino, Y., Goto, Y.-I., Campos, Y., Arenas, J., & Suzuki, T. (2005) Specific correlation between the wobble modification deficiency in mutant tRNAs and the clinical features of a human mitochondrial disease. Proc. Natl. Acad. Sci. USA, 102, 7127–7132.
67) Tomoda, E., Nagao, A., Shirai, Y., Asano, K., Suzuki, T., Battersby, B.J., & Suzuki, T. (2023) Restoration of mitochondrial function through activation of hypomodified tRNAs with pathogenic mutations associated with mitochondrial diseases. Nucleic Acids Res., 51, 7563–7579.
68) Sasarman, F., Antonicka, H., & Shoubridge, E.A. (2008) The A3243G tRNALeu(UUR) MELAS mutation causes amino acid misincorporation and a combined respiratory chain assembly defect partially suppressed by overexpression of EFTu and EFG2. Hum. Mol. Genet., 17, 3697–3707.
69) Suzuki, T. (2021) The expanding world of tRNA modifications and their disease relevance. Nat. Rev. Mol. Cell Biol., 22, 375–392.
70) Pechmann, S., Chartron, J.W., & Frydman, J. (2014) Local slowdown of translation by nonoptimal codons promotes nascent-chain recognition by SRP in vivo. Nat. Struct. Mol. Biol., 21, 1100–1105.
71) Jia, L., Dienhart, M., Schramp, M., McCauley, M., Hell, K., & Stuart, R.A. (2003) Yeast Oxa1 interacts with mitochondrial ribosomes: The importance of the C-terminal region of Oxa1. EMBO J., 22, 6438–6447.
72) Szyrach, G., Ott, M., Bonnefoy, N., Neupert, W., & Herrmann, J.M. (2003) Ribosome binding to the Oxa1 complex facilitates co-translational protein insertion in mitochondria. EMBO J., 22, 6448–6457.
73) Haque, M.E., Elmore, K.B., Tripathy, A., Koc, H., Koc, E.C., & Spremulli, L.L. (2010) Properties of the C-terminal tail of human mitochondrial inner membrane protein Oxa1L and its interactions with mammalian mitochondrial ribosomes. J. Biol. Chem., 285, 28353–28362.
74) Pfeffer, S., Woellhaf, M.W., Herrmann, J.M., & Förster, F. (2015) Organization of the mitochondrial translation machinery studied in situ by cryoelectron tomography. Nat. Commun., 6, 6019.
75) Englmeier, R., Pfeffer, S., & Förster, F. (2017) Structure of the human mitochondrial ribosome studied in situ by cryoelectron tomography. Structure, 25, 1574–1581.e2.
76) Itoh, Y., Andréll, J., Choi, A., Richter, U., Maiti, P., Best, R.B., Barrientos, A., Battersby, B.J., & Amunts, A. (2021) Mechanism of membrane-tethered mitochondrial protein synthesis. Science, 371, 846–849.
77) Richter-Dennerlein, R., Oeljeklaus, S., Lorenzi, I., Ronsör, C., Bareth, B., Schendzielorz, A.B., Wang, C., Warscheid, B., Rehling, P., & Dennerlein, S. (2016) Mitochondrial protein synthesis adapts to influx of nuclear-encoded protein. Cell, 167, 471–483.e10.
78) Arpat, A.B., Liechti, A., De Matos, M., Dreos, R., Janich, P., & Gatfield, D. (2020) Transcriptome-wide sites of collided ribosomes reveal principles of translational pausing. Genome Res., 30, 985–999.
79) Han, P., Shichino, Y., Schneider-Poetsch, T., Mito, M., Hashimoto, S., Udagawa, T., Kohno, K., Yoshida, M., Mishima, Y., Inada, T., et al. (2020) Genome-wide Survey of Ribosome Collision. Cell Rep., 31, 107610.
80) Meydan, S. & Guydosh, N.R. (2020) Disome and trisome profiling reveal genome-wide targets of ribosome quality control. Mol. Cell, 79, 588–602.e6.
81) Tuck, A.C., Rankova, A., Arpat, A.B., Liechti, L.A., Hess, D., Iesmantavicius, V., Castelo-Szekely, V., Gatfield, D., & Bühler, M. (2020) Mammalian RNA decay pathways are highly specialized and widely linked to translation. Mol. Cell, 77, 1222–1236.e13.
82) Zhao, T., Chen, Y.-M., Li, Y., Wang, J., Chen, S., Gao, N., & Qian, W. (2021) Disome-seq reveals widespread ribosome collisions that promote cotranslational protein folding. Genome Biol., 22, 16.
83) Fujita, T., Yokoyama, T., Shirouzu, M., Taguchi, H., Ito, T., & Iwasaki, S. (2022) The landscape of translational stall sites in bacteria revealed by monosome and disome profiling. RNA, 28, 290–302.
84) Weraarpachai, W., Antonicka, H., Sasarman, F., Seeger, J., Schrank, B., Kolesar, J.E., Lochmüller, H., Chevrette, M., Kaufman, B.A., Horvath, R., et al. (2009) Mutation in TACO1, encoding a translational activator of COX I, results in cytochrome c oxidase deficiency and late-onset Leigh syndrome. Nat. Genet., 41, 833–837.
85) Richman, T.R., Spåhr, H., Ermer, J.A., Davies, S.M.K., Viola, H.M., Bates, K.A., Papadimitriou, J., Hool, L.C., Rodger, J., Larsson, N.-G., et al. (2016) Loss of the RNA-binding protein TACO1 causes late-onset mitochondrial dysfunction in mice. Nat. Commun., 7, 11884.
86) Cruz-Zaragoza, L.D., Dennerlein, S., Linden, A., Yousefi, R., Lavdovskaia, E., Aich, A., Falk, R.R., Gomkale, R., Schöndorf, T., Bohnsack, M.T., et al. (2021) An in vitro system to silence mitochondrial gene expression. Cell, 184, 5824–5837.e15.