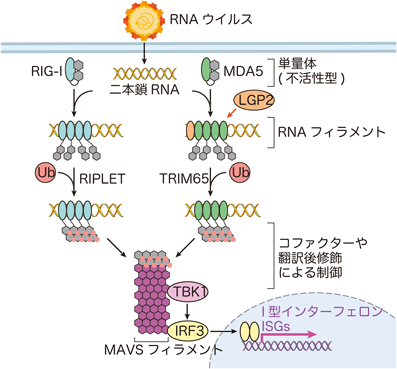
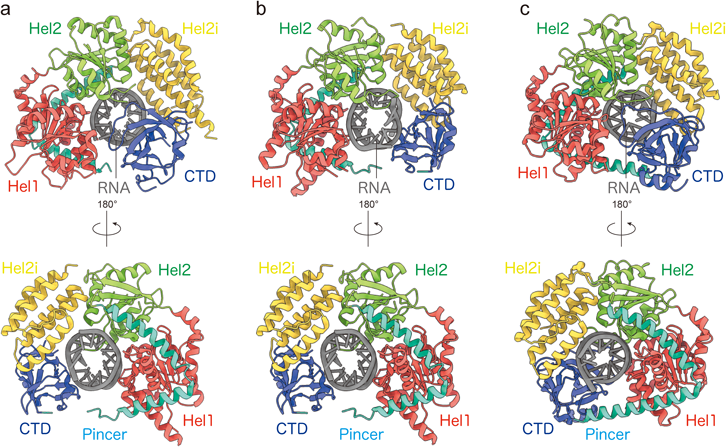
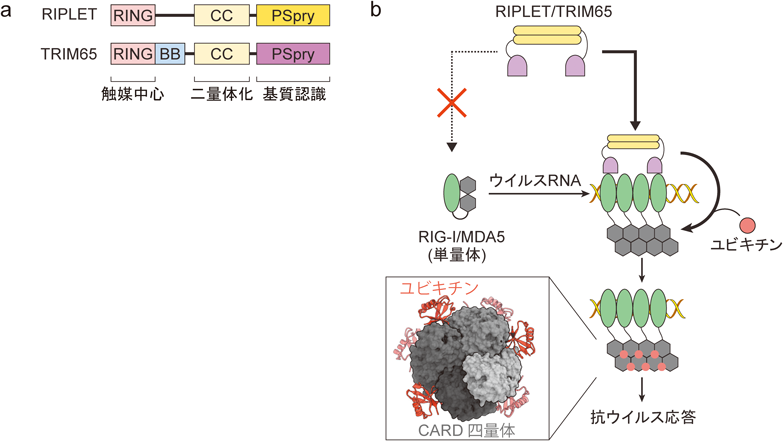
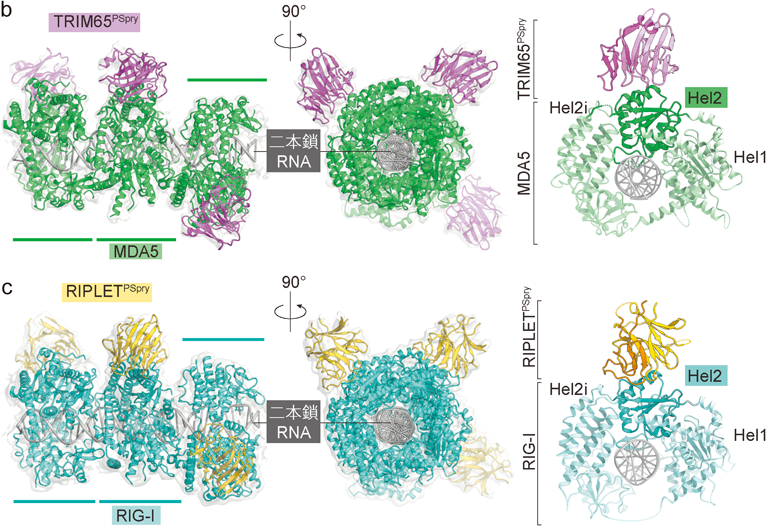
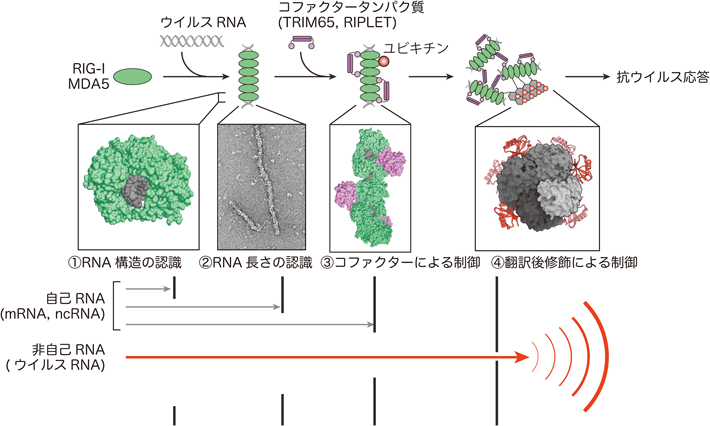
RIG-I様受容体による自己・非自己RNAの認識と抗ウイルス応答Self vs non-self discrimination by RIG-I like receptors in the antiviral immune response
東京科学大学統合研究院免疫機構学研究ユニットInstitute of Science Tokyo, Institute of Integrated Research, Mechanistic Immunology Research Unit ◇ 〒113–8510 東京都文京区湯島1–5–45 3号館9階 ◇ 1–5–45 Yushima building3, 9th floor, Bunkyo-ku, Tokyo 113–8510, Japan