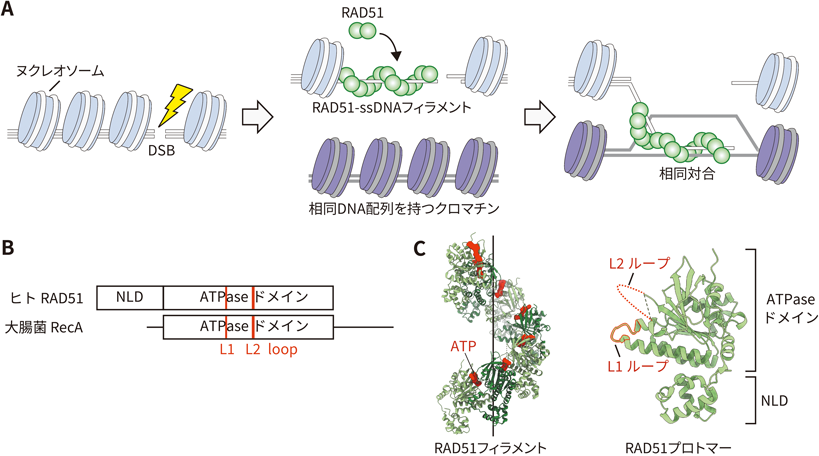
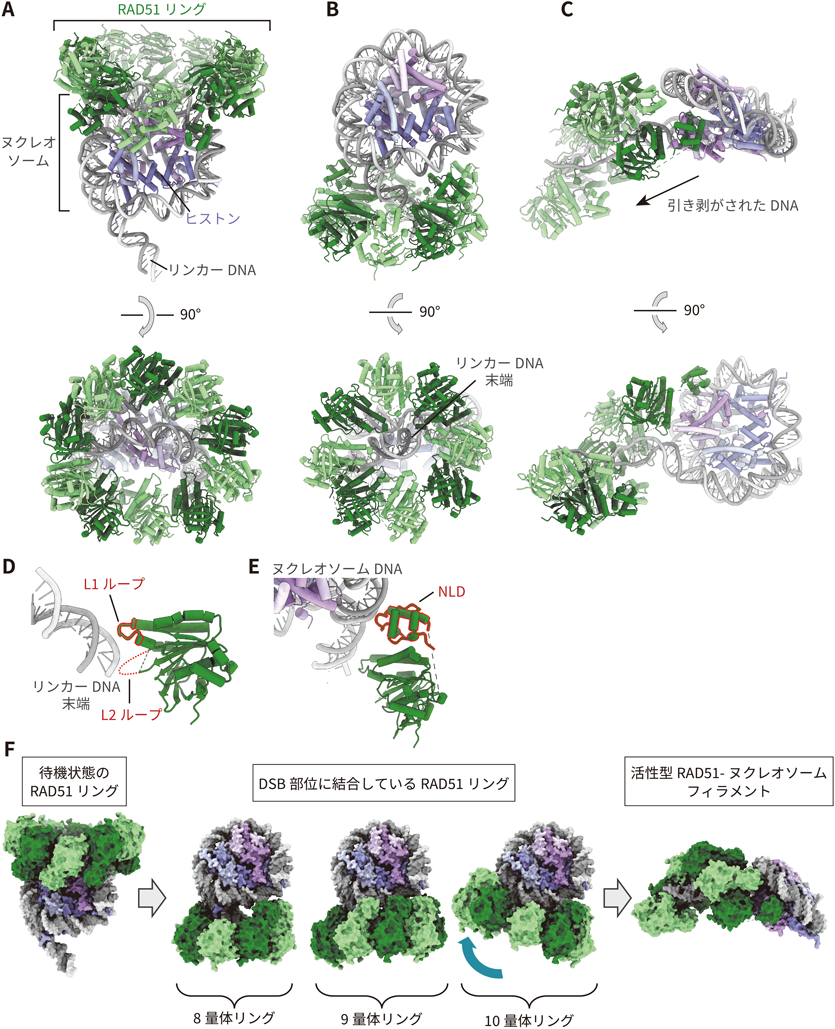
RAD51によるクロマチン上での相同組換え開始機構Molecular mechanism of RAD51-mediated homologous recombination initiation on chromatin
1 東京大学定量生命科学研究所クロマチン構造機能研究分野Laboratory of Chromatin Structure and Function, Institute for Quantitative Biosciences, The University of Tokyo ◇ 〒113‒0032 東京都文京区弥生1‒1‒1 生命科学総合研究棟B棟303 ◇ Life Sciences Research Building B303, 1–1–1 Yayoi, Bunkyo-ku, Tokyo 113–0032, Japan
2 東京大学大学院理学系研究科生物科学専攻Department of Biological Sciences, Graduate School of Science, The University of Tokyo ◇ 〒113–0033 東京都文京区本郷7–3–1 ◇ 7–3–1 Hongo, Bunkyo-ku, Tokyo 113–0033, Japan