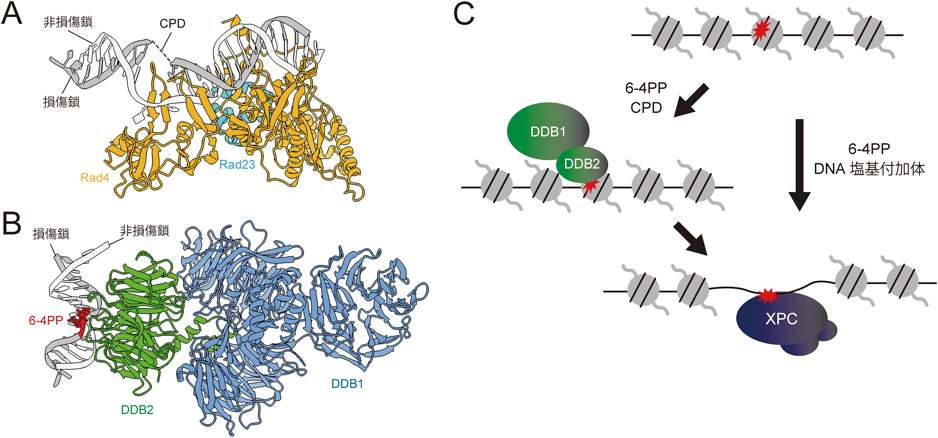
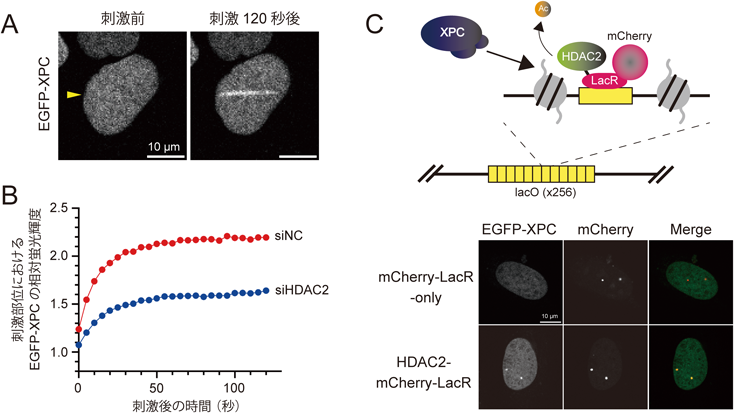
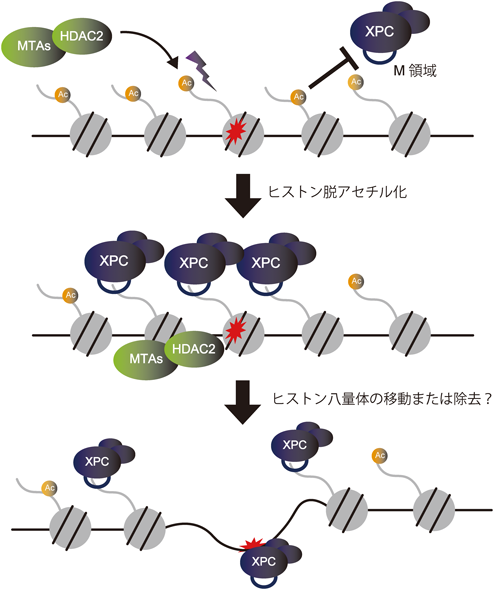
ヌクレオチド除去修復のDNA損傷認識を制御するクロマチンダイナミクスChromatin dynamics regulating DNA damage recognition process of nucleotide excision repair
神戸大学バイオシグナル総合研究センター/神戸大学大学院理学研究科生物学専攻Biosignal Research Center, Kobe University/Department of Biology, Graduate School of Science, Kobe University ◇ 〒657–8501 兵庫県神戸市灘区六甲台町1–1 ◇ 1–1 Rokkodai-cho, Nada-ku, Kobe, Hyogo 657–8501, Japan