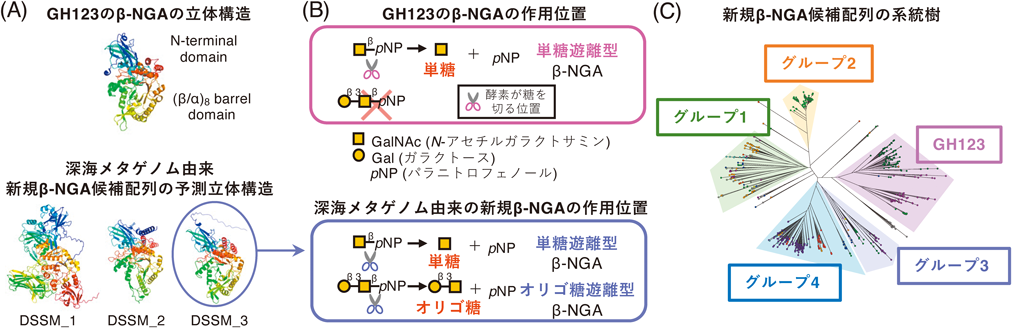
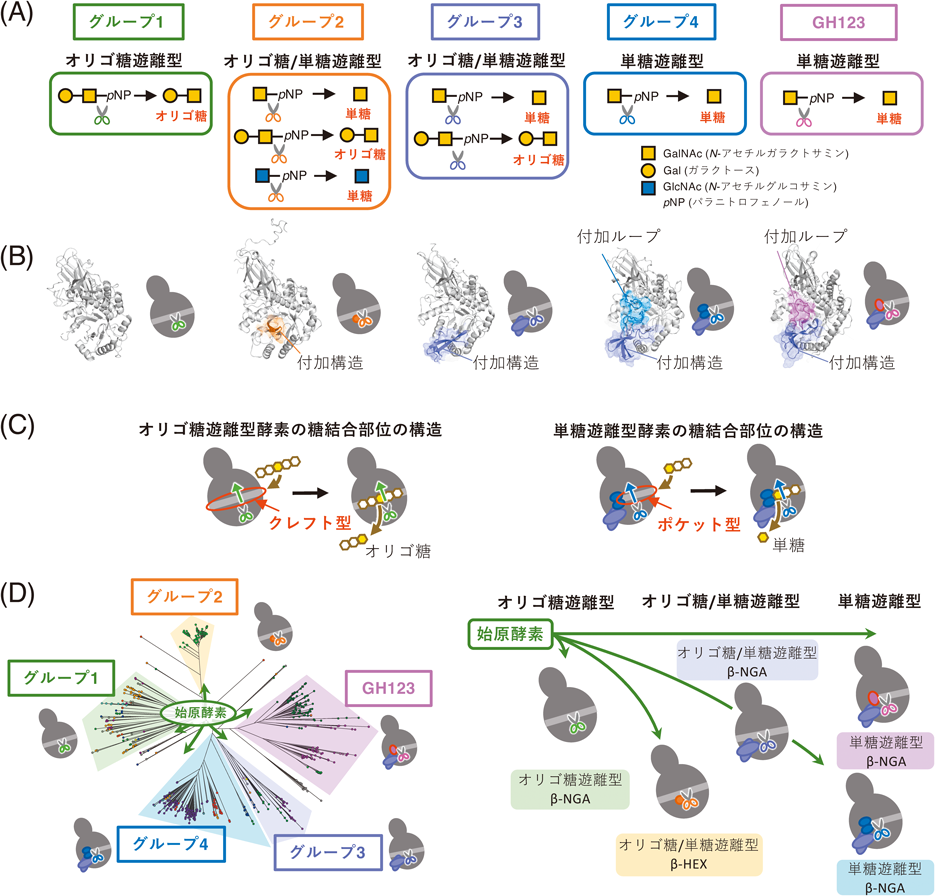
深海微生物のメタゲノム解析が切り拓く未知の酵素の世界~β-GalNAcに作用する新規酵素群の分子進化の歴史をひもとく~Unveiling the molecular history of β-N-acetylgalactosamine-targeting glycosidases
海洋研究開発機構(JAMSTEC)Japan Agency for Marine-Earth Science and Technology (JAMSTEC) ◇ 〒237–0061 神奈川県横須賀市夏島町2番地15 ◇ 2–15 Natsushima-cho, Yokosuka, Kanagawa 237–0061, Japan