生物はDNAから転写されるmRNAにコードされた遺伝情報に基づいて,20種のアミノ酸からタンパク質を合成する.ほとんどの生物が共通の遺伝暗号表(表1)を用いていることは,すべての生物は単一の細胞から進化したとする生物一元説の大きな根拠になっている.いくつかの生物やオルガネラでは変則的な遺伝暗号が使われている1)が,これは進化的に後から獲得したと考えられている.このような遺伝暗号表がどのような仕組みで成立したかについては,アミノ酸とそれをコードするRNAの間に何らかの物理的な相互作用があったとする立体化学説(stereochemical theory)2)や,遺伝暗号は進化の過程で偶然決まったものでそのまま凍結したとする偶然凍結説(frozen accident theory)が提唱3)されているが,実験的な検証は難しく,いまだに決着がついていない.
表1 遺伝暗号表(コドン表)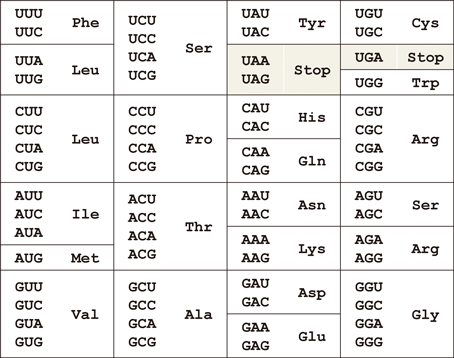 |
| 真正細菌,アーキア,真核生物のほとんどすべての生物に共通であることから普遍暗号表ともいわれる. |
コドンは4種類の塩基が三つの並びで定義されるため64種類あるが,終止コドンを除く61通りのコドンを20種類のアミノ酸に割り当てるために,遺伝暗号表は多対一の対応関係になっている(表1).MetとTrpのみ1種類のコドン,それぞれAUGとUGGにより指定されているが,他のアミノ酸は2~6通りのコドンにより指定されている.これらは同義コドン(synonymous codon)と呼ばれる.遺伝暗号表をみると,コドンの1字目と2字目の塩基の並びがアミノ酸を指定しており,3字目はどの塩基(N=A, G, U, C)であっても同じアミノ酸を指定する(ファミリーボックス),ピリミジン(Y=U, C)かプリン(R=A, G)かによって指定するアミノ酸が決まる(2コドンセット)というルールがある.このルールにより多対一の対応関係が成り立っている.Arg, Leu, Serは例外的に,ファミリーボックスと2コドンセットからなる六つの同義コドンを持つ.Ileは三つの同義コドン,すなわちAUUとAUCの2コドンセットに加えてAUAを持つ.
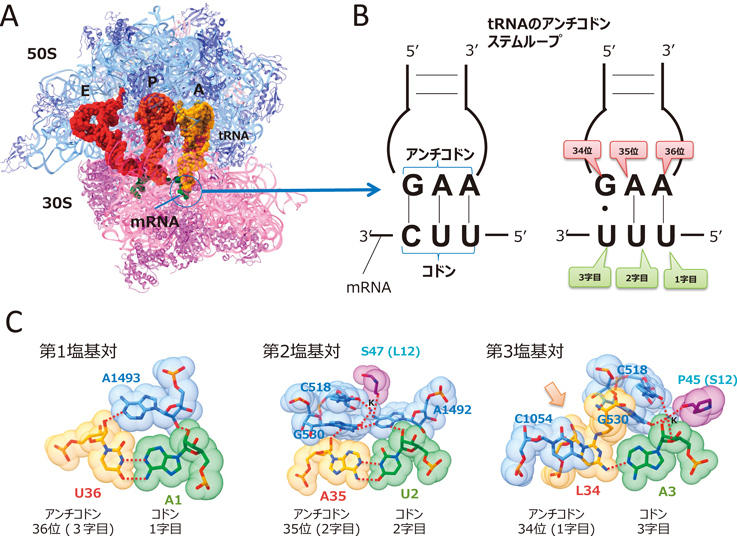
遺伝暗号表の成り立ちは,リボソーム上において,mRNAのコドンがtRNAのアンチコドンによって認識される仕組みによって説明できる.tRNAはタンパク質合成においてコドンを解読しアミノ酸へと変換するアダプター分子であり,多様な化学修飾を持つことが特徴である4).特に,アンチコドン領域にはさまざまな修飾がみられ,これらの修飾はtRNAがアミノ酸を受容するアミノアシル化や,リボソーム上において,mRNAのコドンを解読する際に重要な役割を担う.リボソームにはtRNAが結合する三つの部位,Aサイト,Pサイト,Eサイトが存在し,コドンの解読はAサイトで行われる(図1A).アミノ酸を受容したアミノアシルtRNAがGタンパク質である伸長因子(EF-TuやeEF1α)と結合し,リボソームのAサイトに運ばれる.アンチコドンによるコドンの認識は,リボソーム小サブユニットの暗号解読中心で行われる.コドン1字目はアンチコドン3字目(36位)と,コドン2字目はアンチコドン2字目(35位)とワトソンクリック型の塩基対合によって正確に認識されなければならない(図1B).なぜならば,リボソームのAサイトにおいて,これらの塩基対合をリボソームRNA(rRNA)の保存塩基(A1492, A1493, G530)が副溝側から認識し(Aマイナー相互作用),コドン–アンチコドンの正確な対合をセンシングする仕組みがあるからである5)(図1C).この相互作用により,リボソームの小サブユニットがopen型からclose型へ変化し,この構造変化がアミノアシルtRNAと結合している伸長因子に伝わり,GTPが加水分解されて暗号解読が成立する.ところが,リボソームのAサイトにおいて,コドン3字目とアンチコドン1字目(34位)の塩基対合(通称wobble位)の周辺は比較的スペースが空いており(図1C),塩基が自由に動ける状況になっている.したがって,コドン3字目とアンチコドン1字目は,G–U塩基対に代表されるいわゆるwobble対合をはじめとする非標準的な塩基対合が許容される(図1B,図2A).この位置の塩基対合のあいまいさこそが,複数のコドンが1種類のtRNAに解読される基盤原理であり,ひいてはコドンとアミノ酸が多対一の対応関係になっている所以である.このような仕組みは,限られたtRNA種で複数のコドンを経済的に解読するために適していると考えられる.tRNAのアンチコドン1字目にはしばしば修飾された塩基が存在し,この修飾がwobble対合を微妙に制御することで,遺伝暗号が正確に解読されている4).
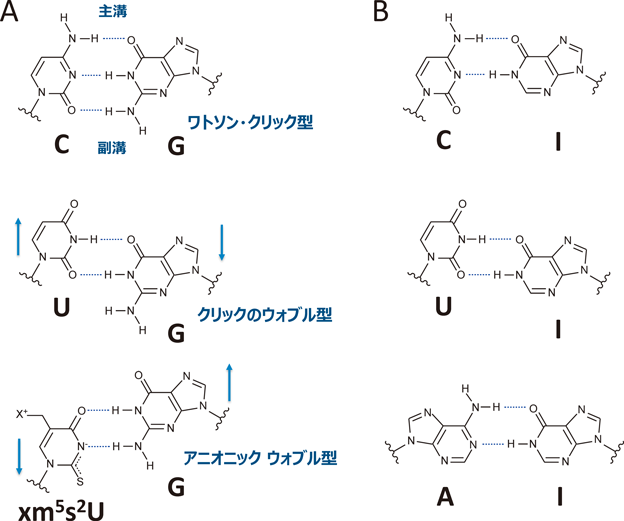
Francis Crickは1966年にwobble仮説を発表し,アンチコドン1字目(34位)はコドン3字目とワトソンクリック型の塩基対合(WC対合)以外に,非標準的な塩基対合が可能であることを提唱した6).実際に,アンチコドン1字目のG34はコドン3字目のC以外にUを解読し,U34はコドン3字目のA以外にGを読むとされている(図1B).G–UやU–Gの塩基対合はwobble対合と呼ばれ,WC対合と比較して,Uが主構側へGが副溝側へ移動するようなジオメトリーをとる(図2A).G–UのWobble対合はアンチコドン1字目とコドン3字目の間でみられる典型的な塩基対合(図1B)である6).アンチコドン1字目に存在する多様なtRNA修飾は,コドン認識能を拡張したり,制限したり,さらに認識するコドンを変化させるなど,タンパク質合成において,非常に重要な役割を担っている(図3)4).
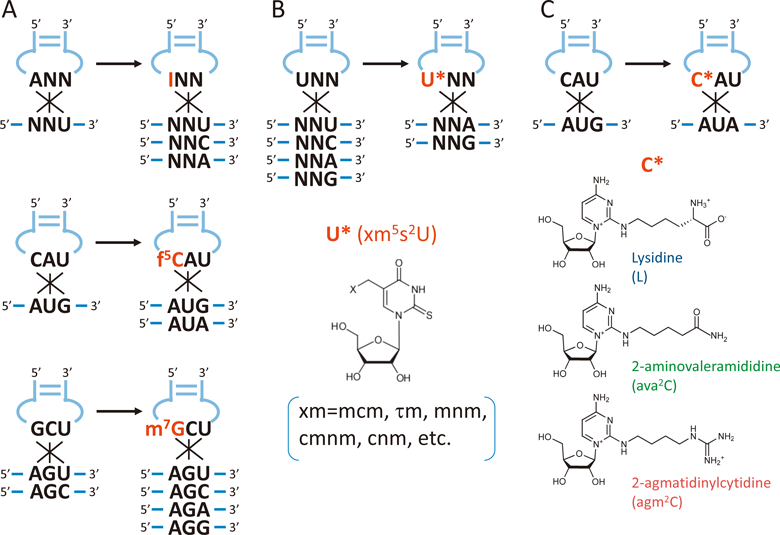
イノシン(I)(図2B)はアデノシンの脱アミノ化反応で生じるRNA修飾であり,真核生物から細菌において,さまざまなtRNAのアンチコドン1字目にみられる7).Iは化学的な性質がグアノシンに類似しているため,I34はコドン3字目のCとUと対合するが,さらにAとも対合することが知られている(図2B, 3A)6).したがって,A34がコドン3字目のUとのみ対合すると考えると,I34への修飾によりコドン解読能が拡張される(図3A).真核生物において,同義コドンのファミリーボックスにはI34を持つtRNAが割り当てられているのが一般的である.細菌においてtRNA特異的なアデノシンデアミナーゼTadAが,A34からI34へ修飾を触媒する8).ヒトでは,ADAT2–ADAT3複合体が同様の反応を触媒する9).これらのタンパク質は生存に必須であり,I34が遺伝暗号を解読する上で重要なtRNA修飾であることを示している.
5-ホルミルシチジン(f5C)は,動物ミトコンドリアtRNAMetのアンチコドンに存在する修飾である10)(図3A).動物ミトコンドリアではAUAコドンがMetに暗号変化しているため,f5C34は,AUGコドンに加え,AUAコドンもMetとして解読するために重要な役割を担っている(図3A).実際に,f5C34はAUAコドン3字目のAとの対合を促進することが知られている11).我々を含む複数の研究グループにより,f5C34がメチル化酵素NSUN3と酸化酵素ALKBH1による複数の反応プロセスを介して生合成されることが示された12, 13).NSUN3およびALKBH1遺伝子が欠損すると,ミトコンドリアのタンパク質合成が著しく減少し,呼吸およびミトコンドリア活性が低下することから,f5C34はミトコンドリアの機能に必須である.また,f5C34の欠損はミトコンドリア病の原因にもなっている14).
7-メチルグアノシン(m7G)はヒトデやウニなどの棘皮動物やイカや貝などの軟体動物のミトコンドリアtRNASerのアンチコドン1字目にみられる修飾である15–17).これらの生物のミトコンドリアでは,AGNの四つのコドンすべてがセリンを指定するように暗号変化している.もともとAGYコドンに対応するtRNASerがm7G34を獲得することで,AGRコドンの認識も可能になると考えられている(図3A).したがって,m7G34はAGYからAGNへコドン認識を拡張する修飾である.
Crickはwobble仮説において,U34はコドン3字目のAとGを認識することを提唱した6)が,その後の研究でU34はコドン3字目の4種類すべての塩基と対合可能であるとする4-way wobble理論が提唱された(図3B)18, 19).実際に,マイコプラズマなどの細菌やミトコンドリアの遺伝暗号解読系において,ファミリーボックスを解読するtRNAはU34を持つことが知られている.
一方で,コドン3字目がプリンの2コドンセット(NNR)に対応するtRNAは修飾されたU34を持つことが知られている(図3B)4).それらは,ウラシル塩基の5位の炭素に側鎖が結合した5-メチルウリジン(xm5U)誘導体やさらに2位のカルボニル酸素が硫黄に置換した2-チオウリジン(xm5s2U)誘導体が知られている.真核生物では,5-メトキシカルボニルメチルウリジン(mcm5U)およびその2-チオウリジン誘導体であるmcm5s2Uが細胞質のtRNAに存在する.細菌では5-メチルアミノメチルウリジン(mnm5U)や5-カルボキシアミノメチルウリジン(cmnm5U)とそれらの2-チオウリジン誘導体であるmnm5s2Uやcmnm5s2Uが用いられる.アーキアでは5-シアノメチルウリジン(cnm5U)とその2-チオウリジン誘導体cnm5s2Uが見つかっている.一方,ミトコンドリアtRNAには,5-タウリノメチルウリジン(τm5U)やcmnm5Uさらには,それらの2-チオウリジン誘導体であるτm5s2Uやcmnm5s2Uが使われている.また,しばしばリボースの2′-水酸基がメチル化された2′-O-メチルウリジン(Um)の誘導体も使われる.2-チオ基や2′-O-メチル基は立体反発の効果により,リボースのパッカリングをC3′endo型に偏らせる役割があり,コドン3字目がピリミジンの2コドンセット(NNY)の誤認識を防ぐ機能が知られている20, 21).最近の研究によると,xm5s2Uの5位の置換基や2-チオ基には,ウラシルN3原子のpKaを低下する役割がある22)ことが判明し,xm5s2Uはアニオニックな互変異性体をとることで,コドン3字目のGとアニオニックU–G塩基対を形成することがわかった23, 24)(図2A).この塩基対は,CrickのU–G wobble塩基対と異なり,Uが副溝側へGが主溝側へ移動するようなジオメトリーをとる.アニオニックU–Gのジオメトリーは,隣の塩基対(コドン2字目とアンチコドン2字目)とスタッキング相互作用することで,コドン–アンチコドン対合の安定化に寄与すると考えられる.
61種類のセンスコドンの中で,AUAコドンは最も解読が難しい遺伝暗号であると考えられる.なぜならば,3字目がプリンの2コドンセットであるNNRは基本的に同じアミノ酸を指定しているが,AUAはIle, AUGはMetと異なるアミノ酸を指定しているからである(表1).NNRコドンが同じアミノ酸を指定するのは,アンチコドン1字目にUまたはU*(ウリジン修飾体)を有するtRNAがNNAとNNGの両方を解読するからである(図3B).もし,tRNAIleがAUAコドンと相補的なアンチコドンであるUAU(またはU*AU)を持つとしたら,AUAコドンのみならずAUGコドンをもIleに翻訳してしまうであろう.したがって,生物はAUAコドンを特異的に解読するための巧妙な仕組みを獲得している.以下に述べるように,AUAコドンをどのように解読するかは,真核生物,細菌,アーキアのそれぞれの生物界で異なっている.共通しているのは,tRNAIleのアンチコドン1字目に修飾塩基を用いている点である25)(図3C).
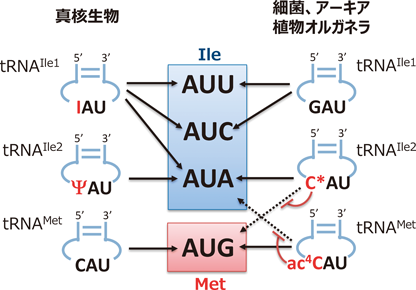
真核生物tRNAIleはI34を用いることで,AUU, AUC, AUAの三つのコドンを経済的に解読する(図4)が,アンチコドン1字目にシュードウリジン(Ψ)を持つ,もう一つのtRNAIleが存在し,Ψ34により,AUAコドンを二つのtRNAを用いて冗長的に解読すると考えられている(図4).
一方で,ほとんどすべての細菌は,AUUとAUCのみを解読するtRNAIle1とAUAコドンに対応するtRNAIle2の二つのtRNAで読み分けている.tRNAIle2のアンチコドン1字目にはCの修飾体であるライシジン(lysidine:L)26)を用いている(図3C, 4).Lはリシンのεアミノ基がシトシン環のC2炭素に結合した修飾塩基(図3C)であり,この修飾により,Gとは対合できずにAと対合する性質を獲得する(図3C).また,L修飾はtRNAの転写後に酵素的に導入されるが,修飾される前のtRNAIleの前駆体はCAUアンチコドンを持ち,あたかもtRNAMetのように振る舞い,AUGコドンを解読するだけでなく,実際にメチオニルtRNA合成酵素に認識され,Metを受容することが知られている.したがって,C34からL34へのたった一つの修飾によって,コドン認識がAUGからAUAに変化するだけでなく,アミノ酸受容能もMetからIleへと変化する27).前述した動物ミトコンドリアでみられる修飾シチジンであるf5CはAUAおよびAUGの二つのコドンを解読するのに対し,L34はAUAコドンのみを解読するという違いがある.筆者らは,tRNAIleにL修飾を導入するライシジン合成酵素(tRNAIle lysidine synthetase:TilS)を発見した28, 29).tilS遺伝子は必須遺伝子であることから,L修飾はAUAコドンの解読に必須であることが,生化学的かつ分子遺伝学的にも判明している28, 29).
細菌tRNAMetのアンチコドン1字目にはN4-アセチルシチジン(ac4C)修飾がある.生化学的な実験によって,ac4C修飾はtRNAMetがAUAコドンを誤翻訳するのを防ぐ役割があることが示されていた30)(図4).筆者らはac4C修飾酵素として,RNAアセチル化酵素であるTmcA31)およびTmcAL32)を発見した.これらの遺伝子を欠損し,ac4C34がない状況で,L34合成酵素TilSの発現を抑制すると,AUAコドンがtRNAMetによって高頻度で誤翻訳されることが判明した32).したがって,tRNAMetのac4C34とtRNAIle2のL34は協調的にAUAコドンとAUGコドンの読み分けを行っていることが明らかとなった(図4).
一方,アーキアにはTilSのホモログが存在しないが,AUAコドンに対応するtRNAIle遺伝子のアンチコドン1字目にはC34を持つため,L34とは異なるシチジン修飾が存在すると考えられてきた.筆者らはアーキアtRNAIleから,アルギニンの代謝物であるアグマチンがシトシン2位に結合したアグマチジン(agm2C)を発見した(図3C)33).また比較ゲノム法により,agm2C34の修飾酵素であるTiaSを発見した33).TiaSはTCKドメインを持つキナーゼであり,ATPピロホスファターゼファミリーのTilSとは,まったく異なるタンパク質ファミリーであることから,アーキアと真正細菌の共通祖先からそれぞれの生物界に分岐する過程で,独立にagm2CとLという異なる修飾シチジンを獲得したことが示唆される.agm2CとLは長い側鎖と末端に極性基を持つという点で類似した化学構造を持ち,同様の仕組みでAUAコドンを解読するとしたら,分子レベルでの収斂進化と捉えることができる25).
植物のオルガネラであるミトコンドリアと葉緑体のtRNAIleも何らかのシチジン修飾を利用してAUAコドンをIleに解読すると推定されてきたが,その化学構造は不明であった34).最近,筆者らは植物の葉緑体およびミトコンドリアから単離したtRNAIleに含まれる塩基修飾を質量分析法およびNMRにより調べ,アンチコドン1字目に2-アミノバレラミジジン(ava2C)(図3C)と命名した新規の修飾体を発見した35).
筆者らはこの実験でホウレンソウを実験材料に選択した.まず,ホウレンソウ40 kgを食品加工会社に送付し,フリーズドライ処理後に粉砕し,1.5 kgのホウレンソウパウダーを得た.この粉体からフェノール処理(30 L)により14.4 gのRNA画分を抽出した.陰イオン交換クロマトグラフィーを用いて低分子RNA画分を濃縮したのち,当研究室で開発された往復循環クロマトグラフィーを用いて葉緑体tRNAIleを3.26 mg得た.精製したtRNAをヌクレオシドに分解し,HPLCにより目的のシチジン修飾体を得た.質量分析,重水素交換,NMRなど各種解析を行い,候補となる修飾シチジンava2Cの化学構造(図3C)を推定した.最終的にava2C標品を有機化学的に合成し,ホウレンソウ由来の修飾体と同一であることを確認した.
ava2Cはシトシン2位に5-アミノバレラミドが結合した化学構造を持つ(図3C).ava2Cは,長い側鎖を持つ点でLやagm2Cに類似しているが,側鎖の末端はアミド基である点が特徴である(図3C).他の生物についても調べたところ,シロイヌナズナやタバコなどの高等植物に加え,原始的な藻類である紅藻のほか,意外なことに緑膿菌やコレラ菌など一部の病原性の細菌にもava2Cが見つかった.Lやagm2Cと同様で,ava2CはtRNAIle2がIleを受容するために必要であり,さらにAUAコドンの解読に欠かせない修飾であることが判明した.ava2Cを持つ生物にはTilSホモログが存在し,ava2CはLを経由して生合成されることが判明しているが,この変換を触媒する酵素は見つかっていない.
8. シチジン修飾によるAUAコドン認識の分子基盤
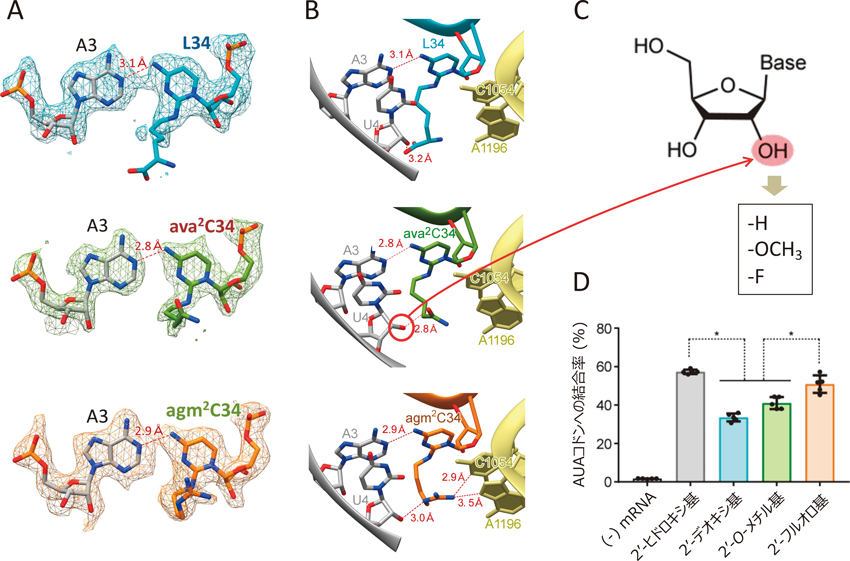
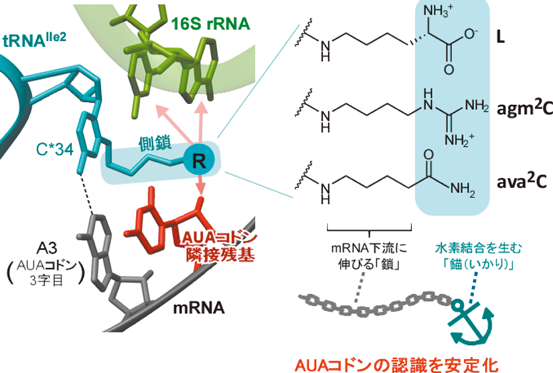
AUAコドンを解読するために必要なtRNA修飾として,L34, ava2C34, agm2C34の3種類のシチジン修飾が見つかっている(図3C).いずれもシトシンの2位にアミノ基を介して長いアルキル鎖とその末端には極性の官能基を持つ,類似した化学構造を有している.これらの構造的な特徴がどのようにしてAUAコドンの解読を可能にするかを明らかにするために,クライオ電子顕微鏡を用いて,リボソーム上におけるコドン–アンチコドン相互作用の構造を可視化した.試料としては,L34, ava2C34, agm2C34それぞれの修飾を持つtRNAIle2を大腸菌,Pseudomonas putida, Haloarcula marismortuiから単離精製した.大腸菌リボソームにAUAコドンを持つmRNAをセットし,それぞれのtRNAと複合体を形成し,グリッドを作製した.クライオ電子顕微鏡(300 kV)を用い,2.3~2.7 Åの高分解能での構造解析に成功した35, 36).リボソームのAサイトにおいて,L34, ava2C34, agm2C34のシトシン環はいずれも,AUAコドン3字目のアデニン(A3)と1本の水素結合を介したC–A塩基対を形成することが判明した(図5A).この対合様式においては,シチジン修飾側鎖が,AUGコドン3字目のグアニン(G3)と空間的にぶつかるため,AUGコドンの誤認識を妨ぐと考えられた.さらに,シチジン修飾の側鎖はいずれもmRNAの下流(3′側)に向かって伸び,その末端の極性官能基がAUAコドンに隣接する残基のリボース2′-ヒドロキシ基と追加的に水素結合を形成することが判明した(図5B).さらにagm2Cは末端のグアニジノ基を介してA部位周辺のrRNAとも水素結合を作り,コドン–アンチコドン対合をさらに安定化していることが示唆された(図5B).これらの修飾末端による相互作用は,C–A塩基対の弱い対合を補強する役割があると考えられる.
L34, ava2C34, agm2C34の修飾側鎖とmRNAの相互作用が,実際にAUAコドン認識に影響を与えるかを調べるため,AUAコドンの3′隣接残基を置換した化学合成mRNAを用いて,リボソーム上におけるtRNAIle2のAUAコドン結合効率を評価した(図5C).3′隣接残基のリボース2′-水酸基を,2′-デオキシ基および2′-O-メチル基に置換すると,tRNAIle2のAUAコドンへの結合率が顕著に低下した(図5D).一方で,同じ位置を2′-フルオロ基に置換すると天然のmRNA同様に,AUAコドンによく結合することが判明した(図5D).2′-デオキシ基や2′-O-メチル基はシチジン修飾の極性基との水素結合を阻害し,2′-フルオロ基は水素結合のアクセプターとして働くため,極性基との水素結合が可能になると考えられる.これらの結果は,シチジン修飾の極性側鎖とコドンに隣接するmRNA残基との相互作用によって,tRNAによるAUAコドンの解読が効率よく行われることを強く示唆している.
3種類のシチジン修飾が,共通の機構でAUAコドンを認識することが判明し,筆者らは鎖と錨モデル(chain and anchor model)を提唱した(図6).mRNAの下流に伸びる長い側鎖部分を「鎖」,周囲のmRNAやrRNAと水素結合を作る末端の極性基を「錨」にたとえると,tRNAがリボソームに錨を下ろして,AUAコドンを認識しているように捉えることができる.これまでの研究で,tRNA修飾がコドン以外のmRNA残基と相互作用する前例はなく,tRNA修飾が持つ知られざるポテンシャルを提唱する発見となった.
9. キューオシン修飾とその誘導体による翻訳速度の調節
キューオシン(Q)は7-デアザグアノシン骨格とシクロペンテン環を持つグアノシンの誘導体で,細菌からヒトに至る多くの生物のアンチコドン1字目にみられるユニークなtRNA修飾である37)(図7).Q34は遺伝暗号表の3列目のNNYの2コドンセットに対応するTyr, His, Asn, Aspの4種類のtRNAにみられる.
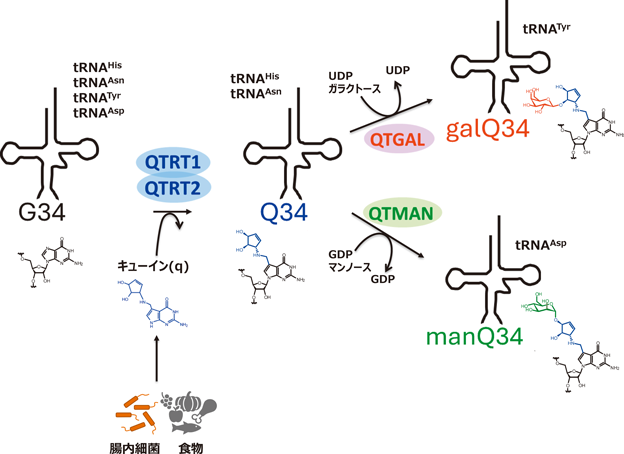
細菌はQの塩基部分であるキューイン(q)を生合成するが,ヒトや脊椎動物はqを自前で生合成することができず,腸内細菌や食物から摂取したqを栄養素として腸管から体内へと取り込み,tRNAのQ34修飾に利用する37)(図7).ヒトでは,QTRT1とQTRT2の複合体がトランスグリコシラーゼとして機能し,tRNAのアンチコドン1字目のグアノシンの塩基部分をqにつけ替えることでQ修飾を形成する.ヒトや脊椎動物では,4種類の細胞質tRNAがQ修飾を持つが,Tyrに対応するtRNATyrはQ34にガラクトースが付加されたガラクトシルキューオシン(galQ)に変換され,アスパラギン酸(Asp)に対応するtRNAAspではQ34にマンノースが付加されたマンノシルキューオシン(manQ)に変換される(図7).galQやmanQは1976年に西村暹博士らによって発見され38),tRNA修飾の中で糖が付与されたユニークな修飾体として注目されているが,その生合成や機能は約半世紀もの間謎に包まれていた.
筆者らの研究グループは,ラットおよびブタ肝臓の抽出液から複数のカラムクロマトグラフィーを用いた生化学的手法により,tRNATyrのQ34にガラクトースを転移する酵素(B3GNTL1)を同定し,キューオシンtRNAガラクトース転移酵素(QTGAL)(図7)と命名した39).さらに,tRNAAspのQ34にマンノースを転移する酵素(GTDC1)を同定し,キューオシンtRNAマンノース転移酵素(QTMAN)(図7)と命名した39).QTGALとQTMANはそれぞれUDPガラクトースおよびGDPマンノースを基質として,galQとmanQを生合成する.
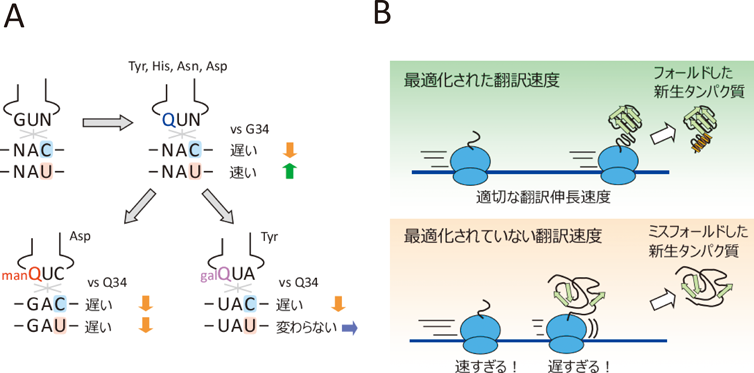
QTGALとQTMANに加え,QTRT1とQTRT2の各遺伝子をHEK293T細胞でノックアウト(KO)し,糖付加QやQ修飾を欠損させた.リボソームプロファイリングを用いて,これらのKO細胞について,コドンごとの翻訳速度を計測した39).その結果,G34と比較して,Q34は3字目がUのコドンの翻訳速度を速める働きがある一方で,3字目がCのコドンの翻訳速度を減弱する役割があることがわかった(図8A).さらに,tRNATyrのQ34とgalQ34の比較で,ガラクトシル化はUACコドンの翻訳速度を遅くする働きがあることが判明した(図8A).また,tRNAAspのQ34とmanQ34の比較で,マンノシル化はGACとGAU両コドンの翻訳速度を遅くすることがわかった(図9).一般的にtRNA修飾は翻訳速度を促進する働きがあるが,Qの糖付加は翻訳速度を抑制することが判明した初めてのtRNA修飾である.また,tRNATyrは潜在的に終止コドンであるUAGとUAAを読み飛ばすことが知られているが,galQのガラクトース付加修飾には終止コドンの読み飛ばしを防止する役割があることも判明した.
一般に,tRNA修飾は適切な翻訳速度の維持に重要な役割を担い,tRNA修飾が欠損すると,新生タンパク質のフォールディングに異常が生じることが知られている(図8B)40).各KO細胞にレポーター遺伝子を導入し,タンパク質の凝集度を計測したところ,野生型の細胞と比較して,いずれのKO細胞においても凝集したタンパク質が蓄積したことから,糖付加QおよびQ修飾は適切な翻訳速度を調節することで,プロテオスタシスの維持に重要な役割を担っていることが判明した39).実際にゼブラフィッシュのQtgalおよびQtmanの変異体は,野生型と比較して生後の成長速度が顕著に遅く,体長が短いという表現型を示したことから,糖付加Q修飾はゼブラフィッシュの健全な生育に必要であることが示された39).現在,筆者らはノックアウトマウスを用いて,哺乳動物における糖付加Q修飾の生理機能を探求している.
10. キューオシン修飾とその誘導体によるコドン認識の構造基盤
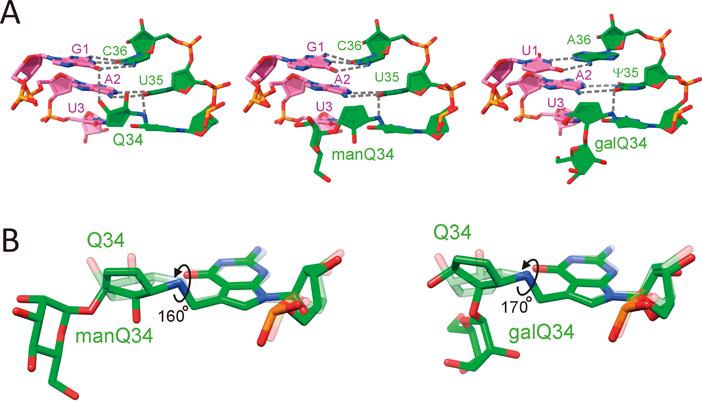
筆者らはクライオ電子顕微鏡を用いて,ヒト80SリボソームとtRNA複合体の構造解析を行い,Q34および糖付加Q34がどのようにコドンを認識するかを視覚化した39).その結果,Q34のシクロペンテン環はジオール基による水素結合を介してコドン–アンチコドン対合が形成する二重らせんの主溝に結合し,主溝結合基(major groove binder)として機能することで,コドン認識の効率を上げていることが明らかとなった(図9A).実際に,シクロペンテン環はコドン1, 2字目とアンチコドン3, 2字目の塩基対合を主溝側から水素結合で認識している様子が観察できる.特にこの効果はコドン3字目がUを持つ,UAUやGAUコドンの際に効果が高いことも判明した.Q34が3字目にUを持つコドンを認識する際に,Q–Uのwobble対合のジオメトリーをとるが,この際にQの塩基は副溝側に移動するため,シクロペンテン環が主構により接近する.逆にQ–Cでは,Qの塩基が主溝側に1.9 Å移動するため,シクロペンテン環は主構から外れ,主溝結合基としての効果が弱まる.実際にQ–Cペアでのシクロペンテン環の密度はかなり弱く,フレキシブルであることが示唆された.
一方で,galQ34およびmanQ34も主溝結合基としての機能を持つ(図9A)が,ガラクトースやマンノースはシクロペンテン環の水酸基に付加するため,コドンを認識する際に,シクロペンテン環がそれぞれ,170°と160°回転する(図9B).この回転により,シクロペンテン環と主構との水素結合ができなくなるため,主溝結合基としての効果が弱まると考えられる.以上の構造的な知見は,Qの糖付加が翻訳速度を抑制するという,リボソームプロファイリングの結果と一致している.
なぜtRNAには多種多様な修飾が存在するのか? 個々のtRNA修飾がどのような仕組みでコドンを識別し,また翻訳速度を調節するのか? 生命科学における新しい解析手法の発展により,この古典的な課題が少しずつ明らかになっている.クライオ電子顕微鏡によるリボソーム複合体の高分解能な構造解析は,コドン–アンチコドン対合を原子レベルで視覚化し,tRNA修飾のこれまでに知られていない機能が解明されている.リボソームプロファイリングの技術も大きく貢献しており,コドンごとにグローバルな翻訳速度を測定することができる.適切な翻訳速度の維持は,mRNAの安定性に関わり,また新生タンパク質の正しいフォールディングに必要である.tRNA修飾はさまざまな生物学的文脈において,ダイナミックに変動し,グローバルな遺伝子発現に大きな影響を及ぼしているであろう.今後もこの分野の発展に貢献していきたい.
謝辞Acknowledgments
本総説で紹介した最近の研究は,研究室のスタッフと学生たちの成果である.また多くの共同研究者にサポートしていただいた.彼らの絶大な貢献に対してお礼を申し上げたい.特に,ava2Cの発見には主に宮内健常さん(博士研究員)と木村聡さん(現コーネル大)が貢献した.鎖と錨モデルは秋山奈穂さん(大学院生)の成果である.糖付加Qの研究は,当時大学院生であった趙雪薇(Xuewei Zhao)さんと馬丁(Ding Ma)さんが,それぞれQTGALとQTMANを同定したところから,研究が始まった.また,cryo-EMによる糖付加Q修飾の構造解析は石黒健介さん(特任助教)の成果である.また石黒さんには本総説の図の作成を手伝っていただいた.この場を借りて感謝申し上げたい.
引用文献References
1) Suzuki, T. & Nagao, A. (2021) Genetic code and its variations. eLS, 2, 147–157.
2) Woese, C.R. (1967) The Genetic Code: The Molecular Basis for Genetic Expression, Harper & Row, New York.
3) Crick, F.H. (1968) The origin of the genetic code. J. Mol. Biol., 38, 367–379.
4) Suzuki, T. (2021) The expanding world of tRNA modifications and their disease relevance. Nat. Rev. Mol. Cell Biol., 22, 375–392.
5) Ogle, J.M., Brodersen, D.E., Clemons, W.M. Jr., Tarry, M.J., Carter, A.P., & Ramakrishnan, V. (2001) Recognition of cognate transfer RNA by the 30S ribosomal subunit. Science, 292, 897–902.
6) Crick, F.H. (1966) Codon--anticodon pairing: the wobble hypothesis. J. Mol. Biol., 19, 548–555.
7) Srinivasan, S., Torres, A.G., & Ribas de Pouplana, L. (2021) Inosine in biology and disease. Genes (Basel), 12, 600.
8) Wolf, J., Gerber, A.P., & Keller, W. (2002) tadA, an essential tRNA-specific adenosine deaminase from Escherichia coli. EMBO J., 21, 3841–3851.
9) Torres, A.G., Piñeyro, D., Rodríguez-Escribà, M., Camacho, N., Reina, O., Saint-Léger, A., Filonava, L., Batlle, E., & Ribas de Pouplana, L. (2015) Inosine modifications in human tRNAs are incorporated at the precursor tRNA level. Nucleic Acids Res., 43, 5145–5157.
10) Moriya, J., Yokogawa, T., Wakita, K., Ueda, T., Nishikawa, K., Crain, P.F., Hashizume, T., Pomerantz, S.C., McCloskey, J.A., Kawai, G., et al. (1994) A novel modified nucleoside found at the first position of the anticodon of methionine tRNA from bovine liver mitochondria. Biochemistry, 33, 2234–2239.
11) Takemoto, C., Spremulli, L.L., Benkowski, L.A., Ueda, T., Yokogawa, T., & Watanabe, K. (2009) Unconventional decoding of the AUA codon as methionine by mitochondrial tRNAMet with the anticodon f5CAU as revealed with a mitochondrial in vitro translation system. Nucleic Acids Res., 37, 1616–1627.
12) Kawarada, L., Suzuki, T., Ohira, T., Hirata, S., Miyauchi, K., & Suzuki, T. (2017) ALKBH1 is an RNA dioxygenase responsible for cytoplasmic and mitochondrial tRNA modifications. Nucleic Acids Res., 45, 7401–7415.
13) Nakano, S., Suzuki, T., Kawarada, L., Iwata, H., Asano, K., & Suzuki, T. (2016) NSUN3 methylase initiates 5-formylcytidine biogenesis in human mitochondrial tRNA(Met). Nat. Chem. Biol., 12, 546–551.
14) Van Haute, L., Dietmann, S., Kremer, L., Hussain, S., Pearce, S.F., Powell, C.A., Rorbach, J., Lantaff, R., Blanco, S., Sauer, S., et al. (2016) Deficient methylation and formylation of mt-tRNA(Met) wobble cytosine in a patient carrying mutations in NSUN3. Nat. Commun., 7, 12039.
15) Matsuyama, S., Ueda, T., Crain, P.F., McCloskey, J.A., & Watanabe, K. (1998) A novel wobble rule found in starfish mitochondria. Presence of 7-methylguanosine at the anticodon wobble position expands decoding capability of tRNA. J. Biol. Chem., 273, 3363–3368.
16) Tomita, K., Ueda, T., & Watanabe, K. (1998) 7-Methylguanosine at the anticodon wobble position of squid mitochondrial tRNA(Ser)GCU: molecular basis for assignment of AGA/AGG codons as serine in invertebrate mitochondria. Biochim. Biophys. Acta Gene Struct. Expr., 1399, 78–82.
17) Ohira, T., Suzuki, T., Miyauchi, K., Suzuki, T., Yokobori, S.I., Yamagishi, A., & Watanabe, K. (2013) Decoding mechanism of non-universal genetic codes in Loligo bleekeri mitochondria. J. Biol. Chem., 288, 7645–7652.
18) Barrell, B.G., Anderson, S., Bankier, A.T., de Bruijn, M.H., Chen, E., Coulson, A.R., Drouin, J., Eperon, I.C., Nierlich, D.P., Roe, B.A., et al. (1980) Different pattern of codon recognition by mammalian mitochondrial tRNAs. Proc. Natl. Acad. Sci. USA, 77, 3164–3166.
19) Suzuki, T., Nagao, A., & Suzuki, T. (2011) Human mitochondrial tRNAs: biogenesis, function, structural aspects, and diseases. Annu. Rev. Genet., 45, 299–329.
20) Yokoyama, S., Watanabe, T., Murao, K., Ishikura, H., Yamaizumi, Z., Nishimura, S., & Miyazawa, T. (1985) Molecular mechanism of codon recognition by tRNA species with modified uridine in the first position of the anticodon. Proc. Natl. Acad. Sci. USA, 82, 4905–4909.
21) Prusiner, P., Yathindra, N., & Sundaralingam, M. (1974) Effect of ribose O(2′)-methylation on the conformation of nucleosides and nucleotides. Biochim. Biophys. Acta Nucleic Acids Protein Synth., 366, 115–123.
22) Sochacka, E., Lodyga-Chruscinska, E., Pawlak, J., Cypryk, M., Bartos, P., Ebenryter-Olbinska, K., Leszczynska, G., & Nawrot, B. (2017) C5-substituents of uridines and 2-thiouridines present at the wobble position of tRNA determine the formation of their keto-enol or zwitterionic forms—a factor important for accuracy of reading of guanosine at the 3-end of the mRNA codons. Nucleic Acids Res., 45, 4825–4836.
23) Westhof, E., Watson, Z.L., Zirbel, C.L., & Cate, J.H.D. (2023) Anionic G*U pairs in bacterial ribosomal rRNAs. RNA, 29, 1069–1076.
24) Rozov, A., Demeshkina, N., Khusainov, I., Westhof, E., Yusupov, M., & Yusupova, G. (2016) Novel base-pairing interactions at the tRNA wobble position crucial for accurate reading of the genetic code. Nat. Commun., 7, 10457.
25) Suzuki, T. & Numata, T. (2014) Convergent evolution of AUA decoding in bacteria and archaea. RNA Biol., 11, 1586–1596.
26) Muramatsu, T., Yokoyama, S., Horie, N., Matsuda, A., Ueda, T., Yamaizumi, Z., Kuchino, Y., Nishimura, S., & Miyazawa, T. (1988) A novel lysine-substituted nucleoside in the first position of the anticodon of minor isoleucine tRNA from Escherichia coli. J. Biol. Chem., 263, 9261–9267.
27) Muramatsu, T., Nishikawa, K., Nemoto, F., Kuchino, Y., Nishimura, S., Miyazawa, T., & Yokoyama, S. (1988) Codon and amino-acid specificities of a transfer RNA are both converted by a single post-transcriptional modification. Nature, 336, 179–181.
28) Soma, A., Ikeuchi, Y., Kanemasa, S., Kobayashi, K., Ogasawara, N., Ote, T., Kato, J., Watanabe, K., Sekine, Y., & Suzuki, T. (2003) An RNA-modifying enzyme that governs both the codon and amino acid specificities of isoleucine tRNA. Mol. Cell, 12, 689–698.
29) Ikeuchi, Y., Soma, A., Ote, T., Kato, J., Sekine, Y., & Suzuki, T. (2005) molecular mechanism of lysidine synthesis that determines tRNA identity and codon recognition. Mol. Cell, 19, 235–246.
30) Stern, L. & Schulman, L.H. (1978) The role of the minor base N4-acetylcytidine in the function of the Escherichia coli noninitiator methionine transfer RNA. J. Biol. Chem., 253, 6132–6139.
31) Ikeuchi, Y., Kitahara, K., & Suzuki, T. (2008) The RNA acetyltransferase driven by ATP hydrolysis synthesizes N4-acetylcytidine of tRNA anticodon. EMBO J., 27, 2194–2203.
32) Taniguchi, T., Miyauchi, K., Sakaguchi, Y., Yamashita, S., Soma, A., Tomita, K., & Suzuki, T. (2018) Acetate-dependent tRNA acetylation required for decoding fidelity in protein synthesis. Nat. Chem. Biol., 14, 1010–1020.
33) Ikeuchi, Y., Kimura, S., Numata, T., Nakamura, D., Yokogawa, T., Ogata, T., Wada, T., Suzuki, T., & Suzuki, T. (2010) Agmatine-conjugated cytidine in a tRNA anticodon is essential for AUA decoding in archaea. Nat. Chem. Biol., 6, 277–282.
34) Francis, M.A. & Dudock, B.S. (1982) Nucleotide sequence of a spinach chloroplast isoleucine tRNA. J. Biol. Chem., 257, 11195–11198.
35) Miyauchi, K., Kimura, S., Akiyama, N., Inoue, K., Ishiguro, K., Vu, T.S., Srisuknimit, V., Koyama, K., Hayashi, G., Soma, A., et al. (2024) A tRNA modification with aminovaleramide facilitates AUA decoding in protein synthesis. Nat. Chem. Biol., 21, 522–531.
36) Akiyama, N., Ishiguro, K., Yokoyama, T., Miyauchi, K., Nagao, A., Shirouzu, M., & Suzuki, T. (2024) Structural insights into the decoding capability of isoleucine tRNAs with lysidine and agmatidine. Nat. Struct. Mol. Biol., 31, 817–825.
37) Suzuki, T., Ogizawa, A., Ishiguro, K., & Nagao, A. (2024) Biogenesis and roles of tRNA queuosine modification and its glycosylated derivatives in human health and diseases. Cell Chem. Biol., 26.
38) Kasai, H., Nakanishi, K., Macfarlane, R.D., Torgerson, D.F., Ohashi, Z., McCloskey, J.A., Gross, H.J., & Nishimura, S. (1976) Letter: The structure of Q* nucleoside isolated from rabbit liver transfer ribonucleic acid. J. Am. Chem. Soc., 98, 5044–5046.
39) Zhao, X., Ma, D., Ishiguro, K., Saito, H., Akichika, S., Matsuzawa, I., Mito, M., Irie, T., Ishibashi, K., Wakabayashi, K., et al. (2023) Glycosylated queuosines in tRNAs optimize translational rate and post-embryonic growth. Cell, 186, 5517–5535.e5524.
40) Nedialkova, D.D. & Leidel, S.A. (2015) Optimization of Codon Translation Rates via tRNA Modifications Maintains Proteome Integrity. Cell, 161, 1606–1618.
41) Taniguchi, T., Miyauchi, K., Nakane, D., Miyata, M., Muto, A., Nishimura, S., & Suzuki, T. (2013) Decoding system for the AUA codon by tRNAIle with the UAU anticodon in Mycoplasma mobile. Nucleic Acids Res., 41, 2621–2631.
著者紹介Author Profile
 鈴木 勉(すずき つとむ)
鈴木 勉(すずき つとむ)東京大学大学院工学系研究科化学生命工学専攻 教授.博士(理学).
略歴1968年東京都生まれ,96年東京工業大学大学院生命理工学研究科博士課程修了,2008年より現職.20年からJST-ERATO鈴木RNA修飾生命機能プロジェクト研究総括.
研究テーマと抱負RNA修飾が担う生命機能およびタンパク質合成のメカニズムの研究.生命が持つ分子的な側面を炙り出すような研究を意識している.
ウェブサイトhttp://rna.chem.t.u-tokyo.ac.jp/index.html
趣味水泳,スキー.