生物が誕生した太古代の海洋は,第一鉄(Fe2+)を溶存するフェルジナスな環境であったが1, 2),深海熱水噴出孔や酸素極小域(oxygen minimum zone:OMZ)などの一部の環境には硫化水素も存在していた3).シアノバクテリアの誕生による大酸化イベント(Great Oxidation Event:GOE)を端緒に,酸素量の増加とそれに伴う海洋の硫酸塩量の増加に続いて,微生物による硫酸塩の還元により,海洋は無酸素で鉄を多く含む状態から,硫化水素を多く含んだユーキシニックな環境へと変化し,原生代の海洋が形成された4).
また,生物の祖先は,深海熱水噴出孔で誕生したという説が支持されつつある5, 6).これら熱水噴出孔には,硫化水素や水素ガス,メタンガスなどが存在している.生物の祖先は,これらの分子からエネルギーを獲得する化学合成と類似したエネルギー代謝を行っていたと推定され,34.7億年前の地球ですでに微生物学的硫酸還元が行われていたことが示されている7).実際に,さまざまな化学合成細菌が深海熱水噴出孔に存在し,それらの細菌は,硫化水素やメタンもしくは水素ガスをエネルギー源および電子供与体として利用することが可能である8).
こうした地球環境の変遷や生命の誕生を考えると,硫化水素と常に隣り合わせの環境で,生物の進化と生命活動が営まれてきたといえる9).一方で,硫化水素は,その資化性を持った細菌や,それを硫黄源として同化することができる植物10, 11)以外の生物にとっては単なる毒物であるという認識が強いかもしれない.しかし,近年,硫化水素がさまざまな生物で生理活性の調節に関与することが次々と報告され,その生命素子としての重要性に注目が集まっている12).さらに,こうした生理機能の調節において,硫化水素自身がシグナル分子の本体ではなく,硫黄原子がカテネーションした構造を持つポリスルフィド化合物がシグナル分子であることがわかってきた13–15).こうしたポリスルフィド化合物は,細胞内に豊富に存在し,その強い求核作用と抗酸化作用により,活性酸素種(reactive oxygen species:ROS),フリーラジカル,求電子性物質の制御や代謝に関与している16–19).また,ミトコンドリアの電子伝達鎖の電子受容体としても働き,エネルギー代謝と密接な関係がある21, 22).このようなユニークな化学的・生化学的性質から,ポリスルフィド化合物は「超硫黄分子」と総称されている19).
本稿では,著者らが紅色光合成細菌Rhodobacter capsulatusから硫化水素依存的な光合成のマスターレギュレーターとして同定した転写因子SqrRをモデルとした,超硫黄分子検知機構の研究23–25)を中心に,超硫黄分子応答について解説する.
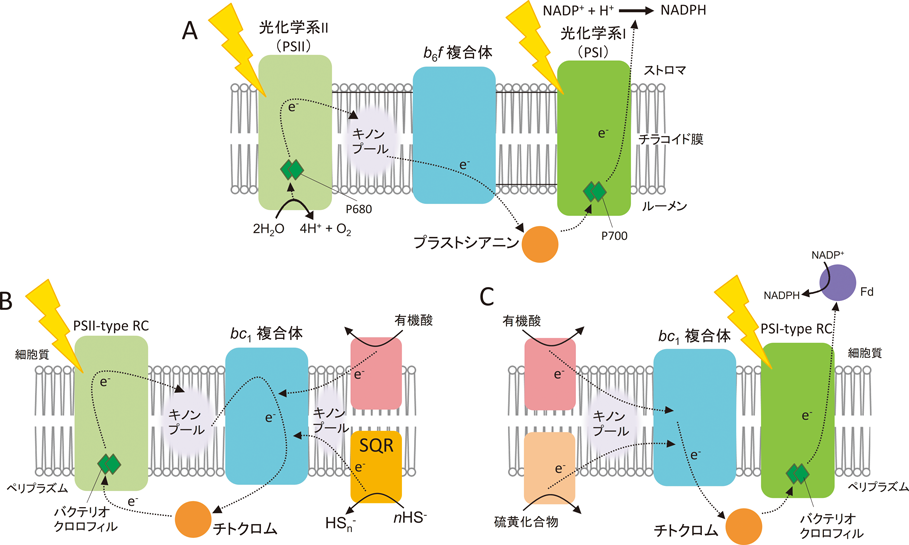
一般的によく知られている,植物やシアノバクテリアが行う光合成は,水を電子供与体とすることで酸素を発生するものである.この光合成では,二つの光化学系(PSI, PSII)を組み合わせた2回の光励起によって,還元力の低い水を電子供与体とした電子伝達と還元力(NADPH)の産生を可能にしている.一方で,シアノバクテリアを除く光合成細菌(正確には酸素非発生型光合成細菌と呼ぶが,本稿では便宜的に光合成細菌と呼ぶ)は,PSIタイプあるいはPSIIタイプの光化学系を1種類だけもち,水よりも酸化が容易な硫化水素などの硫黄化合物などを用いて光合成を行う(図1).
この硫化水素を電子供与体とした光合成電子伝達において,硫化水素の酸化には,sulfide:quinone oxidoreductase(SQR)26),flavocytochrome c sulfide dehydrogenase(FccAB)27–29),Sox酵素群30, 31)の3種類の酵素の関与が報告されている.しかし,Sox酵素群は硫化水素酸化ではなくチオ硫酸の酸化に密接に関係している場合も多く32),また光合成細菌の一種である紅色光合成細菌には,fccAB遺伝子が存在しないことから33),この2種類の酵素の硫化水素酸化における重要性は疑問視されている29).一方,SQRは多くの細菌にとって硫化水素の酸化に重要であり32),紅色光合成細菌のsqr遺伝子欠損株は硫化水素依存的な光合成生育が不可能になる33).以上のことから,SQRが光合成における硫化水素酸化で特に主要な役割を担うと考えられている.
SQRは,光合成細菌およびシアノバクテリアの細胞膜で硫化水素依存的な酸化還元反応を起こす酵素として1970年代からその存在が推測されていた34, 35).1990年代になって,シアノバクテリアOscillatoria limneticaから初めてSQRタンパク質が単離され36),その後,その遺伝子も同定された25).SQRは膜結合型の電子伝達タンパク質であり,硫化水素をスルフェンもしくはS0に二電子酸化することでキノンを還元し,キノンプール経由で光合成電子伝達系へと電子を供給すると考えられている(図1B).しかし,細胞外から取り込んだ硫化水素をエネルギー源もしくは電子源として用いる光合成細菌や化学合成細菌だけでなく,環境から供給される硫化水素とはほとんど無縁と思われる細菌や哺乳類などの真核生物もSQRを保持している.この事実を踏まえると,SQRは単純な硫化水素酸化に基づくエネルギー代謝以外にも重要な機能があることが推測される.実際,SQRは超硫黄分子の産生酵素としての側面も持ち合わせている.著者らは,SQRに着目して,紅色光合成細菌R. capsulatusを材料として超硫黄分子の応答機構を明らかにしてきた22–24).
紅色光合成細菌はアルファプロテオバクテリア門に属するが,ミトコンドリアの祖先はアルファプロテオバクテリア門が分岐する前に分岐したプロテオバクテリアの系統から進化した可能性があるため37),紅色光合成細菌はミトコンドリアの祖先細菌に進化的に近縁だといえる.実際,R. capsulatusは,真核細胞において硫化物を硫酸に酸化するミトコンドリア経路の中心的な構成要素である,ポリスルフィドジオキシゲナーゼ(persulfide dioxygenase:PDO),ロダネーゼ,亜硫酸オキシゲナーゼ,SQRを保持している38).したがって,R. capsulatusにおけるSqrRを介した超硫黄分子シグナル伝達の特徴を明らかにすることは,異なる生物種および研究領域にわたって超硫黄分子シグナル伝達をより理解するためのモデルとなりうる.
超硫黄分子センサータンパク質は,いくつかの細菌で同定され,その特徴が明らかにされている22, 39–42).これらは転写因子であり,システイン残基のチオール基のポリスルフィド修飾を介して転写抑制または活性化を誘導することにより,SQRのような硫黄代謝関連酵素をコードする遺伝子の発現を調節する.こうしたシステイン残基を介した超硫黄分子の検知システムは,異なる構造を持つ超硫黄分子センサータンパク質の間で保存されている特徴だと思われる22, 43).
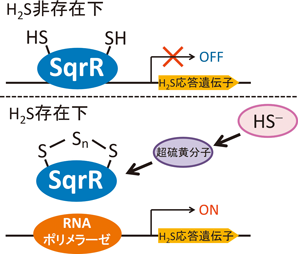
著者らは,R. capsulatusにおいてSQRの発現が硫化水素によって誘導を受けることに着目し33),sqr遺伝子の発現を指標にした順遺伝学的な手法によって,新規の超硫黄分子応答性転写因子SqrRを同定し,その分子機構を明らかにした22).SqrRは,超硫黄分子の一種であるグルタチオンパースルフィド(GSSH)と反応すると,二つの保存されたシステイン残基の間に分子内テトラスルフィド結合を形成し,標的オペレーター領域に対するin vitroでのDNA結合活性が低下する22)(図2).このポリスルフィド修飾について,質量分析を用いた動的プロファイリングにより,SqrRのシステイン残基は,過酸化物やROSとは反応せず,超硫黄分子特異的に酸化されることが示されている43).SqrRの還元型と酸化型(ジスルフィド結合形成型,テトラスルフィド結合形成型)の結晶構造解析から,この超硫黄分子との特異的な反応は,超硫黄分子によるテトラスルフィド状態がROSによって形成されるスルフィド状態に比べてエネルギー的な「不安定さ」が少ないことに起因すると考えられ43),この特性により,超硫黄分子とほかの酸化剤によるストレス応答を切り分けることが可能になると考えられる.ただし,著者らは,R. capsulatusにおける活性酸素ストレスに応答したバイオフィルム形成の制御にSqrRが寄与することを明らかにしている44).また,大腸菌のSqrRオルソログであるYgaVは,細胞内硫化水素レベルに応答して鉄取り込み遺伝子発現を調節することで,酸化ストレス応答に寄与することが見いだされている45).したがって,超硫黄分子と活性酸素への応答機構は一部で冗長性が残されていると考えられるが,その分子機構はわかっていない.
細菌における超硫黄分子センサータンパク質の最初の報告は,米国インディアナ大学のグループよって同定された,黄色ブドウ球菌Staphylococcus aureusの転写因子CsoR-like sulfur-transferase repressor(CstR)である39).SqrRが分子内でテトラスルフィド架橋を形成するのに対し,CstRは分子間でテトラスルフィド架橋を形成して二量体化することでDNA結合親和性が低下する.CstRは,マルチドメイン硫酸転移酵素,PDO, SQRの3種類の硫黄代謝関連酵素をコードする遺伝子の転写制御を介して,超硫黄分子の細胞内レベルの調節に寄与することが示されている39, 46).また,植物病原菌Xylella fastidiosaでは,PDOとロダネーゼの両ドメインを持つβラクタマーゼ様加水分解酵素(Blh)が,SqrRオルソログBigRによる超硫黄分子に応答した転写制御を介して,超硫黄分子をチオール化合物と亜硫酸塩に変換すると予測されている46, 47).このように,細菌における超硫黄分子応答の多くは,硫黄代謝に関わることが推察できるが,細胞内の超硫黄分子代謝とシグナル伝達の関係性を統合的に理解するまでは至っていなかった.
R. capsulatusは,歴史的には「紅色非硫黄細菌」に分類されているが,これは硫化物を硫黄に酸化する能力が低いか,まったくないことを意味している.しかし,R. capsulatusはSQRを持ち,硫化水素依存的な光合成による独立栄養生育が可能であることから33),「硫黄細菌」「非硫黄細菌」の使い分けはされていない.興味深いことに,R. capsulatusはS. aureusのCstBとX. fastidiosaのBlhに類似したドメインを持つ硫黄転移酵素を持つ38).また,大腸菌や哺乳類にみられる超硫黄分子代謝酵素CAT/MSTは,R. capsulatusゲノムにも存在する.このように,R. capsulatusは,超硫黄分子種の代謝と細胞内シグナル伝達の制御の相互作用を理解できるような,超硫黄分子代謝ネットワークの重要な要素をすべて持っていると考えられる.
R. capsulatus野生型(WT)およびsqrR欠損株(ΔsqrR)のトランスクリプトーム解析の結果,大部分の超硫黄分子応答性遺伝子(45%)の転写制御にSqrRが関与することがわかっている22).著者らは,このうち,超硫黄分子代謝に関連する酵素に着目し,細胞内の超硫黄分子代謝とSqrR依存的な転写制御に対する各酵素の寄与を検証した.その結果,SQRと硫酸転移酵素であるロダネーゼの一種(rcc01557)が,ポリスルフィド修飾によるSqrRの転写制御調節と細胞内超硫黄分子の分子種に影響を与え,それによって超硫黄分子応答が調節されていることがわかった24).
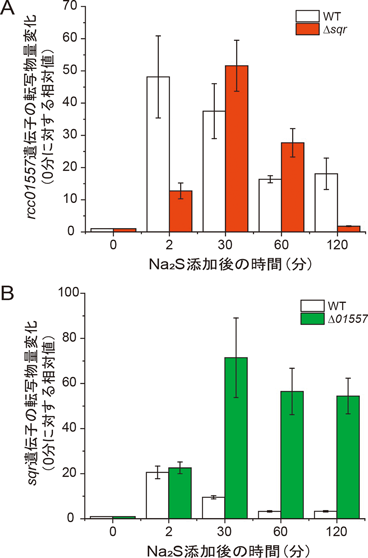
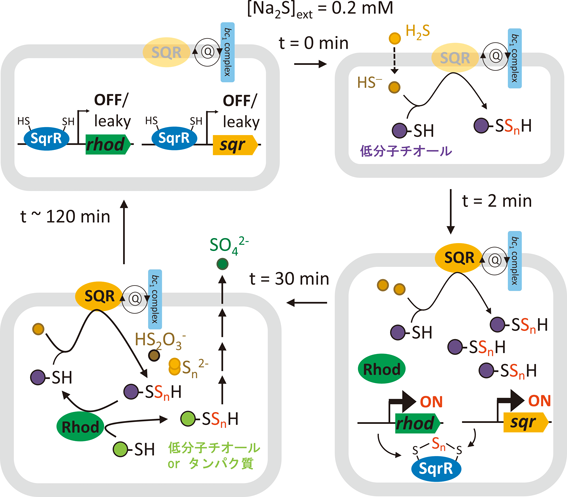
sqr遺伝子欠損株(Δsqr)およびrcc01557遺伝子欠損株(Δ01557)について,SqrRが調節する遺伝子の転写物量をモニターすると,細胞を硫化ナトリウム(Na2S)で処理した後,WTでは転写物量が急激に増加し,その後緩やかに減少するが,60~120分後の時点では有意な減少はみられない(図3).これに対して,Δsqrでは,転写物量が30分後以降に有意に減少し,長時間経過後に顕著に低下する(図3A).一方,Δ01557では,硫化水素処理後に増加した転写物量は高レベルのまま持続される(図3B).SqrRは,超硫黄分子によってポリスルフィド修飾を受けることで転写抑制活性が失われるため,SQRとrcc01557のコードするロダネーゼはそれぞれ,SqrRの修飾に関わる超硫黄分子の合成(供給)と分解(除去)に関わると考えられる.
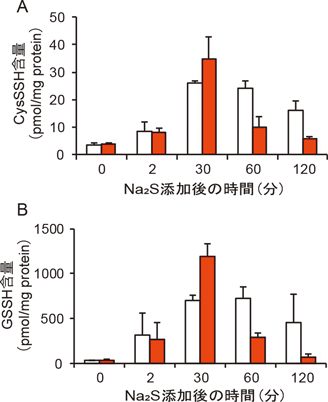
実際に,これら酵素のリコンビナントタンパク質を用いた活性解析から,それぞれ硫黄代謝活性を持つことがわかっている.ロダネーゼは,チオ硫酸とシアン化物(CN−)をそれぞれ硫黄供与体および受容体としたチオ硫酸硫黄転移酵素活性を持つことがわかっているが,細胞内での実際の基質は不明であり,対応する硫黄受容体の特定には至っていない.一方,SQRは,システイン(CysSH)あるいは還元型グルタチオン(GSH)を硫黄受容体,ユビキノンを電子受容体とした硫化水素酸化活性が確認されている.さらに,β-(4-ヒドロキシフェニル)エチルヨードアセトアミド(HPE-IAM)を使用したLC-MS/MS解析から,SQRがシステインパースルフィド(CysSSH)およびGSSHの細胞内産生に寄与することがわかっている.硫化水素処理後の細胞内超硫黄分子レベルの経時的変化は,SqrRによる転写制御を受ける遺伝子の転写物量の経時的変化と相関しており,ΔsqrにおいてNa2S添加後60~120分で観察される転写物量の低下は,SQRによるCysSSHおよびGSSH産生の欠如に起因する可能性がある(図4).これより,SQRによる超硫黄分子産生は,長時間にわたって超硫黄分子を高いレベルに維持する役割を果たしていると考えられている.
また,SQRによって供給される超硫黄分子種のうち,CysSSHがSqrR依存的な転写制御の調節に重要であることが,培養液への超硫黄分子の添加実験からわかっている.培地にシスチン(CysSSCys)または酸化型グルタチオン(GSSG)とNa2Sを等モルで添加すると,低濃度ではあるが,CysSSHやGSSHが合成される.ΔsqrにおけるNa2S添加後60~120分時点での転写物量の低下は,CysSSCys由来超硫黄分子(CysSSH)を添加した場合にはWT同様に相補されるが,GSSG由来超硫黄分子(GSSH)で処理した場合は,そのような効果は観察されない.したがって,CysSSCys由来超硫黄分子のCysSSHが,SQRによって調節されるSqrR依存性の超硫黄分子応答をより効果的に維持すると考えられる.
一方,GSSHは細胞内の超硫黄分子として最も多いことが報告されており13),サルモネラ菌やマウスでは,通常生育条件下において,GSSHはCysSSHよりも5~30倍多く存在する20, 48).R. capsulatusにおいても,GSSHはCysSSHよりも10~30倍多いことが示されており,SQRによるCysSSHおよびGSSHの産生活性がほぼ同じであることを考慮すると,CysSSHがSqrR依存的な転写制御に積極的に利用される仕組みは明確にはわかっていない.これを説明できる可能性として,CysSSHとGSSHのSqrRとの反応性の違いがあげられる.蛍光異方性を用いたSqrRのDNA解離速度の解析から,CysSSHがGSSHよりもSqrRを酸化する速度が約4倍高いことが示されている24).さらに,質量分析により,GSSHが主にテトラスルフィド架橋を形成するのに対し,CysSSHはペンタスルフィド架橋を主要生成物とする混合ポリスルフィド生成物を形成する24).SqrRの迅速かつ持続的な転写応答を引き起こす化学的特性の違いを完全に説明することは困難ではあるが,システインは酸性度がGSHよりも低いため優れた脱離基であり49),より迅速な酸化をもたらす可能性があると考えられている.さらに,SqrRのポリスルフィド修飾を受ける二つのシステイン残基は構造的に空洞(cavity)にあり,片方のシステイン残基が求核攻撃を受けることで,もう片方のシステイン残基への求核攻撃が遮断される可能性があることが示唆されている50).この構造は,ほかの求電子剤に対するポリスルフィドの選択性に寄与することが示されており50),試験管内ではCysSSHがGSSHよりも選択的に反応する理由を説明するかもしれない.これは,CysSSHがGSSHとの反応では観察されない混合ポリスルフィドのような付加的な中間体を形成することと関係があるように思われる24).
SqrRを中心とした超硫黄分子応答でCysSSHが積極的に利用される理由として,超硫黄分子の代謝系にヒントがあるかもしれない.ミトコンドリアでは,CysSSHは,主に外因性硫化物に関係なくシステインからCARS2によって合成され,その後,ポリスルフィドスクランブルを介してGSSHの形成を引き起こす13, 20).R. capsulatusにおいて,培養液にCysSSCys由来超硫黄分子(CysSSH)を添加した際に細胞内の超硫黄分子種の変化を観察すると,CysSSHは一過的に急速な増加を示した後,GSSHなどのほかの超硫黄分子の生成を伴って急激に減少する.同様の効果は,外因性シスチンから生成されたCysSSHでもミトコンドリア内で観察される13).こうした知見を踏まえて著者らは,CysSSHおよびGSSHよりも反応性の高いその他のヒドロパースルフィド(RSSH)などの超硫黄分子が,SqrR依存的な転写の脱抑制を誘導する初期のシグナル分子として機能し,それによって発現が誘導されたSQRによって,さらに多くの超硫黄分子が持続的に供給されることで,SqrRの脱抑制状態が継続されるモデルを提唱している(図5)24).
SQRによる超硫黄分子濃度の維持は,SqrRによる超硫黄分子応答を持続させる.一方で,一般的に,細胞は過剰な超硫黄分子から自らを守る必要がある.実際に,哺乳細胞では,チオレドキシンおよびグルタレドキシンが超硫黄分子およびタンパク質のポリスルフィド修飾を還元できることが知られている51).さらに,ロダネーゼなどの硫黄転移酵素およびPDOも,超硫黄分子過剰生成の「オフスイッチ」として機能する52).R. capsulatusにおいても,SqrRの転写制御下にあるチオレドキシンTrxCが,SqrRのポリスルフィド架橋を直接的に還元し,転写抑制を回復させることがわかっている53).このメカニズムにより,SQR由来の超硫黄分子濃度が過剰に上昇するのを防ぐことができる.
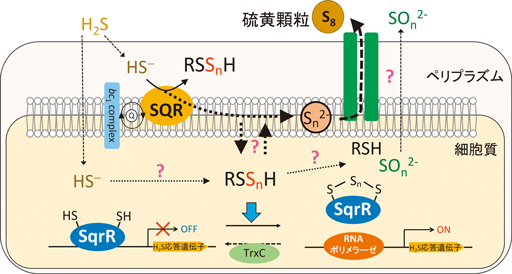
こうしたSqrRの活性調節に加え,超硫黄分子の分解系と排出系が機能することで,超硫黄分子の細胞内恒常性が維持されていると考えられる.R. capsulatusにおいて,硫化水素由来の硫黄はSQRによって超硫黄分子の形に酸化された後に,硫酸などへ代謝される場合と,S8を主成分とした“硫黄顆粒”と呼ばれる直径0.5 µm程度の顆粒の形で細胞外に集積する場合とがある31).こうした超硫黄分子の分解系については,まだよくわかっていない点が多いが,前述したロダネーゼ(rcc01181)は,硫黄転移を介して超硫黄分子の分解に寄与すると考えられる.また,興味深いことに,超硫黄分子含量の経時的変化の解析から,CysSSHとGSSHなどの有機の超硫黄分子とは別に,無機の超硫黄分子である二硫化水素(HS2−)および三硫化水素(HS3−)についても,sqr欠損株における細胞内含量の低下が引き起こされることがわかっている.これは,ヒトのSQRが無機超硫黄分子種を基質およびS0アクセプターとして利用できることや54),細菌Aquifex aeolicusのSQRの結晶構造中にS8の電子密度が観察されること55)と一致している.一方で,硫黄顆粒の排出経路については,トランスポーターが同定されていないため,まったくわかっていない.SQRによる産生可能性のある超硫黄分子種に構造的に安定なS8が含まれること,S8が無極性であることを考慮すると,SQRによって産生されたS8が細胞膜内部に直接移動し,細胞膜内の基質を細胞外に排出するようなトランスポーターによって細胞外に排出されて硫黄顆粒が形成される可能性が考えられる(図6).
超硫黄分子の代謝動態と輸送系が超硫黄分子シグナル伝達において果たす機能的役割を解明することは,R. capsulatusに限らず,ほかの生物におけるこれらのプロセスが生理学的機能に与える影響について,より深い理解をもたらすことが期待される.
光合成細菌は古くから研究されてきた細菌であり,調べつくされた生物だと感じる点もあるが,いくつかの研究分野では,いまだに非常に有力な研究材料であると考える.たとえば,光合成細菌は細胞外の酸素分圧に応じてダイナミックに内膜の構造を変化させ,光合成条件下では特徴的な陥入膜(intracytoplasmic membrane:ICM)を形成することが古くから知られている.ICM形成のメカニズムや制御系についてはほとんどわかっておらず,これを解明することは,ミトコンドリアや葉緑体の内膜系の形態やそこに存在するタンパク質の配置に関する研究に役立つと思われる.
硫黄代謝についても古くから研究があり,多くの硫黄代謝関連酵素が同定され,その局在や機能が明らかになっている56).また,硫黄顆粒についても,その組成分子種や顆粒形成に関する解析が行われている57).こうした硫黄代謝を,超硫黄分子を考慮して再構築し,超硫黄分子応答と結びつけることで,ほかの生物よりも容易に超硫黄分子の検知機構を包括的に理解できると考えられる.特に,硫黄顆粒を超硫黄分子の代謝の視覚的な指標として利用できることは,ほかの材料よりもかなり有利であると考える.
ノーベル賞を受賞した世界初の膜タンパク質のX線結晶構造解析である光合成細菌の光合成反応中心タンパク質のように58),光合成細菌の硫黄代謝から新しい生命現象の理解が進むことを期待している.
謝辞Acknowledgments
本総説は2024年度生化学会奨励賞の受賞に際し,寄稿させていただきました.博士後期課程から私を引き受けて寛大な心で研究のイロハをご教授くださった,東京工業大学(現・東京科学大学)の増田真二教授には,心より感謝申し上げます.また,これまで研究をご指導してくださいました先生方や共同研究者のみなさま,ともに研鑽してきた友人にも,感謝申し上げます.
引用文献References
1) Rosing, M.T., Rose, N.M., Bridgwater, D., & Thomsen, H.S. (1996) Earliest part of Earth’s stratigraphic record: A reappraisal of the >3.7 Ga Isua (Greenland) supracrustal sequence. Geology, 24, 43–46.
2) Dymek, R.F. & Klein, C. (1988) Chemistry, petrology and origin of banded iron-formation lithologies from the 3800 MA isua supracrustal belt, West Greenland. Precambrian Res., 39, 247–302.
3) Canfield, D.E., Rosing, M.T., & Bjerrum, C. (2006) Early anaerobic metabolisms. Philos. Trans. R. Soc. Lond. B Biol. Sci., 361, 1819–1836.
4) Lyons, T.W., Anbar, A.D., Severmann, S., Scott, C., & Gill, B.C. (2009) Tracking euxinia in the ancient ocean: A multiproxy perspective and proterozoic case study. Annu. Rev. Earth Planet. Sci., 37, 507–534.
5) Baross, J.A. & Hoffman, S.E. (1985) Submarine hydrothermal vents and associated gradient environments as sites for the origin and evolution of life. Orig. Life Evol. Biosph., 15, 327–345.
6) Corliss, J.B., Baross, J.A., & Hoffman, S.E. (1981) An hypothesis concerning the relationship between submarine hot springs and the origin of life on Earth. Oceanol. Acta, 4, 59–69.
7) Shen, Y., Buick, R., & Canfield, D.E. (2001) Isotopic evidence for microbial sulphate reduction in the early Archaean era. Nature, 410, 77–81.
8) Fisher, C., Takai, K., & Le Bris, N. (2007) Hydrothermal vent ecosystems. Oceanography (Wash. D.C.), 20, 14–23.
9) Olson, K.R. (2020) Reactive oxygen species or reactive sulfur species: why we should consider the latter. J. Exp. Biol., 223, jeb196352.
10) Buchner, P., Stuiver, C.E.E., Westerman, S., Wirtz, M., Hell, R., Hawkesford, M.J., & De Kok, L.J. (2004) Regulation of sulfate uptake and expression of sulfate transporter genes in Brassica oleracea as affected by atmospheric H(2)S and pedospheric sulfate nutrition. Plant Physiol., 136, 3396–3408.
11) Rennenberg, H. (1984) The fate of excess sulfur in higher plants. Annu. Rev. Plant Physiol., 35, 121–153.
12) Li, L., Rose, P., & Moore, P.K. (2011) Hydrogen sulfide and cell signaling. Annu. Rev. Pharmacol. Toxicol., 51, 169–187.
13) Ida, T., Sawa, T., Ihara, H., Tsuchiya, Y., Watanabe, Y., Kumagai, Y., Suematsu, M., Motohashi, H., Fujii, S., Matsunaga, T., et al. (2014) Reactive cysteine persulfides and S-polythiolation regulate oxidative stress and redox signaling. Proc. Natl. Acad. Sci. USA, 111, 7606–7611.
14) Nishida, M., Sawa, T., Kitajima, N., Ono, K., Inoue, H., Ihara, H., Motohashi, H., Yamamoto, M., Suematsu, M., Kurose, H., et al. (2012) Hydrogen sulfide anion regulates redox signaling via electrophile sulfhydration. Nat. Chem. Biol., 8, 714–724.
15) Cuevasanta, E., Lange, M., Bonanata, J., Coitiño, E.L., Ferrer-Sueta, G., Filipovic, M.R., & Alvarez, B. (2015) Reaction of hydrogen sulfide with disulfide and sulfenic acid to form the strongly nucleophilic persulfide. J. Biol. Chem., 290, 26866–26880.
16) Fukuto, J.M. (2023) The chemistry of hydropersulfides (RSSH) as related to possible physiological functions. Arch. Biochem. Biophys., 743, 109659.
17) Barayeu, U., Schilling, D., Eid, M., Xavier da Silva, T.N., Schlicker, L., Mitreska, N., Zapp, C., Gräter, F., Miller, A.K., Kappl, R., et al. (2023) Hydropersulfides inhibit lipid peroxidation and ferroptosis by scavenging radicals. Nat. Chem. Biol., 19, 28–37.
18) Barayeu, U., Sawa, T., Nishida, M., Wei, F.-Y., Motohashi, H., & Akaike, T. (2023) Supersulfide biology and translational medicine for disease control. Br. J. Pharmacol., Online ahead of print, Octobe 23, 2023, 1–16.
19) Zhang, T., Akaike, T., & Sawa, T. (2024) Redox Regulation of Xenobiotics by reactive sulfur and supersulfide species. Antioxid. Redox Signal., 40, 679–690.
20) Akaike, T., Ida, T., Wei, F.-Y., Nishida, M., Kumagai, Y., Alam, M.M., Ihara, H., Sawa, T., Matsunaga, T., Kasamatsu, S., et al. (2017) Cysteinyl-tRNA synthetase governs cysteine polysulfidation and mitochondrial bioenergetics. Nat. Commun., 8, 1177.
21) Alam, M.M., Kishino, A., Sung, E., Sekine, H., Abe, T., Murakami, S., Akaike, T., & Motohashi, H. (2023) Contribution of NRF2 to sulfur metabolism and mitochondrial activity. Redox Biol., 60, 102624.
22) Shimizu, T., Shen, J., Fang, M., Zhang, Y., Hori, K., Trinidad, J.C., Bauer, C.E., Giedroc, D.P., & Masuda, S. (2017) Sulfide-responsive transcriptional repressor SqrR functions as a master regulator of sulfide-dependent photosynthesis. Proc. Natl. Acad. Sci. USA, 114, 2355–2360.
23) Shimizu, T. & Masuda, S. (2017) Characterization of redox-active cysteine residues of persulfide-responsive transcriptional repressor SqrR. Commun. Integr. Biol., 10, e1329786.
24) Shimizu, T., Ida, T., Antelo, G.T., Ihara, Y., Fakhoury, J.N., Masuda, S., Giedroc, D.P., Akaike, T., Capdevila, D.A., & Masuda, T. (2023) Polysulfide metabolizing enzymes influence SqrR-mediated sulfide-induced transcription by impacting intracellular polysulfide dynamics. PNAS Nexus, 2, pgad048.
25) Schütz, M., Shahak, Y., Padan, E., & Hauska, G. (1997) Sulfide-quinone reductase from Rhodobacter capsulatus. Purification, cloning, and expression. J. Biol. Chem., 272, 9890–9894.
26) Chen, Z.W., Koh, M., Van Driessche, G., Van Beeumen, J.J., Bartsch, R.G., Meyer, T.E., Cusanovich, M.A., & Mathewst, F.S. (1994) The structure of flavocytochrome c sulfide dehydrogenase from a purple phototrophic bacterium. Science, 266, 430–432.
27) Kostanjevecki, V., Brigé, A., Meyer, T.E., Cusanovich, M.A., Guisez, Y., & Van Beeumen, J. (2000) A membrane-bound flavocytochrome c-sulfide dehydrogenase from the purple phototrophic sulfur bacterium Ectothiorhodospira vacuolata. J. Bacteriol., 182, 3097–3103.
28) Brune, D.C. (1995) Advances in Photosynthesis, Vol. 2, Kluwer Academic Publishers, Dordrecht, pp. 847–870.
29) Appia-Ayme, C., Little, P.J., Matsumoto, Y., Leech, A.P., & Berks, B.C. (2001) Cytochrome complex essential for photosynthetic oxidation of both thiosulfate and sulfide in Rhodovulum sulfidophilum. J. Bacteriol., 183, 6107–6118.
30) Ogawa, T., Furusawa, T., Shiga, M., Seo, D., Sakurai, H., & Inoue, K. (2010) Biochemical studies of a soxF-encoded monomeric flavoprotein purified from the green sulfur bacterium Chlorobaculum tepidum that stimulates in vitro thiosulfate oxidation. Biosci. Biotechnol. Biochem., 74, 771–780.
31) Hansen, T.A. & van Gemerden, H. (1972) Sulfide utilization by purple nonsulfur bacteria. Arch. Microbiol., 86, 49–56.
32) Sander, J. & Dahl, C. (2009) The purple phototrophic bacteria (C.N. Hunter, F. Daldal, M.C. Thurnaier & J.T. Beatty, eds.), pp. 595–622, Springer.
33) Schütz, M., Maldener, I., Griesbeck, C., & Hauska, G. (1999) Sulfide-quinone reductase from Rhodobacter capsulatus: requirement for growth, periplasmic localization, and extension of gene sequence analysis. J. Bacteriol., 181, 6516–6523.
34) Shahak, Y., Arieli, B., Binder, B., & Padan, E. (1987) Sulfide-dependent photosynthetic electron flow coupled to proton translocation in thylakoids of the cyanobacterium Oscillatoria limnetica. Arch. Biochem. Biophys., 259, 605–615.
35) Knaff, D.B. & Buchanan, B.B. (1975) Cytochrome b and photosynthetic sulfur bacteria. Biochim. Biophys. Acta Bioenerg., 376, 549–560.
36) Arieli, B., Shahak, Y., Taglicht, D., Hauska, G., & Padan, E. (1994) Purification and characterization of sulfide-quinone reductase, a novel enzyme driving anoxygenic photosynthesis in Oscillatoria limnetica. J. Biol. Chem., 269, 5705–5711.
37) Martijn, J., Vosseberg, J., Guy, L., Offre, P., & Ettema, T.J.G. (2018) Deep mitochondrial origin outside the sampled alphaproteobacteria. Nature, 557, 101–105.
38) Shimizu, T. & Masuda, S. (2020) Persulphide-responsive transcriptional regulation and metabolism in bacteria. J. Biochem., 167, 125–132.
39) Luebke, J.L., Shen, J., Bruce, K.E., Kehl-Fie, T.E., Peng, H., Skaar, E.P., & Giedroc, D.P. (2014) The CsoR-like sulfurtransferase repressor (CstR) is a persulfide sensor in Staphylococcus aureus. Mol. Microbiol., 94, 1343–1360.
40) Li, H., Li, J., Lü, C., Xia, Y., Xin, Y., Liu, H., Xun, L., & Liu, H. (2017) FisR activates σ54-dependent transcription of sulfide-oxidizing genes in Cupriavidus pinatubonensis JMP134. Mol. Microbiol., 105, 373–384.
41) Hou, N., Yan, Z., Fan, K., Li, H., Zhao, R., Xia, Y., Xun, L., & Liu, H. (2019) OxyR senses sulfane sulfur and activates the genes for its removal in Escherichia coli. Redox Biol., 26, 101293.
42) Fakhoury, J.N., Zhang, Y., Edmonds, K.A., Bringas, M., Luebke, J.L., Gonzalez-Gutierrez, G., Capdevila, D.A., & Giedroc, D.P. (2021) Functional asymmetry and chemical reactivity of CsoR family persulfide sensors. Nucleic Acids Res., 49, 12556–12576.
43) Walsh, B.J.C. & Giedroc, D.P. (2020) H2S and reactive sulfur signaling at the host-bacterial pathogen interface. J. Biol. Chem., 295, 13150–13168.
44) Shimizu, T., Aritoshi, T., Beatty, J.T., & Masuda, T. (2022) Persulfide-Responsive transcription factor SqrR regulates gene transfer and biofilm formation via the metabolic modulation of cyclic di-GMP in Rhodobacter capsulatus. Microorganisms, 10, 908.
45) Nonoyama, S., Maeno, S., Gotoh, Y., Sugimoto, R., Tanaka, K., Hayashi, T., & Masuda, S. (2024) Increased intracellular H2S levels enhance iron uptake in Escherichia coli. MBio, 15, e0199124.
46) Shen, J., Keithly, M.E., Armstrong, R.N., Higgins, K.A., Edmonds, K.A., & Giedroc, D.P. (2015) Staphylococcus aureus CstB is a novel multidomain persulfide Dioxygenase-Sulfurtransferase involved in hydrogen sulfide detoxification. Biochemistry, 54, 4542–4554.
47) De Lira, N.P.V., Pauletti, B.A., Marques, A.C., Perez, C.A., Caserta, R., De Souza, A.A., Vercesi, A.E., Paes Leme, A.F., & Benedetti, C.E. (2018) BigR is a sulfide sensor that regulates a sulfur transferase/dioxygenase required for aerobic respiration of plant bacteria under sulfide stress. Sci. Rep., 8, 3508.
48) Khan, S., Fujii, S., Matsunaga, T., Nishimura, A., Ono, K., Ida, T., Ahmed, K.A., Okamoto, T., Tsutsuki, H., Sawa, T., et al. (2018) Reactive persulfides from salmonella typhimurium downregulate Autophagy-Mediated innate immunity in macrophages by inhibiting electrophilic signaling. Cell Chem. Biol., 25, 1403–1413.e4.
49) Szajewski, R.P. & Whitesides, G.M. (1980) Rate constants and equilibrium constants for thiol-disulfide interchange reactions involving oxidized glutathione. J. Am. Chem. Soc., 102, 2011–2026.
50) Capdevila, D.A., Walsh, B.J.C., Zhang, Y., Dietrich, C., Gonzalez-Gutierrez, G., & Giedroc, D.P. (2021) Structural basis for persulfide-sensing specificity in a transcriptional regulator. Nat. Chem. Biol., 17, 65–70.
51) Dóka, É., Pader, I., Bíró, A., Johansson, K., Cheng, Q., Ballagó, K., Prigge, J.R., Pastor-Flores, D., Dick, T.P., Schmidt, E.E., et al. (2016) A novel persulfide detection method reveals protein persulfide- and polysulfide-reducing functions of thioredoxin and glutathione systems. Sci. Adv., 2, e1500968.
52) Ran, M., Li, Q., Xin, Y., Ma, S., Zhao, R., Wang, M., Xun, L., & Xia, Y. (2022) Rhodaneses minimize the accumulation of cellular sulfane sulfur to avoid disulfide stress during sulfide oxidation in bacteria. Redox Biol., 53, 102345.
53) Shimizu, T., Hashimoto, M., & Masuda, T. (2023) Thioredoxin-2 regulates SqrR-Mediated Polysulfide-Responsive transcription via reduction of a polysulfide link in SqrR. Antioxidants, 12, 699.
54) Jackson, M.R., Melideo, S.L., & Jorns, M.S. (2012) Human sulfide:quinone oxidoreductase catalyzes the first step in hydrogen sulfide metabolism and produces a sulfane sulfur metabolite. Biochemistry, 51, 6804–6815.
55) Marcia, M., Ermler, U., Peng, G., & Michel, H. (2009) The structure of Aquifex aeolicus sulfide:quinone oxidoreductase, a basis to understand sulfide detoxification and respiration. Proc. Natl. Acad. Sci. USA, 106, 9625–9630.
56) Frigaard, N.C. & Dahl, C. (2009) Sulfur metabolism in phototrophic sulfur bacteria. Adv. Microb. Physiol., 54, 103–200.
57) Maki, J.S. (2013) Bacterial intracellular sulfur globules: structure and function. Microb. Physiol., 23, 270–280.
58) Deisenhofer, J. & Michel, H. (1989) Nobel lecture. The photosynthetic reaction centre from the purple bacterium Rhodopseudomonas viridis. EMBO J., 8, 2149–2170.
著者紹介Author Profile
 清水 隆之(しみず たかゆき)
清水 隆之(しみず たかゆき)奈良女子大学研究院自然科学系生物科学領域 准教授.博士(理学).
略歴1988年神奈川県に生る.2011年首都大学東京都市教養学部卒業.13年同大学院理工学研究科生命科学専攻修士課程修了.17年東京工業大学大学院生命理工学研究科博士課程修了.17年東京大学大学院総合文化研究科助教.23年より現職.
研究テーマと抱負細菌および植物におけるシグナル伝達機構の生化学・生理学的解析.特に硫化水素・超硫黄分子によるタンパク質のポリスルフィド化を介したシグナル伝達機構の解析に取り組んでいる.
ウェブサイトhttps://tshimizu-lab.jp/
趣味温泉.サウナ.