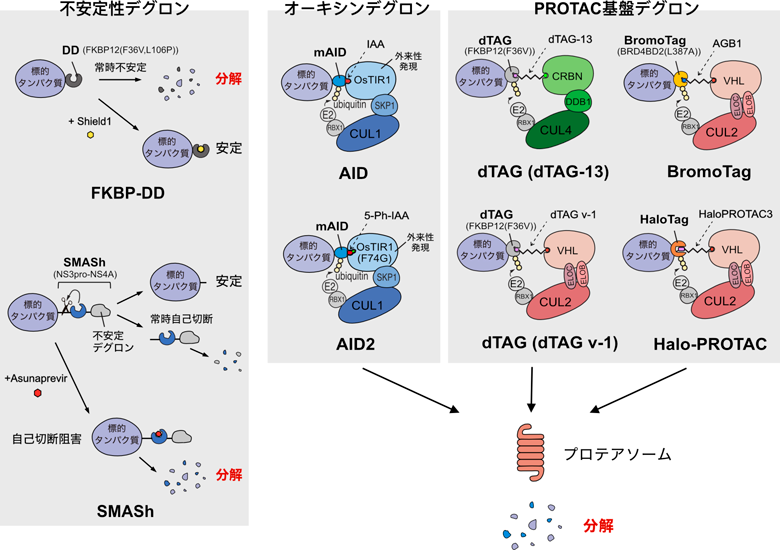
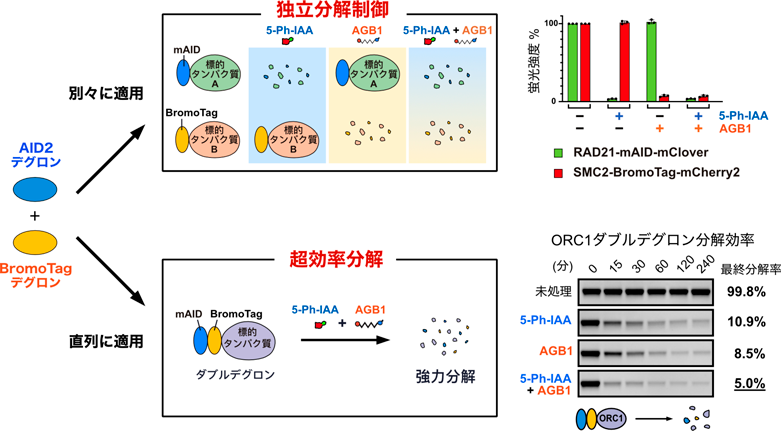
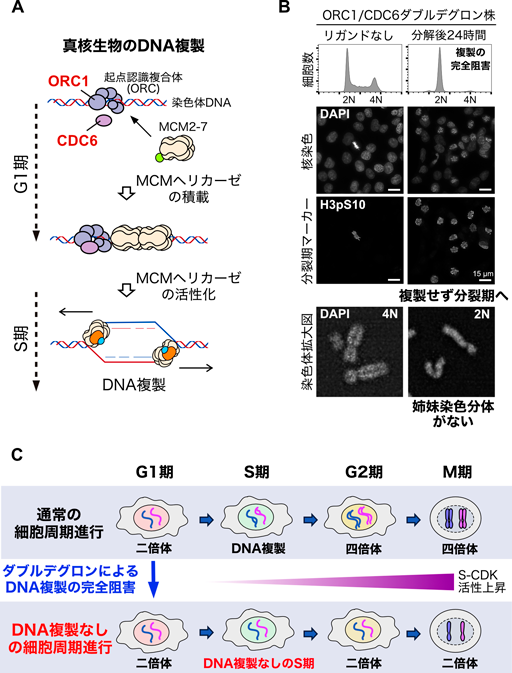
オーキシンデグロン法の改良と機能拡張Optimization and functional enhancement of the auxin-inducible degron system.
1 国立遺伝学研究所遺伝メカニズム研究系National Institute of Genetics, Department of Chromosome Science ◇ 〒411–8540 静岡県三島市谷田1111 ◇ Yata 1111, Mishima, Shizuoka 411–8540, Japan
2 総合研究大学院大学遺伝学専攻SOKENDAI, Graduate Institute for Advanced Studies ◇ 〒411–8540 静岡県三島市谷田1111 ◇ Yata 1111, Mishima, Shizuoka 411–8540, Japan
3 東京大学大学院理学系研究科生物学科The University of Tokyo, Department of Biological Science ◇ 〒113–0033 東京都文京区本郷7–3–1 ◇ Hongo 7–3–1, Bunkyo-ku, Tokyo 113–0033, Japan